刘小乐教授:CRISPR高通量筛选的新算法
高通量的CRISPR筛选,已在功能基因组学研究中显示出巨大的潜力。12月16日,华人女学者、哈佛大学公共卫生学院Dana-Farber癌症研究所的刘小乐教授,在国际著名学术期刊《Genome Biology》发表题为“Quality control, modeling, and visualization of CRISPR screens with MAGeCK-VISPR”的学术研究论文,为CRISPR筛选提出了一套综合质控(QC)、分析和可视化的工作流程――MAGeCK-VISPR。延伸阅读:刘小乐教授连发多项研究成果。
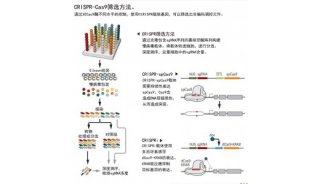
CRISPR/Cas9系统是一种强大的遗传工程技术,可让我们以一种经济合算的方式,在大多数模式生物中直接修饰基因组位点。基于这一系统,最近开发的高通量CRISPR筛选技术,已经在功能基因组学研究中显示出巨大的潜力,从而让研究人员能够系统地识别与各种表型相关的基因。CRISPR筛选,既可以通过用CRISPR/Cas9的直接基因敲除进行,也可以用CRISPR和与激活或抑制效应物融合的dCas9(失去DNA切割活性的Cas9蛋白)干扰基因表达。
虽然CRISPR筛选是一项强大的技术,但是它也产生了许多计算上的挑战,包括:(1)如何评价数据质量;(2)如何识别来自筛查的基因或通路命中以及评估其统计学意义;(3)如何有效地可视化和探讨筛选结果。直到现在,仍然还没有一种综合的质控(QC)、数据分析和可视化方法用于CRISPR筛选。
已有研究开发出几种算法,用于基因芯片或高通量测序数据的筛选分析。这些算法是在两种条件对比的基础上设计出来的,但是,许多筛选是在几个时间点、在许多处理条件下或许多细胞系中进行的。此外,这些算法没有考虑单向导RNA(sgRNA)对靶基因的敲除效率。
在这项研究中,研究人员提出了MAGeCK-VISPR来克服CRISPR筛选的计算挑战。MAGeCK-VISPR定义了一套QC测量,并通过最大似然估计方法(MAGeCK-MLE)扩展了MAGeCK算法,以在不同条件下调用必需的基因,同时考虑sgRNA的敲除效率。
此外,MAGeCK-VISPR,为CRISPR筛选质量控制和分析结果的交互式探测,提供了一个基于Web的可视化框架(VISPR)。MAGeCK-VISPR采用一种Snakemake工作流程,以一种可扩展和可重复的方式,将MAGeCK和VISPR结合起来。
随着CRISPR筛选变得越来越普遍,数据的复杂性(如批量效应),将是不可避免的,这就需要对有意义的下游分析进行适当的校正。现有的批量去除算法,已被广泛用于去除基因表达分析中的批量效应。在将来,这些算法可被整合到MAGeCK-VISPR流程中。从这以后,MAGeCK-VISPR将能够通过不同试验数据集之间的直接对比,确定癌症相关――以及疾病特异性的重要基因,从而为疾病和癌症的病理机制,提供新的治疗见解。
注:刘小乐 (Xiaole Shirley Liu) 青年时代就读于天津南开中学, 1992 年考入北京大学生物系。 1994 年转学到美国史密斯女子学院 (Smith College) 双修生物化学和计算机科学, 三年后以最高拉丁荣誉毕业 (Summa Cum Laude, 授予全校积分最高的 1% 的毕业生)。2002 年于斯坦福大学取得生物医学信息学博士和计算机科学辅修博士学位后, 被直接聘为哈佛大学终身制助理教授。她目前担任哈佛大学公共卫生学院生物统计与计算生物学系的终身正教授、Dana-Farber 肿瘤研究所功能性癌症表观遗传组学中心主任, 和同济大学生物信息学系教授并长江学者讲座教授。
-
项目成果

-
焦点事件

-
焦点事件

-
政策法规
-
科技前沿

-
科技前沿

-
科技前沿

-
市场商机

-
科技前沿

-
产品技术

-
产品技术
-
企业风采

-
焦点事件

-
综述

-
焦点事件

-
产品技术
-
科技前沿
-
科技前沿
-
科技前沿

-
科技前沿

-
科技前沿

-
科技前沿

-
项目成果
-
企业风采

-
项目成果
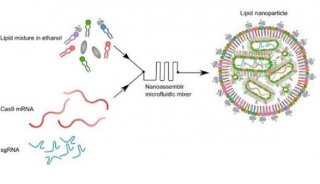
-
焦点事件

-
项目成果
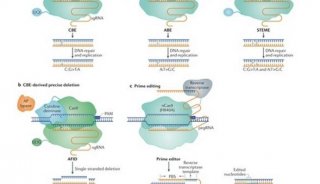
-
焦点事件

-
项目成果
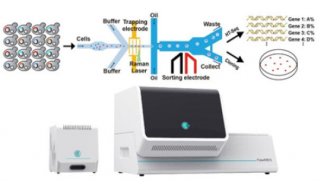
-
焦点事件
-
综述

-
科技前沿
-
项目成果
-
科技前沿
-
科技前沿
-
焦点事件

-
焦点事件

-
焦点事件

-
焦点事件

-
企业风采

-
科技前沿
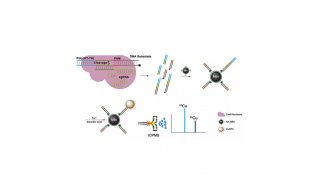
-
项目成果
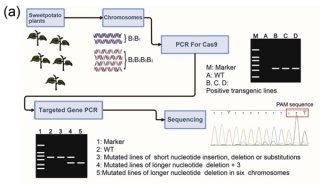
-
焦点事件
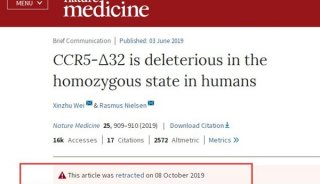
-
项目成果
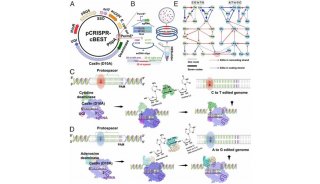
-
焦点事件
-
项目成果
-
焦点事件
-
项目成果
-
项目成果
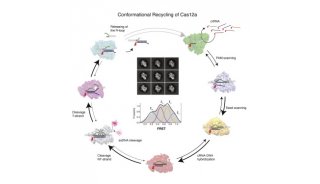
-
项目成果
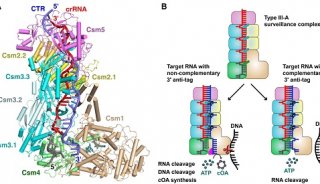
-
项目成果
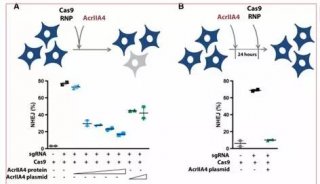
-
并购

-
科技前沿
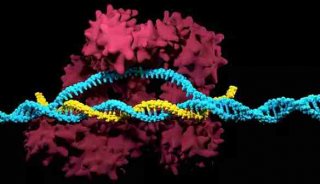
-
焦点事件

-
焦点事件

-
科技前沿
-
技术原理

-
技术原理
-
焦点事件
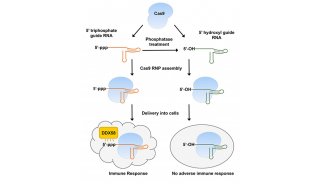
-
项目成果
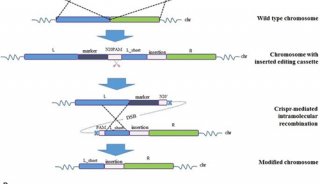
-
精英视角

-
焦点事件
-
焦点事件
-
焦点事件
-
项目成果
-
科技前沿
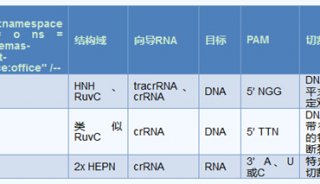
-
焦点事件

-
焦点事件

-
焦点事件
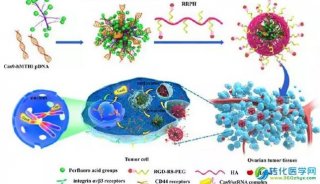
-
焦点事件