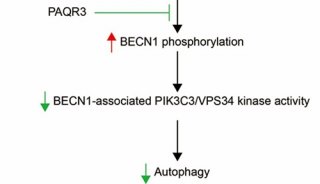
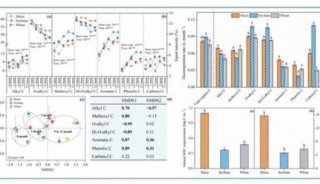
2016值得关注的技术:基因组分析深度学习
《Nature Methods》盘点2015年度技术,选出了最受关注的技术成果:单粒子低温电子显微镜(cryo-EM)技术。 除此之外,也整理出了2016年最值得关注的几项技术,分别为:细胞内蛋白标记(Protein labeling in cells)、细胞核结构(Unraveling nuclear architecture)、动态蛋白质结构(Protein structure through time)、精准光遗传学(Precision optogenetics)、高度多重化成像(Highly multiplexed imaging)、深度学习(Deep learning)、亚细胞结构图谱(Subcellular maps)和综合单细胞图谱(Integrated single-cell profiles)。
功能强大的机器学习能力令计算器开始解决了一些感性问题,如图像和语音识别,而这也越来越多地被应用到了生命科学领域。这些深度学习技术,比如人工神经网络可以利用多重处理层检测庞大数据库中的模式和结构。
这其中的每一层都会从构建的上一层数据中学习到一个概念,越高层,学习概念越抽象。深度学习并不取决于事先的数据处理和自动提取特征。用一个简单例子来说明:一个任务为形状解释的深度神经网络,其第一层是识别简单的边,然而在之后的层中增加更加复杂的形状(由这些边组成),有多少层并没有什么硬性规定,但是大多数专家人物构成深度学习起码要超过两层。
近期一些研究展示了深度学习的作用,这种技术能单单根据DNA序列,就能衍生出基因组中的调控特征,如今年发表在Nature Methods杂志上的一篇文章指出,DeepSEA 可以输入基因组序列,串联出大规模项目(如ENCODE和表观遗传学路线等)的染色质图谱,预测出一些重要调控位点的单核苷酸变异的影响,这些调控位点包括脱氧核糖核酸酶DNase敏感位点,转录因子结合位点,和组蛋白标记位点等。
另一个方法:Basset 则能利用相似的深度神经元网络,预测单核苷酸多态性对染色质可接近性的影响;还有 DeepBind 能发现RNA与DNA上的蛋白结合位点,预测突变的影响。
深度学习能从非常庞大的数据库中挖掘到高水平信息,因此将在大数据分析中大显身手。特别是对于基因组分析,可以解决由于训练数据缺失依赖性和计算成本高造成的诸如过度拟合(overfitting)等问题。学术机构,还有一些新公司(如Deep Genomics)的研究人员去年发起了一项活动,倡导利用深度学习分析基因组,其目的在于预测遗传变异的影响——包括自然发生和由基因组编辑引起的变异,解析这对细胞调控的影响,以及由此引发的对疾病发展的影响。