查看完整版本请点击这里:
[分享]miRNA用荧光定量PCR检测的流程以及汇总
[分享]miRNA用荧光定量PCR检测的流程以及汇总 [转载][分享]miRNA用荧光定量PCR检测的流程以及汇总
目录如下:
一、MiRNA简介
二、反转录引物分类以及设计原理
三、引物探针设计
四、试验操作流程
1、RNA提取
2、反转录
3、荧光定量PCR
一、miRNA简介
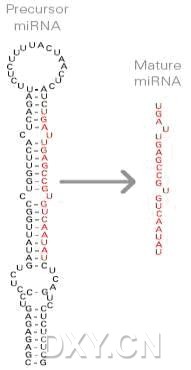
小 RNA是 19~28nt的调控RNA分子,主要包括微 RNA(micro RNA,miRNA)和小干涉RNA(short interfering RNA,siRNA)两类,其中的miRNAs成为继 siRNAs之后新的研究热点之一,名列2002年和2003年美国《科学》杂志评出的年度十大科学成就。
miRNAs是长片段RNA序列的一部分,同siRNAs一样是比较短小的单链小分子RNA,一般来源于染色体的非编码区域,由大约70 nt大小的可形成发夹结构的前体加工而来,它通过与其目标mRNA分子的3 端非编码区域(3-untranslated region,3 UTR)互补导致该mRNA分子的翻译受到抑制。
在miRNA公共数据库miRBase (cuturl('http://www.mirbase.org/'))中已经有1W多条来自不同物种的miRNA序列。
MiRNA检测方法
要了解miRNA在基因调控中扮演的角色,很关键的一个方法就是迅速、准确地定量检测miRNA基因的表达。因此,miRNA表达水平的检测也成为了科学家们研究的热点。但是由于小分子RNA是一类很小的分子,部分小分子RNA表达水平可能很低,因而需要极为灵敏而定量的分析工具。常用的检测方法有:
1.1 Northern blotting
1.2 核糖核酸酶保护分析以及基于此方法的液相杂交。
1.3 RT-PCR也被用来检测miRNA前体的表达水平, 其他基于PCR技术检测miRNA的方法有
引物延伸法,就是在引物的5 末端加一个特异标记,可以定量测定低丰度的RNA含量;
原位杂交技术(CISH,FISH)可以方便的检测miRNA的时空表达的差异。
1.4 芯片(microarrays)技术[46,47]是一种较快的研究miRNA表达的方法。
查看完整版本请点击这里:
[分享]miRNA用荧光定量PCR检测的流程以及汇总
[分享]miRNA用荧光定量PCR检测的流程以及汇总
91455110.jpg



最新回复
avi317 (2011-10-06 12:03:38)
Real-time PCR方法克服了由于miRNA分子太短(~22 nt)带来的定量最大难题而引入靶向特异性的反转录引物,该RT引物可以与成熟miRNA结合,形成反转录引物/成熟miRNA复合物,并在miRNA的5’末端延伸。这样就得到一个较长的反转录扩增因子,为进一步做实时定量PCR提供了符合要求的摸板。
RT引物有两种类型:
1、 Oligod(T)特异的RT引物(QIAGEN产品为主)
由特异序列+(T)20左右+兼并碱基V或VN组成。(所有miRNA可以公用一个Oligod(T)的RT引物,但是RNA在反转录前需要进行末端Poly(A)加尾)
2、茎环状结构的RT引物(ABI产品为主)
由可以自身呈环茎状的特异序列+6到8个miRNA3’端反向互补碱基组成。(一条miRNA序列特异对应一个茎环状结构的RT引物)
95787974.jpg
avi317 (2011-10-06 12:04:26)
由于反转录后得到的cDNA 为(miRNA+RT引物)复合片段。
上游引物可以在miRNA自身序列上找,如果GC含量太低,可以在上游引物5’端加入GCGCC等的保护碱基;下游引物在RT引物的反向互补序列中找;也就是说:上游引物是每个miRNA所特有的,下游引物为通用引物就可以了。
荧光定量PCR检测方法有SYBR Green染料法和TaqMan探针法。前者需要调整引物浓度以及引物扩增效率,把引物二聚体调整到越小越好;探针法则需要设计荧光探针,这其中由于MGB探针需要碱基数量少和特异性好的特点而被推荐(非广告)。
探针设计位置有3个:完全与miRNA序列相同、在miRNA与RT引物的交叉点、完全在RT引物上;这其中又有正向和反向互补两种情况。至于探针要设计在那个位置,根据自己试验情况而定,本人以为效果都差不多。
avi317 (2011-10-06 12:11:59)
1、RNA提取
在以上两种RT引物中,Ologod(T)特异引物所需要的RNA尽量是用特殊试剂盒提取的小RNA,而茎环状结构RT引物需要RNA正常提取就好。
另外由于所需样本不同RNA提取方法也不完全相同,普通组织样本和细胞可以研磨用Trizol+氯仿抽提;血液和植物样本需要前期处理后再用Trizol+氯仿或用专门的Kit抽提。
2、反转录
确认用Oligod(T)特异RT引物时,RNA需要进行3'Poly(A)加尾处理。
反转录的酶没有特殊要求,操作请按照各自反转录酶的说明书进行就好。这里特别注意的是:反转录过程中用到的RT引物(常规的是随机引物、Oligod(T)和特异引物)是前面提到的Ologod(T)特异引物和茎环状结构RT引物,它们分别属于Oligod(T)和特异引物范畴,在反转录中不需要另外添加其他RT引物。
在确定反转录温度中请特别注意您使用的是哪一种RT引物,而设定相应的反转录温度。
3、荧光定量PCR
优化PCR体系(引物浓度、Mg浓度、dNTP浓度、退火温度等),正常操作。
――――――――The End.――――――
@花开花落@ (2011-10-06 12:13:08)
@花开花落@ (2011-10-06 12:13:39)
131415 (2011-10-06 12:14:37)
avi317 (2011-10-06 12:15:26)
===========================================================================
我只是按照反转录酶(如Superscript 2)的相关特异引物的操作说明操作,开始是有65度加热的步骤。
您说的情况我没仔细想过,只是PCR产物3%胶的电泳条带来看还比较单一,应该没事。
avi317 (2011-10-06 12:17:54)
=============================================================================================================
我在设计U5基因和5s引物的时候也遇到这种情况,后来吧引物换了几条。我想您可以多设计几条引物看看。而且如果您用染料法的话,引物非特异扩增一定要降到最低。
98776langtao (2011-10-06 12:18:20)
年轮 (2011-10-06 12:19:41)
①为什么用stem-loop的方法反转录,需要测定一个miRNA就反转录一次?不能一次把要测的几个miRNA的stem-loop引物都加进去?
②为什么用poly(A)的方法要比用stem-loop节省RNA?
②用poly(A)的方法反转录,之后qPCR时,一次能检测几个miRNA?
问题比较傻,刚开始学。谢谢大家了!
年轮 (2011-10-06 12:19:41)
①为什么用stem-loop的方法反转录,需要测定一个miRNA就反转录一次?不能一次把要测的几个miRNA的stem-loop引物都加进去?
②为什么用poly(A)的方法要比用stem-loop节省RNA?
②用poly(A)的方法反转录,之后qPCR时,一次能检测几个miRNA?
问题比较傻,刚开始学。谢谢大家了!
avi317 (2011-10-06 12:20:13)
①为什么用stem-loop的方法反转录,需要测定一个miRNA就反转录一次?不能一次把要测的几个miRNA的stem-loop引物都加进去?
②为什么用poly(A)的方法要比用stem-loop节省RNA?
②用poly(A)的方法反转录,之后qPCR时,一次能检测几个miRNA?
问题比较傻,刚开始学。谢谢大家了!
==============================================================================================
1、如果你想一次反转录就得到所要的RNA,你可以吧几个loop引物放在一起,只是我没试验过,不知道分开好还是放一起好。
2、poly(A)的方法不比用stem-loop节省RNA,相反他需要更多的RNA。
3、如果用OligodT的反转录引物,你能检测你想要的miRNA,但是在反转录过程中,Oligodt的特异性要差一些。
avi317 (2011-10-06 12:20:58)
================================================================================================================
OligodT方法反转录我尝试过,但是由于在反转录前需要PolyA加尾,RNA需要的量比较多,后来就放弃了(我从血液里提取RNA,得量不高)。关于灵敏度我可以告诉你同样的量的RNA用Loop反转录引物会灵敏度大得多。特异性来说,OligodT的反转录引物比特异引物特异性要低一些,但是PolyA的OligodT方法一样能得到特异的PCR产物。相对来说增加PolyA一步骤的成本同反转录相比应该差不多(没仔细算过)
你可以尝试下两种方法:1、把多个你想检测的特异引物放在一起进行反转录(引物之间有没有干扰等影响我没尝试过);2、用OligdT引物进行反转录(如果你有比较多的RNA,)。
avi317 (2011-10-06 12:21:21)
也许用RNA Poly(A)加尾后进行PCR会出现PCR产物没有目的条带的情况,
希望在做目的基因时同时做下内参基因(U6或者5srRNA或者其他),如果内参基因也没有出现条带,说明RNA本身或者在反转录前RNA消耗掉了,或者反转录过程中出现问题没有得到理想的cDNA;如果内参有条带或有曲线,就从目的基因引物开始分析。
caihong (2011-10-06 12:21:49)
蛐蛐儿 (2011-10-06 12:22:17)
avi317 (2011-10-06 12:22:53)
如果使用的是自己设计的RT引物,则我建议选择用AB等公司的成品作为阳性对照,这样在实验过程中可以少走很多弯路。
avi317 (2011-10-06 12:24:14)
================================================================================================================
其实用什么方法都可以的。我一般是先用染料法验证RT引物和PCR引物怎样,然后再设计探针的。
如果你用染料发做不出来,原因你应该找一找,我想这跟用探针或者染料没什么关系。PCR产物跑过琼脂糖凝胶电泳没??目的条带怎样?我想您可以这样试下:1、先用普通PCR跑电泳看看有没有条带,条带好不好?不好就需要找原因、优化 2、条带好的话,然后用染料法做,这样保证PCR体系本身没什么问题 3、如果你觉得探针法特异性更好(事实确实如此),在以上条件的情况下可以设计探针了
avi317 (2011-10-06 12:25:34)
=======================================================================================
您好,您可以找找楼上我提供的两篇文章,分别是关于以上两种RT引物的序列的。也许可以给您一些建议和帮助。
碧空子 (2011-10-06 12:26:44)
1.你在前面的帖子说,加A法比茎环的方法耗费的RNA量更多?但在反转录体系中加入的RNA量增大是可以提高检测灵敏度的,也就是说虽然加入了更多量的RNA,但实验的灵敏度得到了提高,所以不能直接说加A法比茎环法消耗的RNA量更多的。
2.对于茎环法的特异性,因为现在准备开展这方面的实验,也在这两种方法中犹豫。LZ现在的实验数据能够证明茎环法反转录的特异性比加尾法的高吗?虽然按照原理来说,在反转录阶段茎环法有反转的特异性而加A法没有,但我听说茎环法由于引物存在茎环结构,具有空间位阻的情况,因此转录效率比加A法低。
3.茎环法的引物如果购买的话,大概多少米啊?
[分享]miRNA用荧光定量PCR检测的流程以及汇总