物理所蛋白质晶体学中的直接法研究取得新进展
中科院物理所范海福院士等研究人员几十年来从事蛋白质晶体学中直接法的研究,近年来又取得了重要进展:2004年提出基于直接法的“双空间SAD(单波长异常衍射)相位迭代推演”方法;2006年又提出基于直接法的“双空间MR(分子置换)结构模型迭代扩展”方法。这两种方法显著地提高了原有SAD和MR方法的效能(见附图)。
测定蛋白质晶体结构,是研究生物大分子结构与功能之间关系所不可缺少的实验基础。SAD法和MR法是当前测定蛋白质晶体结构的主要方法。如果待测蛋白质有可供参照的、结构已知的类似物,通常都使用MR法求解;否则,多用SAD法求解。近年来登录到国际蛋白质数据库中的新蛋白质晶体结构,约有80%是使用上述两种方法之一测定的。
物理所从1965年开始研究将直接法与SAD(单波长异常衍射)或SIR(单对同晶型置换)法相结合以测定蛋白质晶体结构。这一研究方向在上世纪八、九十年代,发展为国际晶体学直接法研究的一个热点。世界上主要的直接法研究单位大都参与了这一研究,其中包括两个由诺贝尔奖得主领导的小组。1984-1985年间,物理所的晶体结构分析方法研究小组对自己1965年提出的方法作了重大的改进。此后,他们对于直接法与SAD或SIR方法相结合的研究一直具有重要的国际影响。
当前直接法在测定蛋白质晶体结构中的应用有三大类:一、测定蛋白质晶体的重原子亚结构;二、从头测定蛋白质晶体结构;三、与传统蛋白质晶体学方法相结合以破解原有方法中的“相位模糊”问题。其中第一类并不解决蛋白质的整体结构;第二类对实验数据要求很高,在已经测定的蛋白质晶体结构中只有5%左右可以满足其要求;第三类则适用于大多数情况。物理所的工作属于第三类。范海福课题组以自己的研究成果为基础编写的电子计算机程序OASIS是国际上用于执行第三类直接法的最重要的程序。OASIS程序的第一个版本发布于2000年,当年就被国际上使用最广泛的蛋白质晶体结构分析程序库CCP4收录,成为其中用于推演蛋白质SAD或SIR相位的、唯一的直接法程序。OASIS程序的第三个版本OASIS-2006包含了2004年和2006年两项重要的方法研究成果。2008年CCP4的最新版本已将其收录并用以替换原有的OASIS程序。另外,欧洲分子生物学组织EMBO的汉堡分部建立了一个蛋白质结构自动解析网络平台Auto-Rickshaw。该平台从2006年起采纳了OASIS,使这个平台从原来单向运作的模式变成可以循环迭代的模式。发表于J. Mol. Biol. (2008). 383, 49-61上的一篇论文,包含了关于Auto-Rickshaw自动解析蛋白质复合物DctBp/SIN晶体结构的记述:在使用OASIS之前,Auto-Rickshaw可以自动构建出相当于整体结构57%的模型;加入OASIS之后,建模比例提高至89%。
OASIS自发布以来,已有中、英、美、法、日、德等国的研究单位用来解决了多例其它程序难以解决的蛋白质结构分析问题。2005至2008三年间,范海福和同事们先后6次应邀在重要的国际学术会议和讲习班上介绍OASIS程序的原理和实际操作。目前,OASIS程序还在不断的更新之中,2009年内该研究小组将要发布一个性能和自动化程度都有明显提高的新版本。
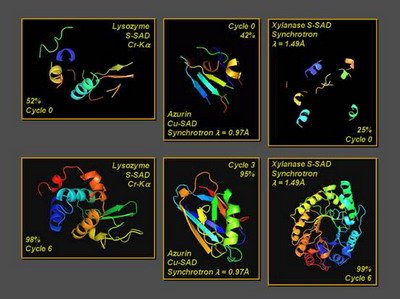
图一、基于直接法的双空间SAD相位迭代推演
[Acta Cryst. (2004) D60, 1991-1996]
图中自左至右三个蛋白质Lysozyme, Azurin和Xylanase,用第一版的OASIS (2000)联合DM, RESOLVE, ARP/wARP等程序求解,所得结果示于图的上半部;如改用OASIS-2004联合DM, RESOLVE, ARP/wARP等程序进行“双空间SAD相位迭代推演”,由此自动构建的三个结构模型(示于图的下半部)其完整度都有成倍的提高。
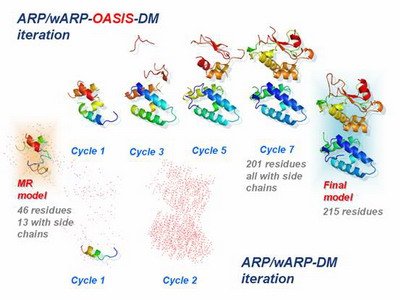
图二、基于直接法的双空间MR结构模型迭代扩展
[Acta Cryst. (2007) D63, 793-799]
图中所示为蛋白质复合物E7_C–Im7_C的MR模型迭代过程。左边是由分子置换(MR)法求得的部分结构模型;右边是经过精修之后的“最终”模型。从MR模型出发用ARP/wARP-DM迭代,所得结构模型逐轮退化如图底部所示。及至第三轮还出现运行错误。但是,在ARP/wARP和DM之间加入OASIS-2006,则可以自动构建出相当于整体结构90%以上的结构模型,如图顶部所示。
-
焦点事件
-
并购