在单细胞水平预测DNA甲基化的新方法
如今,DNA甲基化的检测已经达到了单细胞的分辨率。不过,目前的方法还不能完整覆盖CpG,总是留下一些缺口。英国的一个团队最近开发出一种计算方法,可准确填补这些缺口。这项结果发表在《Genome Biology》杂志上。
研究人员使用所谓的深度神经网络策略来开发他们的方法,并命名为DeepCpG。他们将这种方法应用在几种小鼠或人类细胞的单细胞DNA甲基化数据上,证明DeepCpG在预测DNA甲基化上比现有的策略更佳。
这篇文章的通讯作者是欧洲分子生物学实验室的团队负责人Oliver Stegle。他在文中写道:“对于所有的细胞类型,DeepCpG能够比以往的方法更准确地预测甲基化状态。这种方法同时也揭示了已知和de novo序列motif,它们与细胞之间的甲基化变化存在关联。”
DeepCpG将标有序列线索的DNA模块与双向门控递归网络相结合,此网络追踪基因组中CpG位点的甲基化是否存在。所得到的的“联合模块”将学习DNA-CpG相互作用,以便产生普遍预测DNA甲基化的模型。
研究人员指出,一旦接受了适当的训练,DeepCpG就能完成各种任务,包括预测粗略测序细胞中的甲基化,以及鉴定细胞中的DNA motif,它们通常与特定的甲基化状态或细胞之间的甲基化改变相关联。
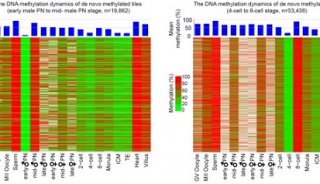
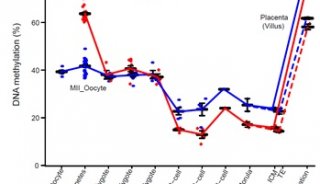
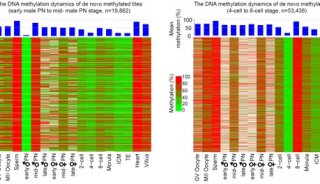
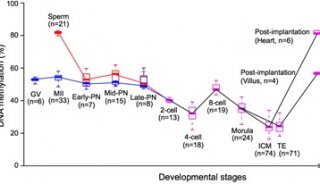
之后,研究人员将DeepCpG方法用在18个小鼠胚胎干细胞的单细胞亚硫酸氢盐序列数据上。他们发现,无论是在全基因组范围,还是在不同的基因组背景,这种方法都能够比其他方法更准确地预测DNA甲基化。
团队随后又将DeepCpG用在数十个小鼠或人类细胞上,这些细胞要么经历过单细胞亚硫酸氢盐测序(scBS-seq),要么经历过简化的测序(scRRBS-seq)。他们研究了其甲基化预测的性能,以及将特定的DNA motif与胞嘧啶甲基化特征相关联或评估DNA突变的甲基化影响的能力。
“DeepCpG发现的一些motif与参与DNA甲基化调控的已知motif相匹配,”作者在文中写道。“它可以发现的motif实际上取决于所研究的细胞类型以及驱动甲基化变化的潜在因素。”
-
科技前沿
-
企业风采

-
科技前沿

-
科技前沿