三篇文章为蛋白质组学研究提供重要信息
由EMBL等处的研究人员公布的三篇文章对大规模蛋白质组学的数据分享和信息比较方面提出了新的方法见解,令这些数据能更易被科学家们分享使用,这些文章发表在8号和26号的《Nature Biotechnology》杂志上。
这三篇文章中的两篇作为Perspectives papers公布在杂志上,可免费获得,主要是提出了蛋白质组学和分子相互作用实验的公布要求,并讨论了这些方法的执行(mplementation),影响(impact)和益处(benefit)。最后一篇文章,公布在26号的杂志上,提出了这些标准如何不仅对于研究人员而言有帮助,而且也通过更详细和完整的信息来源有利于更广泛的交流。
原文检索:
Nature Biotechnology 25, 887 - 893 (2007)
Published online: 8 August 2007 | doi:10.1038/nbt1329
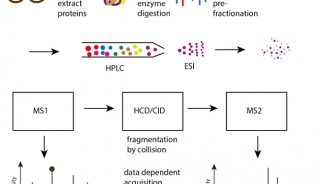
The minimum information about a proteomics experiment (MIAPE) [Abstract]
Nature Biotechnology 25, 894 - 898 (2007)
Published online: 8 August 2007 | doi:10.1038/nbt1324
The minimum information required for reporting a molecular interaction experiment (MIMIx)
[Abstract]
随着可获得的生物学信息的数量,以及公共数据库的增加,这些数据的一致性就成为了比较,整合和交流的关键点。作为欧洲生物学数据的主要来源,EMBL——欧洲生物信息研究所(EBI)提出了应用于其自身数据库,比如ArrayExpress(生物芯片及基因表达数据),IntAct(分子相互作用数据),以及PRIDE(实验证据及发表文章相关的蛋白鉴别)的统一标准。
蛋白质组学实验最小信息(The Minimum Information About a Proteomics Experiment,MIAPE)和分子相互作用最小信息(Minimum Information required for reporting a Molecular Interaction Experiment,MIMIx,生物通编译)指南分别为蛋白质组学和分子相互作用数据提出了信息归类。这些标准可以减少不明确性,捕获一个实验中所有的必要信息(包括生物学和方法学内容),从而为其他人研究这些数据提供更深水平的理解。
文章的共同作者,EMBL-EBI的Henning Hermjakob表示,“通过获得‘agreed minimum reporting standards’,我们可以获益于这些更容易的识别方法,以及与我们研究相关领域最多的信息。下一步就是提供免费的,可获得的,最高质量的数据库。”
Setting the standards
MIAPE和MIMIx,这两种reporting requirements,由科学团体发展出来,用于减轻个体研究人员的负担,其中MIAPE提供了一种作为个体模块,可以覆盖整个工作流程——从研究设计到统计数据分析。而作为第一个finalised module,MIMIx指南则能将普遍的MIAPE规则用于描述分子相互作用,以及提供了数据积累方面的建议。
Standards in action
这两套requirement由Bantscheff等人在其一个大规模方法描述蛋白激酶与小分子抑制剂之间的相互作用上得到了实际的应用。Cellzome研究人员利用这一方法验证三种药物的药效——这三种药物共有一个特殊的激酶靶标,而且也识别新的药物靶标,这些绑定的信息帮助解释了药物结合特异性和信号途径的下游效应。
MIAPE和MIMIx适用的质谱分析(mass spectrometry)和相互作用数据都被纳入了EBI的PRIDE,IntAct和ChEBI数据库,研究报告中新增的内容也可以提供直接的蛋白组学信息,质谱和分子相互作用数据。相互链接的EBI数据来源不仅能支持直接的实验结果,而且也将它们与更广泛的生物学内容联系在了一起,比如将已识别的激酶与UniProt数据库的已知功能连接起来。
-
精英视角

-
会议会展

-
综述
-
焦点事件
-
焦点事件

-
技术原理