张锋教授Nature子刊发文:编辑人类基因组需谨慎!
过去的几天里,CRISPR领域又带来了一波吸引眼球的新闻。上周,MIT科技评论发布了一项重磅消息,俄勒冈健康与科学大学的Shoukhrat Mitalipov使用了CRISPR基因编辑技术首次在美国进行胚胎基因编辑(附:原文链接)。而紧接着,CRISPR技术先驱张锋便在Nature Medicine上刊文,从人类遗传多样性的角度,呼吁谨慎将基因编辑技术应用到人体。
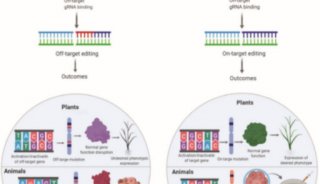
先简单介绍一下CRISPR基因编辑技术的原理。这一技术需要两类关键成员,一类是能够剪切DNA的酶(如Cas9和Cpf1),另一类则是负责将这些酶带到特定区域的向导RNA(gRNA)。在这两类成员的作用下,CRISPR系统能有效地对研究人员选定的基因组位点进行高效、精准的剪切,使得基因组编辑的大规模应用成为可能。人们畅想,使用CRISPR基因编辑技术,我们或许很快就能修复患者的基因,使他们健康生活。
但张锋教授发表的这篇文章则提醒我们,对人类基因组的编辑不宜过于草率。他指出,人类的遗传信息具有多样性。这不但会影响到靶向特定序列的可靠性,还会影响到对酶的选择。此外,由于人与人之间的遗传信息有着明显差异,每个人也都面临着不同的“脱靶风险”(即在意料外的基因组区域进行编辑)。
张锋教授发布在Nature Medicine上的分析指出,通过全基因组测序技术对患者进行预先筛选,而后通过实证研究整合信息,这样指导RNA的选择可以帮助研究人员设计出有效安全的CRISPR治疗技术。
“与传统药物研发只需要开发出药物针对患者高度保守的靶点不同,基因水平的治疗需要面对个体间巨大的遗传差异。”张锋教授谈到。“遗传变异能混淆某些Cas内切核酸酶对靶点的识别,如果恰巧遗传变异的出现破坏了治疗靶点,那就会对CRISPR相关治疗的功效产生极大的影响;如果遗传变异产生了靶标外的治疗位点,则会对CRISPR相关疗法的安全性产生极大的影响。”
为此,张锋教授及相关研究人员分析了近期发布的ExAC数据集中60706个个体以及千人基因组计划数据集中个体的遗传变异数据,用于确定遗传变异对基因编辑技术,如SpCas9、SpCas9变体VQR和VRER、SaCas9以及AsCpf1的可能影响。
他们发现,遗传变异对每种酶的功效都可能产生影响,并造成患者特异性的脱靶。事实上,ExAC数据集中的变异涵盖了约93%~95%的基因编辑靶标,因此遗传变异很可能造成靶点剪切功效的降低,张锋教授补充道,“这支持了他们之前的观点:应增加治疗相关基因座可攻击的靶点数量,以此来增强具有明确靶DNA序列的编辑酶的安全性和功效。
此外,他们注意到,群体中多种单体型的存在也增加了脱靶位点的数量,让情况变得复杂。因此,他们利用千人基因组计划的数据集来鉴定人群中脱靶位点最少的靶点。“特异性增强的eSpCas9和Cas9-HF1酶的使用将进一步降低脱靶位点的切割,不过仍然需要避免重复区域或高频率单体型中的脱靶位点,”张锋写道。
张锋教授还指出,千人基因组计划数据集中每个个体的人口统计学信息(包括性别和祖先)同样也可以用来研究已知个体遗传变异带来的脱靶风险。他们发现,“在治疗前应对每个患者进行全基因组测序,以便选择一种与患者基因组完全匹配并且不涉及患者特异性脱靶位点的向导RNA酶组合进行治疗。”
基于一些研究人员和公司已经开始设计CRISPR相关的临床试验,这种类型的分析将尤为重要。张锋教授认为,如果他们没有考虑到遗传变异,他们的试验将存在巨大的风险。
-
焦点事件

-
焦点事件

-
项目成果