用于显示和诊断分析DNA 序列踪迹变体的高精确、高灵敏、高速度的方法
摘要:介绍了一种利用新技术开发的软件工具,它的设计符合各种新变体显示研究成果和诊断实验室对于灵敏度、准确度的要求,此次合作研发的新软件可以将病人的DNA序列与参考序列进行实际点对点的物理比较,这项技术提供了以前所不能达到的灵敏度、准确性,并且在降低了假阳性几率的同时提高了分析速度。
Enhanced Accuracy, Sensitivity, and Speed of
DNA Variant Detection of Sequence Traces for
Discovery and Diagnostics Analysis
Chang Sheng Liu
Abstract: A software tool that utilizes new technologies designed to meet the sensitivity and accuracy requirements of DNA variant discovery research efforts and diagnostics laboratories is described. The result of a collaboration between academe and industry, the software provides a new generation of tools that performs an actual point-to-point physical comparison of patient DNA sequence traces to a reference trace and monitors the mobility of the DNA. This provides previously unobtainable sensitivity, accuracy, and low false-positive rates, with the added benefit of high analysis speed.
科学正处于人类基因和医药学重大进展的开端。目前人类基因图已经完整,个体中致病变异的识别对于生命的质量具有重大的推动作用。然而由于缺少合适DNA测序的分析工具,DNA变体的识别仍处于停滞不前的状态。
分析工具的缺乏使得研究工作者不得不采用准确度稍差的定性技术进行DNA测序,例如:变性高效液相色谱(dHPLC)、单纱结构多态性(SSCP)和温度梯度毛细管电泳或采用既费时又准确度不高的手动可视序列踪迹分析。
另外,已有的序列分析设计是为了迎合人类基因组计划的要求并解决难以获得的毛细管电泳数据。尽管大家竭力利用这项技术进行DNA变异显示,但结果都不太理想。主要是因为碱基判定(base-calling)程序所固有的不准确性(Phred 20的工业标准为99%准确率或1%误差)不能满足DNA 变异显示的要求( 平均发生在1/ 1300碱基或0.08%,比base-calling 精度低)DNA变异对遗传变异平均发生在1到1300碱基之间,对肌体变异变1到100万碱基之间(环境引起的),这种变异发生在特定的细胞组织如癌中。
意识到对变异显示缺少合适的分析方法所产生的不良影响,JohnsHopkins大学(Baltimore,MD)Sidney Kimmel Comprehensive Cancer Centre肿瘤学副教授、肿瘤遗传学主任Victor Velculescu博士和SoftGenetics, LLC (State College, PA) 利用新技术合作开发一种分析工具,它的设计符合变异显示研究成果和诊断实验室对于灵敏度、准确度的要求。此次合作研发的新软件可以将病人的DNA序列与参考序列进行实际点对点的物理比较,监控DNA的变化,这项技术提供了以前所不能达到的灵敏度和准确性,并且在降低了假阳性几率的同时提高了分析速度。
软件的设计减少了人的显著交互作用及其他软件程序的安装,毗连(序列)群构成和修整分析只需要两个基本的步骤:输入所分析的序列数据、确定发现的变异。直接点击两个图标,软件将自动形成contigs, aligns and traces进行突变分析。软件大约在90s中能够处理400条通道。
软件提供彩色编码和数字记录系统,直接将任何变异展现在分析者眼前以便进行监控和确认,从而使用者不必观测电泳谱图上的每一个碱基。当进行双向序列踪迹数据分析时,准确率超过99%,比以前使用的变异识别方法的准确率高20%。
软件Mutation Explorer 和Mutation Surveyor,运用几种数学运算法则,可以模拟临界因子运用到手动可视序列踪迹数据(即荧光核苷峰强度、瞬时碱基叠加[线性位置]、相对强度降低和系统噪音[仪器基线噪音]),检测并提供一个可靠的手段,以发现在变异电泳谱图上的差异(参比与样品之间的差异),这样就可以通过手工或仪器方便地进行鉴定识别。
合作者们测试软件的的结果表明准确率超过99%。合作者们确认进行单向测序时软件的准确率大于95%,当使用双向测序时准确率可以超过99%。经计算,分析Phred 20quality主峰程序时,灵敏度可达12.5%,利用Phred 20序列踪迹时,假阳性的的几率低于50 ppm。
1 发现策略
软件提供的高效快速分析使研究者能执行新的发现策略,例如:整个基因的变异分析[1,2]或制定病人的治疗方法。这些在以前都是很耗时的。
2 分析能力
共同研究的另一目标是分析软件包能够灵活简单地提供适应当天研究环境的分析能力。由WindowsTM操作系统(Microsoft, Redmond, WA)发展的程序并遵循直观逻辑路径、能在Pentium 3或等效台式电脑及拥有可用于存储的512 RAM的laptop系统上操作。程序可以独立运行,每8h可处理106碱基对序列。由于操作程序只需两个基本步骤,分析者在几小时之内即可精通程序的操作。Mutation Explorer和Surveyor 均可以设定程序全自动运行以进行数据的输入与分析。
数据输入后软件将自动形成毗连(序列)群、进行校准、整理数据,在90 s内给出分析报告,报告单元直接链接到踪迹电泳图上,点击电泳图,分析者将进入确认界面,分析者不需要浏览和比较软件给出的整个电泳图,在软件变异电泳图上显示为尖峰,变异确认后,数据以几个表格的形式给出或打印出诊断报告。整个电泳图可以打印到一个报告表中,使用者也可以自制个性化的报告。
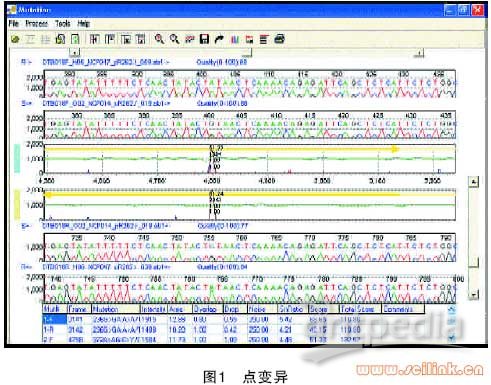
3 点变异和插入/ 删除(indels)
对于点变异(图1),在产生变异电泳图中尖峰明显地标记出变异部位。这样使用者就不必检查序列踪迹上的每一个峰。当分析双向数据时,准确率超过99.5%,并且在每106碱基的分析中假阳性低于50。此方法在90s内可以处理400组数据。
图1中上方的3个长条图是正向序列踪迹,下方的3个长条图是反向序列踪迹。上方和下方的踪迹是正向和反向的参照踪迹;第二与第五个长条图为病人的踪迹,中间的两个长条图显示变异,以很容易识别的尖峰的形式给出。
Indel检测(图2)是通过监测与参照相比较的DNA片断的迁移时间完成的,与此同时按参照踪迹不断重新排列片断。软件检测indel是从1到100个碱基对进行的,中心的长条图从绿色变为红色,其中indel点在变异电泳图中显示为深红色的线。
4 检测灵敏性
Texas大学健康中心(Dallas,TX)的Yidong Bai 博士经独立研究发现,程序的检测灵敏性对于异质接合体的主峰检测大于5%;另外软件可以在正常细胞中以大约8:1的比例定位癌细胞(图3),它也可以在同一波长和空间位置下检测埋藏在基线噪音中的正向和反向峰,定位新的未发现的变异。
5 hetero-indels 的去卷积
在含有样品的异质接合体indels 分析中,软件进行自动去卷积检测,从1 到100 碱基对的indel 可以从Phred 20 序列踪迹去卷积,而不需要手动处理数据。图4中上方长条图是wild-type 参比;第二个长条图表示包括异质接合体indels 的样品序Chang Sheng Liu :用于显示和诊断分析DNA 序列踪迹变体的高精确、高灵敏、高速度的方法列踪迹;第三个踪迹为反向样品踪迹或样品的踪迹,第四个踪迹是样品去卷积突变株部分或次要踪迹;第五个踪迹为突变株样品踪迹紧接着去卷积和灵活性校正,第六或最后一个踪迹trace 是变异电泳图,表示indel点和indel内容。
6 结论
本文所描述的软件是DNA 变异??变异点和indel 点--在易操作的模式下快速定位的强有力工具。软件加快了变异发现的处理,非常适合诊断应用。
参考文献
- Bardelli A, Parsons DW, Silliman N, et al. Mutational analysis of the tyrosine kinome in colorectal cancers. Science 2003; 300(5621):949.
- Samuels Y, Wang Z, Bardelli A, et al. High frequency of mutations of the PIK3CA gene in human cancers. Science 2004; 304(554).
TAG: