布鲁克发布PaSER重磅更新,强大新算法支持dia-PASEF

布鲁克PaSER™ 2022软件加入TIMS DIA-NN模块,具有处理dia-PASEF®数据的能力
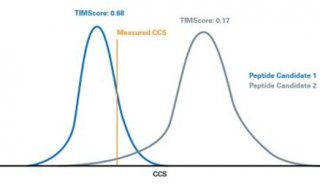
创新的TIMScore算法支持dda-PASEF数据和dia-PASEF库,与TIMS DIA-NN相结合,仅需35分钟梯度即可从200ng K562裂解液中鉴定约9000种蛋白质组
TIMScore能将磷酸化肽段鉴定结果进一提升约25%
美国南卡罗来纳州查尔斯顿 -2022年2月28日 -在美国HUPO会议上,布鲁克公司(纳斯达克代码:BRKR)宣布了其TIMS DIA-NN软件,可用于高通量、深度和无偏的4D-蛋白质组学工作流程。它采用了现在最新的TIMScore算法,该算法利用机器学习(ML) 来预测胰蛋白酶裂解肽段和磷酸化肽段的CCS值。传统搜索算法会因低信噪比和嵌合谱干扰造成的不确定性,导致肽谱无法成功匹配,但通过TIMScore算法将测量的CCS值与CCS值进行参考比对,可以得到最可能的匹配结果。该软件是在2021年收购的IP2软件的基础上进行开发,这些功能可在基于GPU运行的PaSER 2022新版本软件中实现。
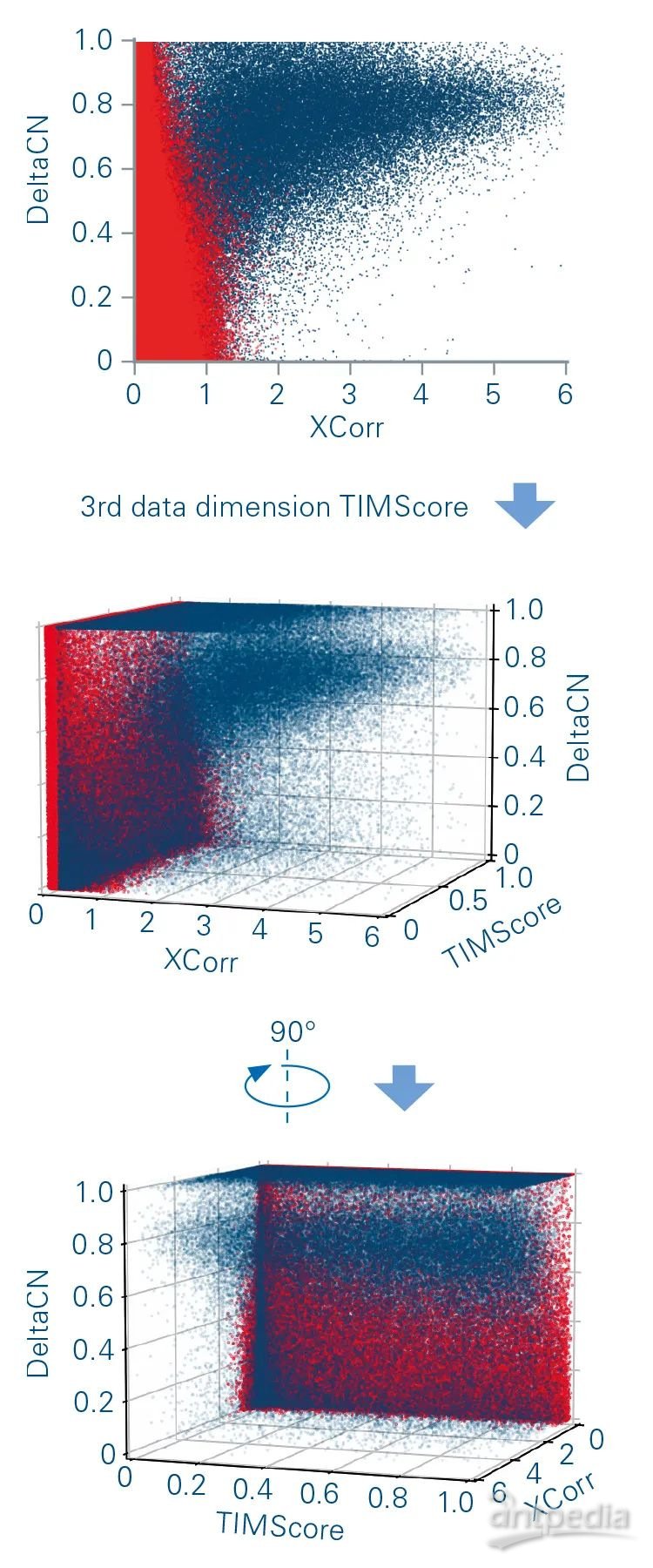
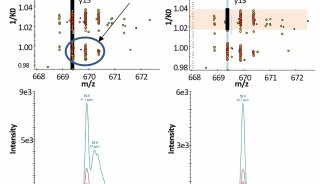
图:启用 CCS 的原数据库(target,蓝色)/反向数据库(decoy,红色)搜索的3D箱线图。额外的第3个维度减少了蛋白质鉴定的不确定性,并提供更精确和准确的肽谱匹配结果。
布鲁克·道尔顿生物信息学项目经理Dennis Trede博士解说:“我们采用成千上万上胰蛋白酶裂解肽段和磷酸化肽段数据集作为机器学习的训练数据,其中包括双电荷、三电荷和四电荷价态的肽段,开发了一个肽段CCS的转换模型。该模型对胰蛋白酶裂解肽段的预测准确率高达95%,对磷酸化胰蛋白酶裂解肽段的预测准确率高达92%,这是普遍最具有生物学意义的翻译后修饰。随后,我们通过使用已发布的数据集(Ogata 等人),将识别的肽段数量从42,930提高到98,949,这比最初发布的多近3.5倍。TIMScore信息库为dia-PASEF工作流程奠定了基础,从而可以更深入地研究蛋白质组,并具有高通量和高灵敏度的优势,从而大规模地获取更多的序列和更广的翻译后修饰覆盖范围。”
DIA-NN的共同发明者之一、德国柏林Charité爱因斯坦生物化学教授Markus Ralser说:“在与布鲁克的持续合作中,我们将DIA-NN定制成流线型处理工具并放入以CCS为核心的dia-PASEF数据处理中。它也可以简化并加速数蛋白质的鉴定,即使在非常短的梯度中,也可以很容易地定量数千种蛋白质。我们很高兴在与布鲁克密切合作中,推出了供应商集成版本,即TIMS DIA-NN,并将其变成PaSER生物信息学平台的一部分。”
布鲁克蛋白质组学生物信息学架构师Robin Park博士补充说:“由于我们的深度学习(DL)训练模型还包含磷酸化肽段数据,TIMScore将磷酸化肽谱匹配(PSM)和肽段鉴定结果在数据集层面平均提高了61%。更令人兴奋的是,使用严格的5% FLR卡值确定的磷酸化肽段鉴定结果增加了57%。由于翻译后修饰的不稳定性、化学计量或其优先解离性,翻译后修饰的位点定位仍存在困难;随着更多训练数据的获取,我们也将在TIMScore算法中添加更多翻译后修饰。”
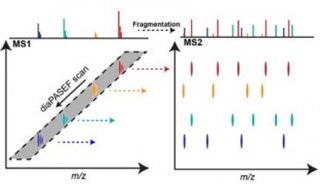
PaSER 2022包括TIMScore算法和TIMS DIA-NN,可用于处理来自timsTOF Pro 2、timsTOF SCP和timsTOF fleX系统的dia-PASEF数据。
在实时质谱采集中,PaSER将同样的数据流机制应用于dia-PASEF,并能在在采集运行结束时触发谱图库搜索,实现“运行即完成”(Run and Done)以支持高通量4D-蛋白质组学流程。
参考文献
Demichev, Nat Methods. 2020: https://dx.doi.org/10.1038%2Fs41592-019-0638-x
Demichev, bioRxiv 2021: https://doi.org/10.1101/2021.03.08.434385
Ogata K, Chang CH, Ishihama Y (2010). Effect of Phosphorylation on the Collision Cross Sections of Peptide Ions in Ion Mobility Spectrometry. Mass Spectrom (Tokyo); doi:10.5702/massspectrometry.A0093.