Nature | 单密码子分辨率翻译测序新方法—scRibo-seq
单细胞测序的方法对于不同的生物体、不同组织以及不同细胞种类达到了更为深入的探究。这些工具集中于对单细胞的基因组【1】、表观遗传组【2】以及转录组进行分析【3】。但是现今想要在单个细胞中进行翻译过程的测量并非易事。
2021年9月8日,荷兰皇家艺术和科学学院与乌得勒支大学医学中心Oncode研究所Alexander van Oudenaarden研究组以及Michael VanInsberghe(第一作者)在Nature期刊上发表Single-cell Ribo-seq reveals cell cycle-dependent translational pausing,构建了能够达到单密码子精度的翻译测量测序技术scRibo-seq,并且通过该技术对细胞周期依赖的翻译暂停特点进行了揭示。

近年来,关于单细胞水平、翻译过程的检测的技术有一定的进展,比如在单细胞分辨率上进行的质谱分析技术SCoPE-MS【4】。SCoPE-MS技术可以对小鼠胚胎干细胞分化过程中的一千多种蛋白进行量化。但是,分辨率还不够。由此,scRibo-seq技术应运而生。
scRibo-seq技术是将核酸酶足迹与小RNA文库构建、尺寸富集技术结合起来,以测量单细胞中的翻译动态变化过程(图1)。单个活细胞被分选到含有放线菌酮(Cycloheximide)的裂解缓冲液中,以稳定和停止转录本上的核糖体,随后暴露的RNA被微球菌核酸酶MNase消化,由此产生核糖体保护足迹(Ribosome-protected footprints,RPFs)。这些足迹通过连接包含独特分子标识符(Unique molecular identifier,UMI)和引物位点的适配子,转化为测序库,用于后续的cDNA合成和PCR扩增。最后,对PCR翻译过产物进行尺寸富集,选择符合典型核糖体保护足迹的产物进行测序。
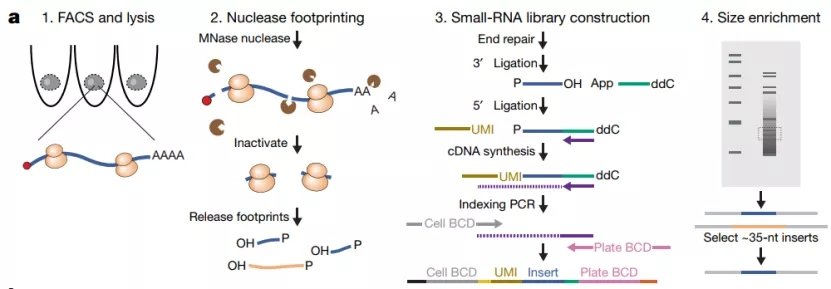
为了验证该方法的有效性,作者们首先对HEK293T细胞以及hTERT RPE-1中scRibo-seq技术的应用效果进行检测。结果证明该方法能够有效的显示蛋白编码转录本的5’UTR、CDS以及3’UTR区域的核糖体分布。先前的研究表明在培养基移除后细胞中的核糖体会在某些关键的密码子上停顿,这一停顿是受到密码子类型影响的【5,6】。为了进一步验证scRibo-seq测量翻译动力学的能力,作者们从HEK 293T培养基中去除精氨酸和亮氨酸的不同时间点后再进行细胞分选和检测,发现核糖体的停顿的确是不同处理依赖性的暂停。比如,精氨酸的缺失会造成CGC以及CGU密码子上足迹的频率升高,而去除培养基中的亮氨酸则不会有此现象。而UAA密码子上核糖体占位的增加则只在亮氨酸饥饿的情况下出现。因此,该结果证明了scRibo-seq技术的准确性和有效性。
随后,作者们想知道细胞周期的状态是否会对细胞响应氨基酸限制具有影响,所以作者们将scRibo-seq技术与FUCCI(Fluorescent ubiquitination-based cell cycle indicators)技术进行联用,对不同细胞周期的细胞进行分选和收集。作者们发现核糖体的活性位点的确受到细胞周期的显著影响。
在体外培养的细胞中证明了scRibo-seq的作用之后,作者们希望对原代小鼠内分泌细胞(Enteroendocrine cells)中的应用进行检测。EECs细胞群在胃肠道细胞中所占的比例极低,数量少于细胞总量的1%,会在营养刺激下产生和分泌不同的激素【7】。通过scRibo-seq检测,作者们根据已经建立的激素细胞标记物基因的翻译特征鉴定发现了8个细胞类型,并对EECs细胞中的密码子特异性核糖体停顿进行了鉴定。因此,该结果说明scRibo-seq可以直接应用于原代细胞样品,并对稀有细胞群的翻译动力学过程进行测量。
总的来说,该工作建立了用于检测单密码子精度的翻译过程的高通量测序方法scRibo-seq,填补了目前单细胞基因组学方法的关键空白。另外,由于scRibo-seq技术不依赖于标记物以及转基因体系,可以直接对核糖体谱进行分析,其灵敏度和分辨率相较于先前的方法都更胜一筹。未来,该技术将会得到进一步广泛的应用,可以用于揭开高度动态系统中罕见细胞群的转录特征。
原文链接:https://doi.org/10.1038/s41586-021-03887-4
【参考文献】
1. Navin, N. et al. Tumour evolution inferred by single-cell sequencing. Nature 472, 90-94, doi:10.1038/nature09807 (2011).
2. Smallwood, S. A. et al. Single-cell genome-wide bisulfite sequencing for assessing epigenetic heterogeneity. Nature methods 11, 817-820, doi:10.1038/nmeth.3035 (2014).
3. Tang, F. et al. mRNA-Seq whole-transcriptome analysis of a single cell. Nature methods 6, 377-382, doi:10.1038/nmeth.1315 (2009).
4. Budnik, B., Levy, E., Harmange, G. & Slavov, N. SCoPE-MS: mass spectrometry of single mammalian cells quantifies proteome heterogeneity during cell differentiation. Genome biology 19, 161, doi:10.1186/s13059-018-1547-5 (2018).
5. Darnell, A. M., Subramaniam, A. R. & O'Shea, E. K. Translational Control through Differential Ribosome Pausing during Amino Acid Limitation in Mammalian Cells. Molecular cell 71, 229-243.e211, doi:10.1016/j.molcel.2018.06.041 (2018).
6. Subramaniam, A. R., Pan, T. & Cluzel, P. Environmental perturbations lift the degeneracy of the genetic code to regulate protein levels in bacteria. Proceedings of the National Academy of Sciences of the United States of America 110, 2419-2424, doi:10.1073/pnas.1211077110 (2013).
7. Gribble, F. M. & Reimann, F. Enteroendocrine Cells: Chemosensors in the Intestinal Epithelium. Annual review of physiology 78, 277-299, doi:10.1146/annurev-physiol-021115-105439 (2016).







