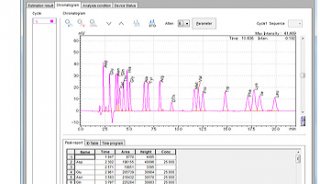
基于PCR评估sgRNA特异性的方法
CRISPR/Cas9已经成为一种通用的基因组工程工具,依赖于一个单导向RNA(sgRNA)和Cas9酶进行基因组编辑。研究人员可以用简单、快速和经济的方法来产生sgRNAs,因此能够在培养细胞、小鼠、斑马鱼和其他模型系统中进行靶向诱变。为了靶向效率,预先筛选sgRNAs,对于成功诱变和减少动物饲养成本,都是可取的。八月七日在国际学术期刊《Nucleic Acids Research》发表的一项研究中,来自美国国立卫生研究院的研究人员,提出了一种简单、快速和具有成本效益的荧光PCR为基础的方法——CRISPR Somatic Tissue Activity Test (CRISPR-STAT),来确定sgRNA的靶向特异性效率。
最近,利用锌指核酸酶(ZFNs)、TAL效应物核酸酶(TALENs)和CRISPRs进行基因组编辑所取得的进展,已经使人们有可能在许多系统(包括斑马鱼)中进行靶向诱变。而靶向特异性的ZFNs和TALENs组装,由于设计、成本的限制和/或繁琐的程序,具有很多局限性,CRISPR /Cas9需要设计一个引导RNA(sgRNA),可以很低的成本快速地合成。延伸阅读:检测CRISPR/Cas9脱靶效应的简单方法。
此外,CRISPR/Cas9的靶序列是灵活的,仅仅受到一个protospacer相邻基序(PAM位点)的需要的限制。因此,快速设计和生成多个靶标的sgRNAs是简单的。然而,所有的sgRNAs在产生靶位点突变的时候,并没有表现出类似的活性水平。通过计算方法预测活性,对于所有应用程序可能是不可靠的,因为标准是来自于有限的、背景特定的数据。因此,对任何给定sgRNA活性水平进行快速的实验验证,是必不可少的。
目前,已经开发了许多方法来评估靶向核酸酶(包括CRISPR/Cas9)的活性。确定体细胞活性水平的“金标准”方法,是通过对靶区域进行 PCR,然后对大量克隆进行测序。虽然这种方法在确定活性水平方面,具有很高的灵敏度和特异性,但是它是昂贵的,并且是劳动密集型的。简单的错配试验,如高分辨率熔解分析(HRMA)和DNA裂解(通过T7核酸内切酶),在某些生物(如斑马鱼)中具有很差的特异性,基因组中具有高频的多态性,可造成假阳性的结果。
同样,限制性片段长度多态性分析受到靶位点选择的限制。Yu等人开发的基于定量PCR反应的程序,是检测体细胞活性的一种可行方法。然而,我们可能很难设计与预测切割位点重叠的稳定引物。最近两种以序列为基础的方法——称为TIDE和CRISPR-GA,被用于细胞培养实验中sgRNA活性的定量评价。然而,这两种方法都有其应用的局限性。目前还没有任何一种检测,可以很容易地用于所有模型系统。
在这项研究中,研究人员证明了CRISPR-STAT在斑马鱼中用作预筛选方法来评估sgRNA特异性的效用。通过将CRISPR-STAT评估与产生sgRNA的的无克隆方法相结合,任何实验室应该都可以在感兴趣的基因中产生突变,并在饲养动物之前确证打靶的有效性。研究人员建议,每个基因应该设计至少两个sgRNA,并评估一小部分的注射胚胎。这种方法有利于成功的诱变,并最大限度地减少了动物饲养和首建动物筛选的时间和成本。
作为原理验证,研究人员在斑马鱼中检测了这种方法,他们用具有已知和不同种系传递效率的28个sgRNA,分析了它们在注射胚胎中的体细胞活性。这些数据显示,在注射胚胎的荧光PCR分析结果和种系传递效率之间,存在着强烈的正相关性。此外,这种方法非常敏感,足以评估多个基因打靶。虽然,该研究是利用CRISPR/Cas9和斑马鱼测试了这种方法,但是它也可以应用于其他动物模型系统,以及其他基因组靶向核酸酶。