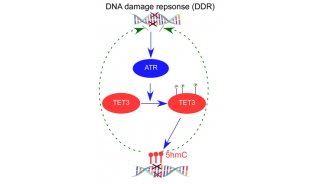
裴端卿组综述DNA去甲基化的级联调控模型
中国科学院广州生物医药与健康研究院裴端卿课题组综合近期体细胞重编程过程中TET相关DNA去甲基化的研究进展【1】和之前的相关研究,对细胞命运变化过程中的DNA甲基化模式重排过程和级联调控模型进行了调研,提出偶联TET进行DNA去甲基化可能是转录因子打开染色质的一种基本模式,该综述以The Battle between TET Proteins and DNA Methylation for the Right Cell为题于2018年11月3号发表在Cell 杂志系列子刊 Trends in Cell Biology。

DNA甲基化通常指胞嘧啶5位的甲基化,在基因转录调控和细胞命运决定中具有重要的作用。自催化DNA甲基化氧化并最终介导DNA主动去甲基化的TET家族蛋白于2009年发现以来【2-5】,TET家族及DNA主动去甲基化过程在细胞命运决定中的功能被逐渐地揭开。通过TET家族成员(Tet1/2/3)的全敲除,发现TET家族对于胚胎发育原肠作用和体细胞重编程都是必须的,在原肠作用中,TET通过激活Lefty反向调控Nodal通路(TET家族蛋白在胚胎发育中的重要作用)【6】,在体细胞重编程中,TET通过介导miR-200激活miR-200进而启动MET过程【7】。然而,DNA去甲基化过程在重编程中是否存在一个整体调控的模式?Tomas Graf团队运用其基于B细胞的快速重编程系统,显示Tet2在其系统中被早期激活并对重编程是必须的【1】。
Tet2可被C/EBPα 招募到Klf4等关键基因的调控区【1, 8】,进行去甲基化修饰而激活重编程关键因子;在重编程早期,Klf4亦可与Tet2形成复合物靶向多能性基因的调控区,促进去甲基化并使局部染色质变得更松散,从而激活早期多能性网络;而在重编程后期,多能性关键因子Tfcp2l1也招募Tet2到关键多能性基因的调控区进一步激活多能性网络,完成细胞命运转变。之前王建龙团队也发现转录因子Nanog和Zfp281通过招募Tet调控体细胞重编程和干细胞干性转化【9, 10】。这些结果提示转录因子偶联TET蛋白可以对染色质上的DNA甲基化分布进行重塑,进而影响细胞命运调控。
转录因子介导的TET蛋白级联调控去甲基化模型对理解发育过程中细胞命运调控的机制具有非常重要的作用,这一模型对由TET蛋白引起的相关疾病的治疗也有一定的借鉴意义。
据悉,中国科学院广州生物医药与健康研究院李东伟博士为第一作者,裴端卿研究员为通讯作者。

转录因子介导的TET蛋白级联调控去甲基化模型
参考文献
1. Sardina JL, Collombet S, Tian TV, et al. Transcription Factors Drive Tet2-Mediated Enhancer Demethylation to Reprogram Cell Fate. Cell Stem Cell, 2018:
2. Tahiliani M, Koh KP, Shen Y, et al. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science, 2009, 324(5929): 930-5
3. He YF, Li BZ, Li Z, et al. Tet-mediated formation of 5-carboxylcytosine and its excision by TDG in mammalian DNA. Science, 2011, 333(6047): 1303-7
4. Ito S, Shen L, Dai Q, et al. Tet proteins can convert 5-methylcytosine to 5-formylcytosine and 5-carboxylcytosine. Science, 2011, 333(6047): 1300-3
5. Ito S, D'Alessio AC, Taranova OV, et al. Role of Tet proteins in 5mC to 5hmC conversion, ES-cell self-renewal and inner cell mass specification. Nature, 2010, 466(7310): 1129-33
6. Dai HQ, Wang BA, Yang L, et al. TET-mediated DNA demethylation controls gastrulation by regulating Lefty-Nodal signalling. Nature, 2016, 538(7626): 528-532
7. Hu X, Zhang L, Mao SQ, et al. Tet and TDG mediate DNA demethylation essential for mesenchymal-to-epithelial transition in somatic cell reprogramming. Cell Stem Cell, 2014, 14(4): 512-22
8. Di Stefano B, Sardina JL, van Oevelen C, et al. C/EBPalpha poises B cells for rapid reprogramming into induced pluripotent stem cells. Nature, 2014, 506(7487): 235-9
9. Costa Y, Ding J, Theunissen TW, et al. NANOG-dependent function of TET1 and TET2 in establishment of pluripotency. Nature, 2013, 495(7441): 370-4
10. Fidalgo M, Huang X, Guallar D, et al. Zfp281 Coordinates Opposing Functions of Tet1 and Tet2 in Pluripotent States. Cell Stem Cell, 2016, 19(3): 355-69
-
科技前沿

-
科技前沿
-
项目成果
-
项目成果
-
项目成果

-
焦点事件
-
项目成果