数字PCR和基因组拷贝数变异研究
概述
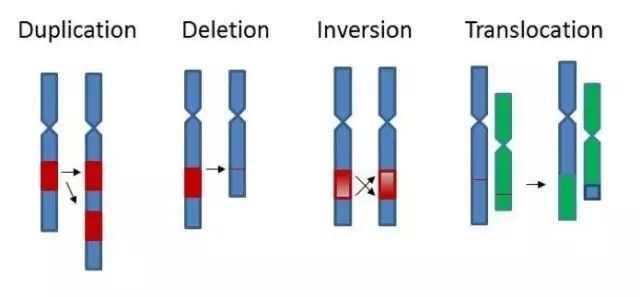
什么是拷贝数变异?为什么要对拷贝数变异进行研究?复旦大学杜仁骞、金力和张锋三位老师发表于2011年8月《遗传》的综述,给出了简明扼要的说明与回答,特引述如下:
“拷贝数变异(Copy number variation, CNV)是由基因组发生重排而导至的,一般指长度为1 Kb以上的基因组大片段的拷贝数增加或者减少,主要表现为亚显微水平的缺失和重复。CNV是基因组结构变异(Structural variation, SV)的重要组成部分。CNV位点的突变率远高于SNP(Single nucleotide polymorphism),是人类疾病的重要致病因素之一。
目前,用来进行全基因组范围的CNV 研究的方法有:基于芯片的比较基因组杂交技术
(array-based comparative genomic hybridization, aCGH)、SNP分型芯片技术和新一代测序技术。CNV的形成机制有多种,并可分为DNA重组和DNA错误复制两大类。CNV可以导至呈孟德尔遗传的单基因病与罕见疾病,同时与复杂疾病也相关。其致病的可能机制有基因剂量效应、基因断裂、基因融合和位置效应等。对CNV的深入研究,可以使我们对人类基因组的构成、个体间的遗传差异、以及相关人类疾病的发病机制有新的认识,并指导疾病的分子诊断和新的治疗手段的开发。”
作为第四代遗传标记,对于CNV的研究一方面集中在分子水平的遗传学研究,NGS是热门技术之一,因为把目标区域的序列一个碱基一个碱基测出来是最直接的方式,也就是中国人所谓的“眼见为实”,这个区域甚至可以是整个基因组。
NGS拿到的原始数据是许许多多长度较短的reads,再通过数据处理,将这些reads逐渐拼接成contig、scaffold直至染色体。这个拼接的过程是门大学问,不同的拼接就产生不同的contig。这就是生物信息学分析,核心依据是各种算法。算法越好,最终的contig越接近真实、客观的序列。话说回来,再好的数学模型也不可能放诸四海而皆准,也可能出错。
假定NGS得到的每个reads都正确无误,那么如何降低拼接过程中的错误?一方面可以在数学模型上下功夫,另一方面必然需要对结果进行验证。在对NGS和芯片CNV结果验证方面,dPCR由于其绝对定量的方式可以说是不二之选。
从2011年年底开始商业化数字PCR平台开始规模上市后,哈佛医学院遗传学助理教授Steven A McCarroll领导的实验室最早采用ddPCR对NGS结果进行验证,文章发表于2012年7月的Nature Genetics。目前Steven A McCarroll所在的实验室已将ddPCR作为常规技术整合入遗传学研究的整体流程,共有多篇高水平的文章发表,本公众号“数字PCR的可能性”后续将进行专门介绍。
对于NGS的生物信息学分析,数字PCR也是很好的验证工具。例如发表于2014年4月Nucleic Acids Research的论文,采用外显子组测序,并结合一种称为CANOES的统计学方法,寻找基因组上稀有的拷贝数变异。对于28个经过de novo测序得到的CNV位点,经数字PCR的核对发现:在NGS分析得到的28个CNV位点中,19个位点得到了确认。随着数字PCR应用的推广,相信这类文章会越来越多。
CNV是基因组结构变异的重要形式,已经明确和肿瘤发生、自身免疫病、神经系统疾病息息相关,那么CNV分析也应该成为分子检测的一部分。包括分子诊断在内的诊断方法,评价方法好坏的标准可以简单地总结为四个字:多、快、好、省。一种方法越符合“多、快、好、省”的标准,就越理想,越容易推广、开展。所谓“多”,是指检测样品的通量越高越好;所谓“快”,是单个样品的检测时间越短越好,对于这一点每个在医院等待检验报告的人都深有体会;所谓“好”,是结果可靠并易于判断,而且这种判断越傻瓜越好,在临床检验这件事情上来不得模棱两可、见仁见智;所谓“省”,是指单个样品的检测成本。
如果我们以经典的HER2 CNV检测为例,按照上面“多、快、好、省”的原则来检验目前作为HER2检测金标准的FISH,其实FISH算不上理想的检测手段。那么数字PCR在CNV临床检测方面是否有机会呢?目前已有很多国内外实验室对包括HER2在内的疾病相关基因CNV展开了研究,这些研究结果让我们看到了dPCR技术应用于临床诊断的可能性。
-
综述
-
焦点事件