Nucleic Acids Res:DNA同源重组中Shu复合物的重要分子机制
来自中国科学院生物化学与细胞生物学研究所国家蛋白质科学中心(上海)丁建平研究组在国际学术期刊《核酸研究》(Nucleic Acids Research)在线发表了题为“Structural basis for the functional role of the Shu complex in homologous recombination”的论文,该研究揭示了Shu复合物在DNA同源重组过程中发挥生物学功能的分子机制。
当作为遗传信息载体的DNA受到损伤后,会造成其结构和遗传信息的改变,继而影响到DNA的复制和转录等过程,导致细胞周期停滞和基因表达失调,最终可能引发细胞的癌变或死亡。对于数量庞大、类型多样的DNA损伤,细胞进化出一系列DNA损伤修复机制,用于保证基因组的完整性和稳定性。
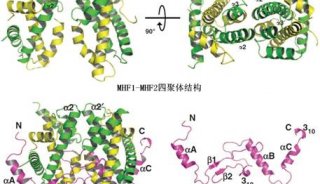
丁建平研究组近年来致力于DNA损伤修复通路相关蛋白质的结构与功能研究,先前已报道了范可尼贫血信号通路中MHF1-MHF2复合物(Structure,2012)和FANCM-FAAP24复合物(Nucleic Acids Research,2013)的晶体结构,并阐述了这些复合物与DNA结合并发挥其生物学功能的分子机制。
Shu复合物是近年研究发现的一类保守的、广泛存在于真核生物,参与调控DNA同源重组过程的多元复合物。在酿酒酵母中,Shu复合物是由Csm2、Psy3、Shu1和Shu2四个蛋白组成,通过参与同源重组途径而在DNA的损伤修复和耐受过程中扮演了重要的角色。迄今为止,Shu1和Shu2蛋白的晶体结构及全复合物的结构信息仍然不清楚,这影响了对Shu复合物在DNA损伤修复途径中发挥生物学功能的理解。
丁建平组博士生张世成等人成功解析了酿酒酵母源Shu复合物的晶体结构。整体结构显示,全长的四个组分蛋白Csm2、Psy3、Shu1和Shu2依次顺序相互作用形成一个V型结构,其两个亚复合物Csm2-Psy3和Shu1-Shu2分别位于V型的两侧。
结构分析发现,Shu1采用了ATPase核心结构域的经典折叠方式,是一个新的Rad51旁系同源蛋白。Shu2是第一个结构获得解析的Shu2/SWS1家族蛋白,采用了一种全新的蛋白折叠方式。Shu2蛋白除了包含家族保守的SWIM结构域外,还包含一个插入结构域,并且都参与了与Shu1的相互作用。Shu1和Shu2通过大量疏水相互作用和少量亲水相互作用形成一个稳定的亚复合物。在Shu复合物中,Psy3的N端伸出的大约20个氨基酸相比于Csm2-Psy3二元复合物结构中的Psy3发生了大约60度的偏转,并与Shu1发生了紧密的相互作用。此外,还发现Shu1上的Val51在全复合物组装过程中也扮演着重要的角色。
在结构分析的基础上,研究人员通过酵母双杂和pull-down等实验验证了相互作用界面上关键氨基酸对于复合物形成的作用,并通过酵母MMS敏感性实验发现,Shu复合物的完整性对于其在DNA损伤修复途径中发挥功能是必需的。进一步通过对复合物表面电荷分布的分析,并结合已有的生化实验数据,得到了复合物结合DNA的两个可能区域。根据两个区域的结构和化学特征,进一步推测区域I主要结合单链DNA,而区域II则主要结合双链DNA。
基于研究结果,研究人员提出了Shu复合物结合3'-overhang或fork等同源重组过程中不同形式结构DNA、以及在DNA损伤修复途径中发挥生物学功能的分子机制。
-
项目成果
-
焦点事件

-
焦点事件