基因编辑进展梳理 Part II 基于CRISPR-Cas9的技术应用篇(一)
前言:近年来,CRISPR基因编辑技术正在席卷整个生物医学研究领域,上一期我们已先从CRISPR系统开发及机制研究方面梳理了2018年相关大事件。伴随着基础技术不断优化,CRISPR技术的应用也更加广泛,如动物造模、药物筛选、单碱基编辑技术、细胞谱系示踪、基础疾病研究、疾病诊断、体内编辑和遗传病校正等方面。值得一提的是,全球首例基于CRISPR的基因编辑临床试验已于2018年开始实施[1],同年12月,FDA又批准Editas的一项利用CRISPR技术治疗先天性黑朦病的临床试验申请,有望成为世界上第一种在人体内使用CRISPR技术的疗法。本期小编就先从动物造模及单碱基技术等方面带大家浏览一下近一年里CRISPR技术应用的重大突破。
CRISPR技术的五大应用领域
一、大动物造模
CRISPR-Cas9系统以其简易的操作为动物模型的构建提供了高效的方法。目前基于CRISPR-Cas9构建的各种人类疾病的动物模型数以千计,主要以啮齿类动物(如小鼠)模型为主,其相关的发病机制和治疗进展为临床应用提供了许多有价值的参考。邦耀实验室也一直致力于相关研究,在国际知名期刊上发表多篇关于基因编辑大小鼠构建及其在多种疾病上应用的文章,并为广大的科研用户提供了数百种基因编辑动物模型。
尽管小鼠模型可以很好地展示疾病的表征,但由于寿命和生理构造等方面的巨大差异,对于许多神经退行性疾病以及同年龄相关等的疾病,并不能很好地模拟许多人类疾病的相应表征。因此,在遗传学、解剖学和生理学上与人类更接近的大型哺乳动物模型的构建显得尤为重要。由于胚胎干细胞技术较难、繁殖周期太长和基因编辑效率低等障碍,大型哺乳动物模型的构建一直都是科技攻关的难点。
然而,随着CRISPR-Cas9技术的快速发展,现有的基因编辑工具已被证明能够成功构建各类大型哺乳动物模型。2018年中国科学家在基因编辑猪和猴上做出了重要贡献,在基于CRISPR-Cas9技术构建基因编辑大型哺乳动物模型方面引领了世界的脚步。
“基因敲入”猪:亨廷顿舞蹈病猪模型的构建
2018年3月,来自中国的多个团队合作建立了一个“基因敲入”的亨廷顿舞蹈症(HD)猪模型,利用CRISPR-Cas9技术将导致HD的mHTT基因片段整合入猪成纤维细胞中,再通过体细胞核移植技术获得mHTT转基因猪模型,这一研究为测试HD疗法提供了一种重要的动物模型[2]。
2. 六篇文章奠定了我国科学家在构建基因编辑猴模型中的世界领先地位
l 2018年年初,中科院神经科学研究所的研究人员首次报道了,通过在胚胎细胞分化为特殊细胞时去除DNA上的化学修饰的方式,成功克服了非人灵长类动物的体细胞克隆的限制,首次实现了基因克隆猴的诞生(见图1a),标志着非灵长类动物体细胞克隆技术的里程碑[3]。
l 一年后,该课题组又相继发表两篇文章,通过CRISPR-Cas9技术构建了睡眠紊乱与精神相关异常的BMAL1敲除猴[4],随后将BMAL1敲除猴的皮肤成纤维细胞作为细胞核供体通过体细胞核移植的方法获得了遗传背景一致的疾病敲除猴模型(见图1b)[5],为非灵长类动物疾病模型的构建及在临床研究中的应用奠定了坚实的基础。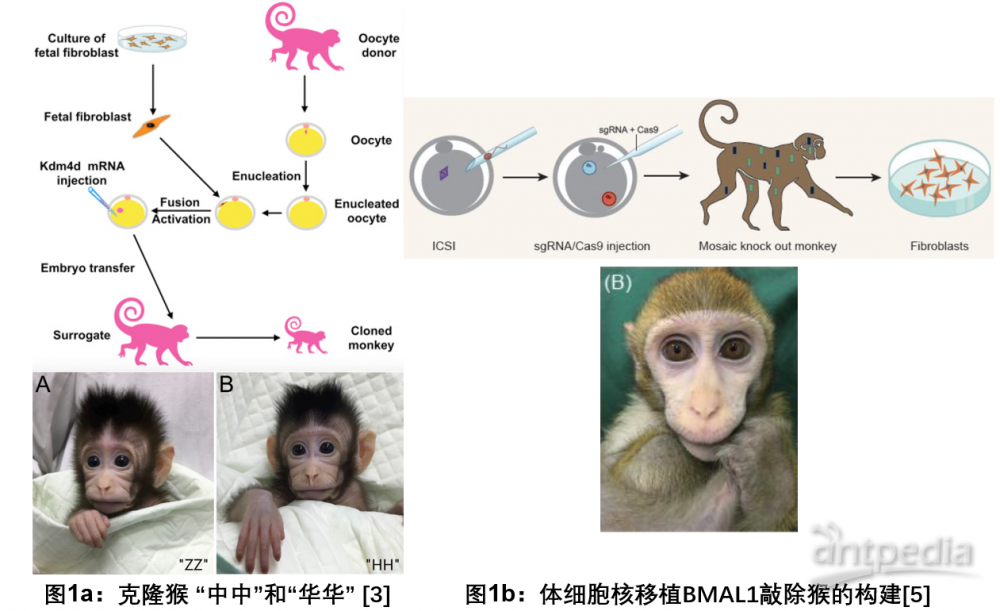
l 2018年1月12日,Cell
Research杂志同时发表了两篇来自中国科学家的研究工作,报道了世界首例基因敲入食蟹猴的诞生。其中来自中科院神经科学研究所的杨辉研究组利用之前开发的一种同源末端介导整合(HMEJ)的方法,以胚胎原核注射方式通过优化注射条件首次成功获得了ACTb-T2A-mcherry基因定点敲入食蟹猴(见图2),并证实这些猴子具有生殖遗传的可能。这一研究为通过提高整合效率来构建基因敲入猴提供了新的解决思路[6]。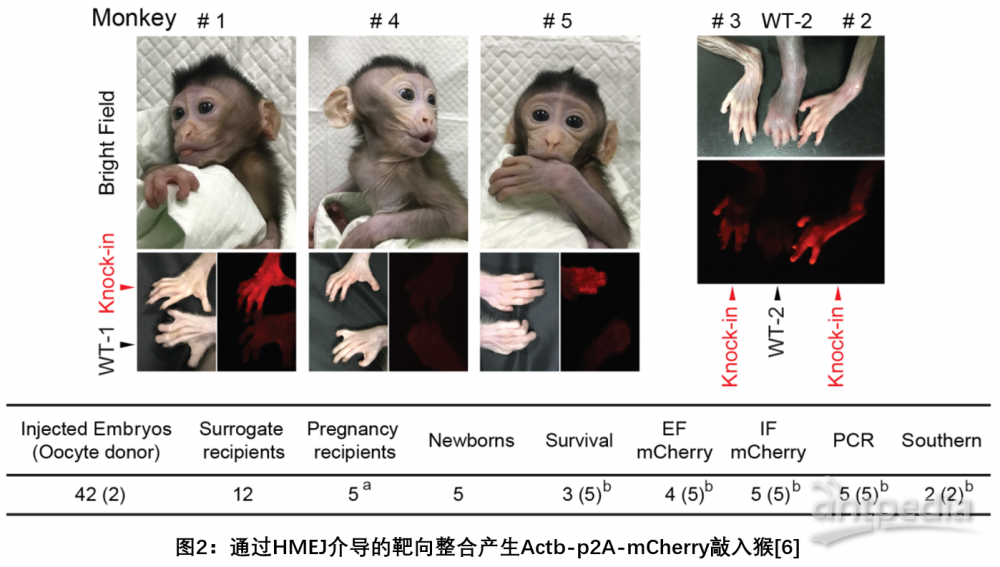
另一项研究则由早年利用CRISPR技术成功构建基因敲除食蟹猴的季维智院士团队合作完成,他们基于之前利用CRISPR-Cas9在大鼠上进行精确基因编辑的技术进一步拓展到食蟹猴中,成功获得了Oct4最后一个密码子后定点整合GFP的食蟹猴,并检测到少许的错配以及脱靶现象。这项研究证明了灵长类动物可成功进行精确基因编辑的可行性,为灵长类动物的重编程研究提供了一种通用的工具[7]。
l 2018年8月23日,中国科学院干细胞与再生创新研究院的研究团队在Nature发文,首次实现了在非人灵长类动物中全身敲除SIRT6,获得了世界上首例特定长寿基因敲除的食蟹猴模型(见图3),确定了SIRT6在非灵长类动物中的生物学功能。研究的结果表明,SIRT6参与调节非人灵长类动物的发育,SIRT6的缺乏会导致胎儿在子宫内发育延迟。这一模型的建立可能为模拟和研究人围产期致死综合征的发病机制开辟了新的途径[8]。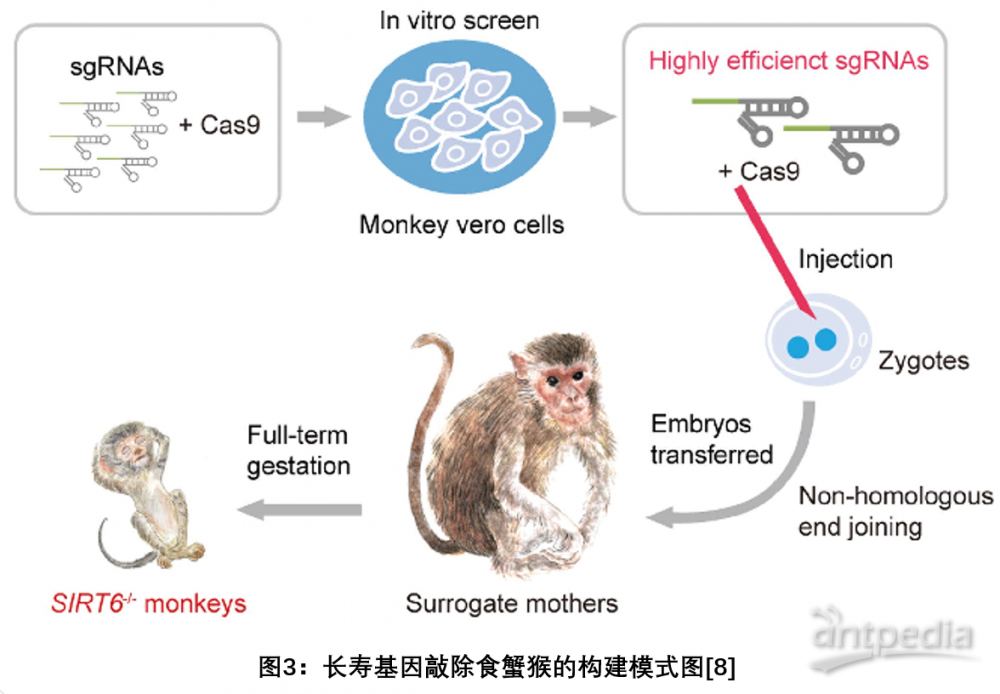
二、单碱基基因编辑技术的优化
单碱基基因编辑技术是指能在基因组上引起单个碱基改变的基因编辑技术。基本原理是将胞嘧啶脱氨酶(APOBEC)或腺苷脱氨酶与现存Cas9n(D10A)融合而形成,依赖于CRISPR原理使得靶点远离PAM端的4-7位的单个碱基发生修改的基因编辑技术。单碱基编辑系统以其不用切割基因组就能实现碱基的置换而被作为目前最为安全有效的基因编辑工具,但其编辑范围、编辑窗口、编辑效率以及特异性等未完善的问题仍然需要进一步地改造和优化。
xCas9: Cas9快速进化系统扩大碱基编辑工具靶向范围
2018年2月,David Liu 实验室在Nature发文,使用噬菌体辅助的连续进化系统进化Cas9从而获得了一种能识别多种PAM的SpCas9变体( xCas9 ),它可以识别包括NG、GAA和GAT在内的多种PAM序列,可以靶向人类基因组范围内约1/4的靶点。xCas9支持在人类细胞中的多种应用,包括靶向转录激活、核酸酶介导的基因敲除以及胞苷和腺嘌呤碱基编辑。相比spCas9,xCas9具有更高的DNA特异性,且当用非NGG PAMs靶向基因组位点时,xCas9也表现出极低的非靶向活性。这些发现扩大了CRISPR系统的DNA靶向范围,与ABEs 融合,也同样使ABEs PAM的选择更加灵活[9]。
2. dCpf1-BEs:一种能够识别富含T的PAM的碱基编辑器
2018年5月,上海科技大学陈佳实验室在Nature
Biotechnology发文,通过将大鼠胞嘧啶脱氨酶APOBEC1融合到催化失活的Cpf1中,开发了一种基于CRISPR-Cpf1的dCpf1-BEs,从而克服了目前BEs仅能靶向富含G/C的PAM序列的限制,使之能够识别富含T的PAM序列,并催化人体细胞中的C
- T转换,同时产生较低的编辑副产物,具有更高的编辑精度。这些发现为碱基编辑系统的全面深入应用提供了新思路、拓展了应用范畴[10]。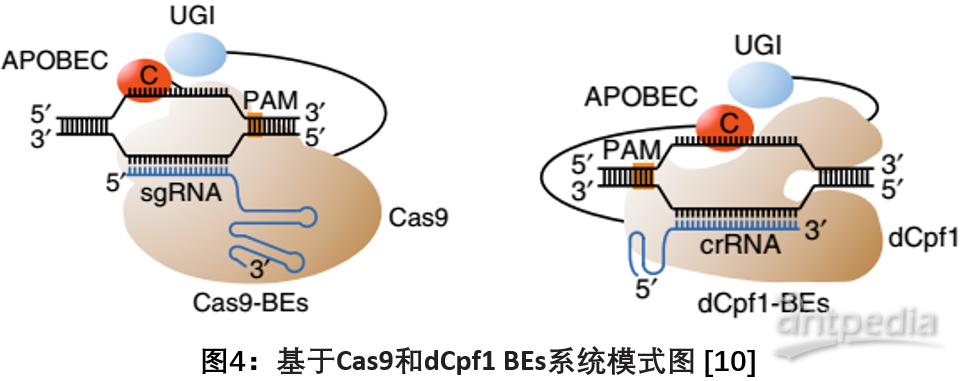
3. BE4max 和ABEmax:优化碱基编辑器解决表达水平的技术瓶颈
2018年5月,David Liu实验室在Nature biotechnology继续发文,认为表达水平是碱基编辑器的技术瓶颈,通过引入核定位信号( NLS )和密码子优化,以及脱氨酶组分的原始重建,优化胞苷( BE4 )和腺嘌呤( ABE7.10 )碱基编辑器,获得了编辑效率更高的BE4max 和ABEmax,分别提高1.9倍和1.3-7.9倍。优化的碱基编辑器可校正致病性SNPs,大大提高了在各种哺乳动物细胞中的编辑效率[11]。
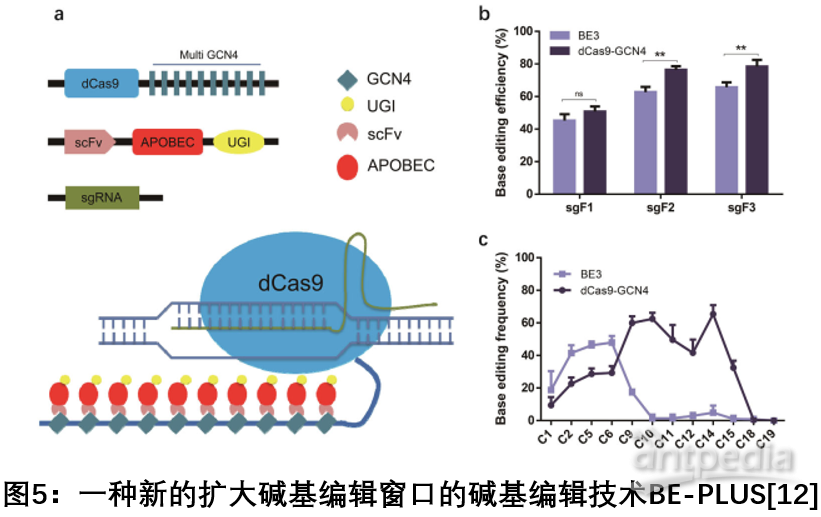
4. BE-PLUS:新型碱基编辑工具具有更宽的编辑窗口和更高的保真度
2018年 7月,陆军军医大学陈洁平课题组和上海科技大学黄行许课题组合作在Cell
Research上发文,开发了一种新的编辑技术BE-PLUS,将10个拷贝的GCN4肽融合到nCas9 ( D10A )中,向靶位点招募scFv
- APOBEC - UGI - GB1,从而更大范围地进行C to U ( T
)的编辑,极大地拓展了碱基编辑器的编辑窗口。新系统通过扩大窗口实现了碱基编辑,从而扩大了基因组的靶向范围,并且还能提高保真度[12]。