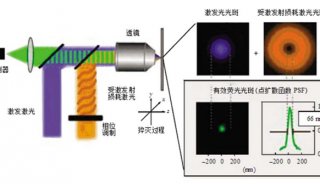
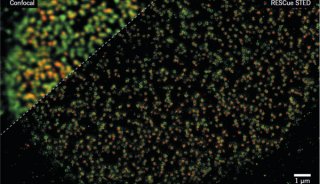
2016年《科学》综述:超分辨率显微技术
从列文虎克到21世纪,显微镜由一个看似牢不可破的原则所控制:分辨两个对象的能力受限于观察它们的光波波长。 但在2000年,研究人员显示出, 这种所谓的衍射极限可以被打破, 在接下来的十年中揭示了从 GSDIM和 PALM到 SIM、STED 和 STORM 的一系列像“字母汤”一样的超分辨率技术 。
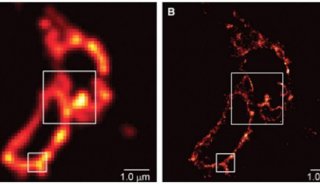
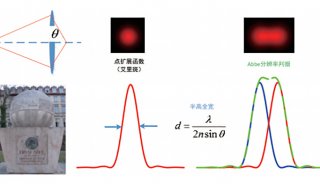
不,这并不是审稿编辑出了问题,这句话末尾真的有两个句号。。你的大脑抓住了这个明显的错误,因为你眼睛晶状体所具备的分辨能力足以区分两个紧挨着的物体。当然,显微镜可以解析更细微的细节,但这种能力不是无限的。所谓的阿贝(或衍射)极限限制着显微镜的能力,而且在某种情况下,两个物体会显示为一个。
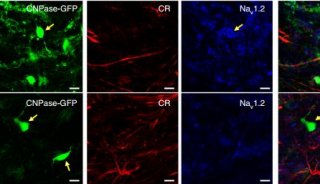
2006年,哈佛大学庄小威团队首先阐述了被称为随机光学重建显微镜(STORM)的超分辨率技术,这是一种单分子定位方法,其20纳米的横向(xy)分辨率超过衍射极限达整整一个数量级。

在接下来的十年中,庄小威的团队稳步改进技术,做到了多色、更高分辨率和三维化。
“几乎所有生物结构都是三维的。”霍华德·休斯医学研究所研究员、哈佛David B. Arnold Jr.科学教授庄小威说, “因此如果你只在xy(平面)上打破你的衍射极限,而不是z,你就会受到限制。”
为了克服这个限制,庄小威的团队在显微镜的光路上插入了柱面透镜。在该装置中,从单个分子产生的光不再是圆的而是椭圆的,表明分子的轴向位置具有50纳米的分辨率。随后,实验室通过将物镜定位在样品上方和下方来进一步提高分辨率,使柱面透镜组合时所产生轴向位置的分辨率优于20纳米。
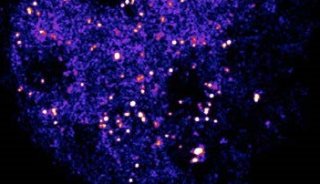
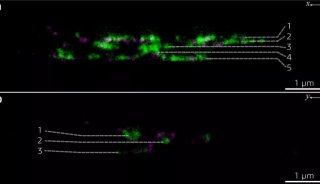
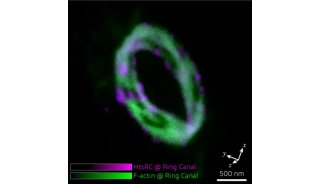
当她的团队应用这种技术固定大鼠海马神经元时,他们得到了完全出乎意料的发现,庄小威说。在常规荧光显微镜下,肌动蛋白在这些神经元的轴突中看起来像一致而无特色的绿色。但是当他们使用STORM放大时,就在轴突轴的质膜下面,他们发现了一个实实在在的细胞骨架:绿色肌动蛋白的周期环隔着有180 纳米,被洋红色血影蛋白间隔着。
“这是一个美丽的结构。”庄小威说,同时提供了机械化的灵活性和组织化的架构。有了它,细胞可以精确定位关键的膜蛋白,如离子通道,以确保神经冲动的有效传送。“有了这些非常重要的信号分子固定在结构上,为你提供了解释这种情况的可能性。”她补充说,“这是我最自豪的那些STORM图像之一。
庄小威发表于2013年的此项研究是“一个重大的贡献”,耶鲁大学医学院细胞生物学和生物医学工程副教授Joerg Bewersdorf表示认同,他在其实验室中构建了超分辨率显微镜(包括现在已由Bruker商业化的显微镜)。 “从未有人看到过这些肌动蛋白环,即使人们为了了解轴突中的肌动蛋白组织,已经用电子显微镜看了20年。”
诠释超分辨率
德国马普生物物理化学研究所Stefan Hell教授在2000年首次打破了衍射屏障,他认为,所有超分辨率方法都是基于相同的基本原理。
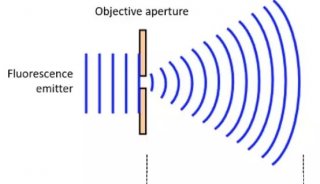
“如果你有自由传播的光,这束光将总会被衍射。你不可能将它更精确地聚焦在小于光波波长约一半的区域。”Hell这样解释。结果就是,距离小于200纳米的对象将被同时照射并一起发出荧光。长话短说:多个对象一起变成了一个无法区分的模糊影像。“这个问题的解决方案是要确保,并不是所有充满激发光的对象最后都能发射荧光。”
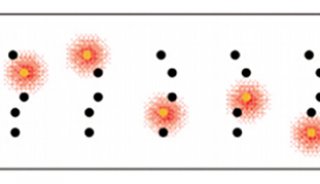
STORM、光激活定位显微技术(PALM)和具备单独分子返回的基态损耗显微技术(GSDIM)分别得到了Nikon,Zeiss和Leica公司的商业化。它们是基于定位的随机方法,使用可光活化或可光开关的荧光蛋白或染料随机打开几个荧光团,剩余的大多数保持黑暗。在这些条件下,研究人员可以相对确定每个荧光点仅响应于单个活性分子,因此可以以纳米精度分配它们的位置。然后这些荧光团关闭,再打开另外几个,并且循环重复,直到完成完整的数据集收集。
“基本上我们是利用了我通常所说的‘第四维’。”庄小威解释说。如果有多个的话,原本难以区分的对象会在空间中紧密定位,她说,“我们不允许它们在时间上有所重叠。
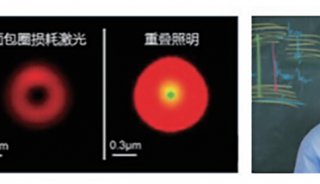
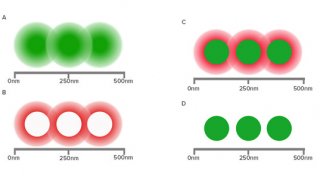
相反,受激发射损耗(STED)、可逆可饱和光学荧光跃迁(RESOLFT)和结构化照明显微技术(SIM)使用“光的模式决定了分子在何处是有活性的,何处是关闭的”,Hell说。Hell开发了STED和RESOLFT方法,并共同创立了一家名为Abberior的公司,为这些及其它超分辨率方法提供商业化的荧光团。例如,STED通常被用于实现点扫描方法,使用叠加在激发束上的“环形”光,通过一个被称为“受激发射”的过程迫使在照明体积周边的荧光团保持黑暗, “从而将发射区域收缩到圆环的中心。(RESOLFT使用可光开关的荧光团以达到相同的效果。)
根据Leica Microsystems的STED和GSDIM显微镜产品经理Jochen Sieber所说,与STED或RESOLFT相比,定位系统通常能提供更清晰的分辨率,但所需的时间要长的多,因为它们需要采集和合并成千上万帧影像以构建单个图像。(的确,美国国立卫生研究院杰出研究员Jennifer Lippincott-Schwartz注意到,这第一批图像的收集需要几个小时;而如今,典型的帧速率大约是5分钟。Lippincott-Schwartz的实验室发明了PALM。)
由Nikon、GE生命科学和Zeiss公司所商业化的SIM通常被认为是市面现有的最快也是最简单的方法,因为它不需要特殊的样品制备。但若到达100纳米,它所产生的分辨率也最低。
最近,Janelia研究园研究员Eric Betzig因其在超分辨率上的贡献,与斯坦福大学的Hell和William E. Moerner共同获得了2014年的诺贝尔化学奖,证明了即使这个限制也非不可逾越的。2015年末,Betzig和他的团队找到了两种策略,包括“具有模式化激活的非线性SIM”,从而将该方法的分辨率提高到约45纳米。“我个人的感觉是,SIM的超分辨率方法很可能在未来十年能够回答最多的生物学问题。”Betzig笑着说,“但这并不在诺贝尔奖之中。”
英国曼彻斯特大学医学研究委员会(MRC)职业发展研究员Raman Das在2014年解决了类似的问题,当时他使用SIM(以及其它方法)发现了一种细胞分裂的新形式,被称为“顶端脱落”。Das感兴趣于胚胎脊髓中的祖先细胞如何分化成神经元并离开组织。通过使用延时衍射限制荧光显微镜和SIM,Das的团队发现这些细胞实际上修剪掉了细胞的“尖端”,一下子就去除了这些蛋白质——Das将它比作合上带拉绳的手提袋的过程。 “这导致这些细胞的细胞极性快速丢失。”他解释说,从而引起功能性神经元结构的形成。
也许最简单、当然也是最便宜的超分辨率选择是扩展显微镜。该技术由麻省理工学院的Ed Boyden开发,通过将样品包裹在“可膨胀聚合物”凝胶中来提高成像分辨率,使得样品等轴地增长了约五倍。在功能上,这与提升分辨率(从200纳米降至40纳米)是一样的。“这有点像光学变焦。”德国哥廷根大学教授Silvio Rizzoli解释说,他在神经突触研究中采用了这种技术(以及STED和STORM)。
荧光团的开关
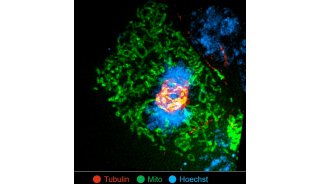
马普生物物理化学研究所线粒体结构和动力学研究小组负责人Stefan Jakobs使用超分辨率方法研究线粒体在细胞死亡或细胞凋亡中的作用。目前,已知一种被称为Bax的蛋白质在响应凋亡信号中诱导线粒体膜破裂。但不清楚的是该蛋白质是如何做到的。
使用固定细胞STED,Jakobs的团队展示出多个Bax分子组装成线粒体外膜中的环,如透明的绿色光环笼罩在红色的膜上。该结构解释了在凋亡期的Bax易位如何导致线粒体内容物溢出,Jakobs说。“看来这些环状物代表着毛孔。”他解释说。然而,他们不可能通过常规显微镜分辨出来,这些环在其中表现得“一团模糊”。
为了看到这些环,Jakobs用到了荧光染料标记的抗-Bax抗体。然而,适用于超分辨率显微镜的荧光探针有时难以获得。“你想要快速的图像采集和高分辨率;你想要有很多颜色和低光毒性。目前没有一种技术可以完全实现所有这些参数。”他说。
Jakobs已经开发了可逆可切换的荧光蛋白(RSFP)——属于绿色荧光蛋白(GFP)的变体,例如rsEGFP2——它可以被重复地从荧光状态推到非荧光状态,再使用特定波长的光推回。他指出,这种蛋白质适用于RESOLFT和STED在活细胞中的应用,因为它们不需要加入外部抗体进行标记。但是绝对不要过量表达这些蛋白质,他说,因为这可能引起人为干扰。因此,Jakobs的团队使用被称为CRISPR / Cas9的基因编辑系统将rsEGFP2与蛋白质编码基因偶联,从而确保了内源性表达水平。这项工作是“具有挑战性的”,他承认,“但我认为对这个领域来说这是非常重要的一项工作。”
另一种遗传标记的选择涉及到使用自标记蛋白标签来标记基因,包括SNAP标签和CLIP标签(可从New England BioLabs获得)或Promega的Halo Tag标签。研究人员可以通过加入连接到适当底物的透膜染料,特异性标记活细胞中感兴趣的蛋白质。当然,窍门在于,这种染料必须是明亮并具有细胞渗透性的,当前只有少数几个荧光团符合该标准,瑞士洛桑联邦理工学院化学科学和工程研究所教授Kai Johnsson说。
Janelia研究园的研究员Luke Lavis最近报道了一组具细胞渗透性和高量子效率的罗丹明变体,可与Halo Tag化学标签配合使用。Johnsson同样也开发了罗丹明变体并称其为“SiR染料”(羧化的硅—罗丹明),已经由Spirochrome公司开展商业化。
通过将这些染料与靶向配体(例如DNA荧光染料Hoechst或癌症药物紫杉醇)偶联,Johnsson创建了能够在活细胞STED和SIM(而不是PALM或STORM)中分别特异性点亮遗传物质和微管的探针。
鲜活的色彩
由于在定位超分辨率显微镜中编译图像需要更长的时间,该方法很可能不适用于活细胞。
一种解决方案是用高速科学互补金属氧化物半导体(sCMOS)相机,代替传统的电荷耦合原件(CCD),它可以每秒捕获成千上万帧。举例来讲,2013年,Bewersdorf及其同事展示了噪声处理算法,让sCMOS相机可以高达每秒32帧的速度记录超分辨率的图像。如今,Leica公司的GSDIM仪器包括一个sCMOS摄像头,可以将数据采集时间缩短到“一分钟以内”,产品经理Peter Laskey说,因此让其对活细胞的应用“触手可及”。Nikon公司新推出的N-STORM 4.0系统也声称有更亮的激光,可以“让闪烁的开关率非常之高”,总经理Stephen Ross说。
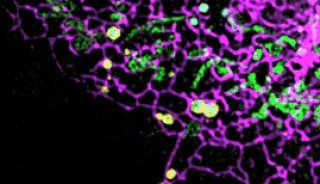
研究人员还设计了其它替代策略来规避速度的问题。例如,Melira Lakadamyali是西班牙巴塞罗那光子科学研究所的团队负责人,她研究微管囊泡运输,特别是如何使运输这些囊泡的动力蛋白可以“在活细胞拥挤的环境中逃避路障和交通拥堵”。
为了回答上述问题,Lakadamyali发挥了她的系统的优势。通过记录囊泡在高时间尺度(但低空间尺度)分辨率的活细胞影像,她的团队可以跟踪囊泡运动,将这些轨迹叠加在微管位置的STORM图像上。
研究人员发现,囊泡倾向于在微管相交处停顿;这种暂停“与这两个微管的间隔很小、紧密相交相关”,Lakadamyali说。但他们同时发现,马达最终可以导航通过相交点。该团队现在正在使用3D粒子跟踪来关注与微管相关的原丝,这可能会为穿过路障提供类似于路肩一样的帮助。
伦敦国王学院皇家学会大学研究员Susan Cox同样对活细胞定位显微技术感兴趣。Cox与Lippincott-Schwartz合作开发了一种算法,被称为“闪烁和漂白的贝叶斯分析(3B)”,允许研究人员同时成像更多的荧光团,其中一些实际上甚至是在空间上重叠的,从而将每个实验所需的图片数量减少了大约一个数量级。“我们已经看过像心肌细胞这样的系统,并且能够在细胞的‘节拍’之间进行成像。”Cox说。
然而,高分辨率所有能教会我们的是,显微技术是一种平衡。在报告中,Betzig说,他通常会展示一个四面体,其轴代表着不同的目标:分辨率、速度、低光毒性和成像深度。“你不能拥有所有的一切,”他告诉听众。对于Betzig来说,速度是至关重要的,他将自己的努力聚焦在从他的样品中挤出每一个光子。这就是他为什么开始从单纯的超分辨率逐渐走向超高速成像,有着被称为“格子光片显微技术”的新设计,很快就要在Zeiss和3i公司进行商业化。
当然,其他人可以从不同的角度解释四面体。但底线在于:当谈到显微镜,未来从没有如此光明——或如此清晰。
(译者之一高大海系中国科学院海洋研究所助理研究员)
Jeffrey M. Perkel 是爱达荷州波卡特洛市的科学自由撰稿人。
DOI: 10.1126/science.opms.p1600105
鸣谢:“原文由美国科学促进会(www.aaas.org)发布在2016 年5 月12 日《科学》杂志”。官方英文版请见http://www.sciencemag.org/custom-publishing/technology-features/superresolution-microscopy。
-
科技前沿
-
科技前沿