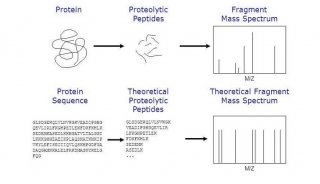
复杂基体中目标肽的测定
本文介绍了使用Thermo Scientific公司的TSQ Quantum Ultra质谱仪进行复杂生物机体中目标肽的方法,结果表明,基于串连质谱的选择反应监测(SRM)流程,该方法可以在具有高化合物背景的复杂基体中检测低浓度水平肽,同时保证良好的定量准确度和精密度。
目前一般生物标志物实验的终点是发现公认的标志性蛋白名单,接下来的一步就应该是在扩展患者群体内完成对这些靶向蛋白的定量测定,以评价它们作为标志物的有效性。对这些在复杂基体中的靶向蛋白进行可靠的定量分析,其分析准确度和精密度是必需的。对低丰度蛋白质进行高通量检测,对于宽动态范围、高灵敏度和稳健易用性都有严格的要求。基于串连质谱的选择反应监测(SRM)流程,对于复杂生物基体中所研究蛋白的特异肽可以进行高灵敏度和高选择性的定量,同时也可以独立进行生物标志物验证[1-3]。该方法的主要优点是具有很低的检测限,可以在具有高化合物背景的复杂基体中检测低浓度水平肽,同时保证良好的定量准确度和精密度。
本文验证了TSQ Quantum Ultra质谱仪在用于人血清酶解液中待测肽的定量分析时,显示出高选择性和高灵敏度的优势。研究建立了通过高分辨率的母离子分离和目标肽多碎片离子时间序列检测,同时进行目标肽的鉴定和定量的测定方法,对H-SRM方法的定量准确度也进行了评价。
实验条件样品制备
25μl人血清(Sigma Corp., St. Louis,MO)用975μl 浓度为100mM的碳酸氢铵缓冲液40倍稀释,超滤处理(截留分子量10 000 MW),酶消化。将消化液真空干燥,并在0.1%甲酸250μl(10倍稀释)水溶液里重组。
液相条件
纳流高效液相(Nano-HPLC)
Thermo Scientific SurveyorTM MS 泵和MicroAS自动进样器
色谱柱:75μm×100 mm C18
Post-split流速: 300nl/min
流动相: A为0.1% 甲酸的2%乙腈溶液;B为0.1% 甲酸的乙腈溶液
线性梯度: 90min,2%B升为50%B
进样量: 2μl,直接进样色谱柱
质谱条件
TSQ Quantum Ultra质谱仪
Thermo Scientific Ion MaxTM离子源,配有纳流柱接头(New Objective)
高选择性反应监测(H-SRM):Q1,0.2FWHM;Q3,0.7FWHM
选择反应监测(SRM):Q1,0.7 FWHM;Q3,0.7FWHM;Q2,1.5mTorr
定量增强数据关联扫描(QED-MS/MS):(1)选择反应监测扫描(SRM)为Q1和Q3,0.7FWHM;Q2,1.5mTorr;318种跃迁H-SRM实验中驻留时间为5ms,循环时间2.2s。(2)定量增强数据关联扫描(QED-MS/MS): 根据扫描1的母离子关联扫描;信号阀值 5000;Q1和Q3,0.7FWHM;Q2,1.5mTorr;CE, 0.034×母体质量m/z + 3.134;动态排除,重复2次;持续时间,60s;排除时间,60s;排除名单大小,50;循环时间3.84s。
数据库检索
为方便检测结果比较,所有的数据采用MascotTM数据软件(Matrix Sciences)处理,使用SwissProt数据库(分类:人类)。
肽选择和SRM离子对参数设置
有两种进行肽选择和SRM离子对参数设置的基本方法[3]。如果靶向蛋白曾经在以前的LC-MS/MS实验中被检测过,该蛋白产生的特异肽就被选出,用于随后的SRM实验。相反,如果没有LC-MS/MS数据可以用于靶向蛋白,肽选择和SRM实验设置则使用已知的蛋白序列(模拟方法)在电脑中完成。我们开发了一个用于靶向蛋白定量的SRM工作软件,它可以用于预测候选肽,并可以为SRM选择多碎片离子,从而建立仪器方法和序列文件,并可自动进行肽鉴定和定量数据处理(见图1)。本文所述所有SRM实验都使用了该软件。
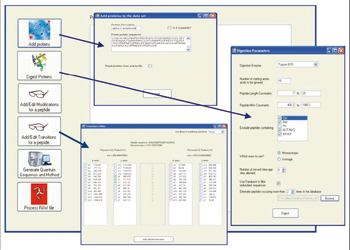
图1. SRM工作软件中肽选择和SRM离子对参数设置。
结果和讨论
H-SRM目标肽定量方法评价
本文所提出的H-SRM工作流程如图2所示。高分辨分离(0.2FWHM)的主要优势是它从复杂基体中选择性检测低丰度肽峰的能力3。通过监测在同一持续时间内选择的多种碎片离子,完成对肽的鉴定。该方法没有限定对每种肽可以选择的碎片离子的数目。一般而言,每种肽选择4~7种y-型离子足以保证实验的高度专一性。对高分辨母离子分离的增强选择性避免了费时的全扫描MS/MS。采用H-SRM (318组 H-SRM实验为2.2s,驻留时间设为5ms)缩短了工作循环时间,使得在一个LC流程内不仅可以同时进行肽的鉴定和定量,而且还最大程度保证了检测灵敏度。为了评价H-SRM方法的优势,我们用二个不同实验分别检测了代表24种人类血清蛋白的50种肽,一个是传统的SRM全扫描MS/MS方法检测,每个SRM测试使用了152组传输(每种肽2~3种离子对,每种蛋白1~4种肽)。第二个实验没有采用数据关联全扫描MS/MS,使用了318组SRM传输(5~7)(每种肽5~7种离子对,每种蛋白1~4种肽)。为评价H-SRM方法消除血清背景离子干扰的能力,此实验采用了高分辨(0.2 FWHM)和单位分辨(0.7FWHM)两种方式。
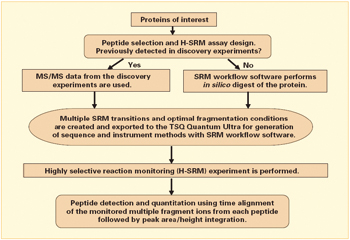
图2. 同时进行肽检测和定量的H-SRM工作流程。
TSQ Quantum Ultra质谱仪获得的MS/MS数据分别使用SEQUEST?和Mascot在SwissProt数据库进行了搜索,两种搜索引擎给出了相似的结果。表1总结了Mascot得到的QED-MS/MS实验数据的检索结果。可以看出,从24种靶向蛋白中都确认出21种蛋白(补体成分1抑制剂、血浆激肽释放酶和P<0.05的血红蛋白α链没有鉴定出来),TSQ Quantum Ultra质谱仪鉴定出36种肽。
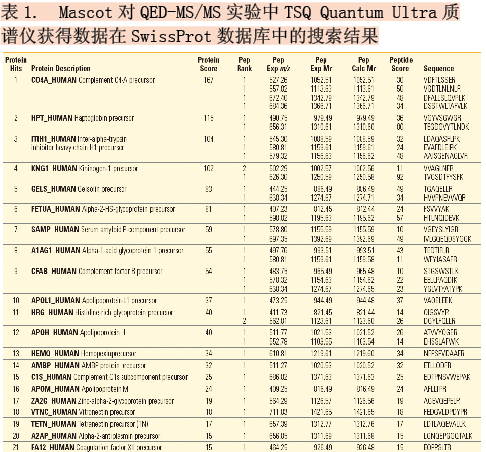
采用QED-MS/MS方法仍然没有成功识别鉴定血清消化液中所有的50种目标肽。这主要是由于传统方法在检测复杂基体中的低丰度肽具有局限性,尤其是肽具有共流出的同量异位干扰物时,无法对其进行可靠鉴定2。从复杂的血清消化液中鉴定24种蛋白产生的50种目标肽的任务,只有用我们新提出的方法才可以完成,该方法采用高分辨分离来检测低丰度肽,并用多碎片离子的时间序列来进行肽身份鉴定。图3是一种低丰度肽(LLDSLPSDTR代表补体组分1抑制剂),传统方法没有将其鉴定出来,H-SRM方法则将其成功鉴定出来。H-SRM的TIC谱(上图)信号峰信噪比与SRM的TIC谱(下图)相比大约高12倍。母体通过5种y-离子的时间序列检测得到了可靠的鉴定;Mascot序列搜索通过这些充分的数据搜索SwissProt数据库,可以唯一地鉴定该肽。该方法无需全扫描MS/MS,具有分辨率分离能力,在低检测限下快速可靠的鉴定目标肽方面具有很明显的优势。采用多碎片离子时间序列检测的H-SRM方法鉴定了所有的50种肽。
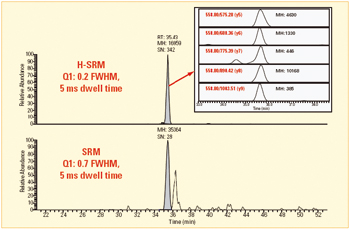
图3. 通过多碎片离子时间序列检测,H-SRM方法鉴定了代表补体组分1抑制剂蛋白的LLDSLPSDTR肽。与SRM方法相比,H-SRM方法显著提高了信噪比,降低了血清基体非特异性干扰,可以明确地鉴定目标肽。
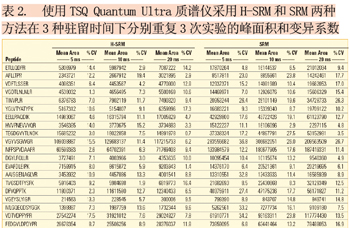
使用TSQ Quantum Ultra的H-SRM及SRM方法在不同驻留时间下测得的平均峰面积及变异系数(% CV)变化列于表2。H-SRM显著降低了来自血清基体的非特异性干扰,在不同驻留时间下均获得了较高的方法精密度,大约95%的肽的变异系数值小于15%。
定量准确度
H-SRM方法优良的分析精密度与其较高的定量准确度一致。H-SRM方法测定13种靶向蛋白的相对定量结果其平均误差范围为±4% (n=3)。
结论
本文介绍了TSQ Quantum
Ultra三级四极杆质谱仪采用H-SRM模式同时鉴定和定量靶向蛋白的新方法。该方法通过显著降低血浆背景离子的非特异性干扰,在人类血清中靶向蛋白的分析中,显示出良好的灵敏度、分析方法精密度和定量准确度。H-SRM方法极大地提高了方法专一性和检测限,在生物标志物的验证和确认研究中具有显著优势。
【参考文献】
1. Leigh Anderson and Christie L. Hunter (2006) Quantitative Mass
Spectrometric Multiple Reaction Monitoring Assays for Major Plasma
Proteins. Mol. Cell. Proteomics 5 (4), 573-588.
2. Hasmik Keshishian, Terri Addona, Michael Burgess, Eric Kuhn and Steven A. Carr (2007) Quantitative, Multiplexed Assays for low Abundance Proteins in Plasma by Targeted Mass Spectrometry and Stable Isotope Dilution. MCP Papers in Press. Published on October 15, 2007 as manuscript M700354-MCP200.
3. Richard D. Unwin, John R. Griffiths, Michael K. Leverentz, Agnes Grallert, Iain M. Hagan and Anthony D. Whetton (2005) Multiple Reaction Monitoring to Identify Sites of Protein Phosphorylation with High Sensitivity. Mol. Cell. Proteomics 4 (8), 1134-1144.3.