为什么检测2019-nCov用ORF1ab基因
为什么当前nCov的核酸检测试剂盒以ORF1ab、S和N作为目标基因?为什么单用ORF1b做进化就能推测nCov来源于蝙蝠?为什么用如此短的时间就能比较分析那么多种coronaviruses基因组从而否定重组阴谋论?我睡前思考着这些问题,索性搜搜文献看。精神食粮让我意识到自己是个无知的人类。
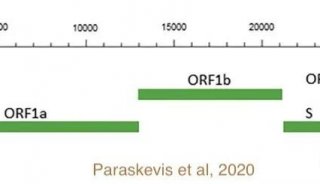
我检索了有关nCov基因组可视化的文献,截取了Paraskevis等人文章的组装注释图[1](按道理讲我更希望贴我国科学家发表在nature上的,无奈尺寸和配色不适用于手机屏幕),如下:

所以看看吧,这么小的基因组,就这么几个主要的基因,你还想用哪个做qPCR?!哈哈哈,我真的是如此愚钝。
此外,如此小的基因组,加上病毒基因组属于单正义链RNA(所以也不会出现高等植物多倍体、杂合等问题),对下游计算分析的硬件要求及耗时也将相应降低,分析个重组不重组的当然会更快。
Zumla等人对冠状病毒的生活史做了比较全面的综述[2]。病毒借助宿主系统进行后代的基因组扩增和外壳组装是一个最基本的战术动作。其中ORF1a和ORF1b开放阅读框有重叠,编码large replicase polyprotein 1a (pp1a)和pp1ab。这俩大蛋白在被蛋白酶剁吧剁吧以后,释放出病毒转录翻译依赖的蛋白。S基因决定病毒的相貌,从英文Spike protein就可以很好地理解它的功能。N(Nucleocapsid)、M(Membrane)、E(Envelope)基因编码病毒后期封装和胞吐相关的结构蛋白。理论上讲,选取那个基因做检测分析,重要的还是引物的特异性要好,灵敏度要高;同时我个人理解,尽量选取变异几率小的基因,这样能避免试剂盒的反复开发,加快“早发现早隔离”的进度。最初Takara第一版试剂盒明确说明了对N蛋白基因特异性不太好,估计近期会有改进。Hu等人提到ORF1a/b的变异性相对较小,而S变异性略高[3]。我猜最后检测策略应该是以ORF1a/b为主,其他基因为辅吧。
既然ORF1a/b是相对保守的,那么用ORF1b做病毒的进化分析就是相对科学的。
[1] Paraskevis D, Kostaki E G, Magiorkinis G, et al. Full-genome evolutionary analysis of the novel corona virus (2019-nCoV) rejects the hypothesis of emergence as a result of a recent recombination event[J]. Infection, Genetics and Evolution, 2020: 104212.
[2] Zumla A, Chan J F W, Azhar E I, et al. Coronaviruses—drug discovery and therapeutic options[J]. Nature reviews Drug discovery, 2016, 15(5): 327.
[3] Hu B, Zeng L P, Yang X L, et al. Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus[J]. PLoS pathogens, 2017, 13(11).
-
科技前沿
-
焦点事件
-
焦点事件











