前沿显微成像技术专题——超分辨显微成像(1)
从16世纪末开始,科学家们就一直使用光学显微镜探索复杂的微观生物世界。然而,传统的光学显微由于光学衍射极限的限制,横向分辨率止步于 200 nm左右,轴向分辨率止步于500 nm,无法对更小的生物分子和结构进行观察。突破光学衍射极限,一直是科学家们梦想和追求的目标。
虽然随着扫描电镜、扫描隧道显微镜及原子力显微镜等技术的出现,实现纳米级的分辨率已经成为可能,但是以上这些技术存在对样品破坏性较大,只能观测表面等缺点,并不适合生物样品,特别是活体样品的观测。近十几年来,一系列适合生物样品成像的超分辨成像技术应运而生,包括结构光照明(SIM),受激发射损耗荧光显微技术(STED),光激活定位显微技术(PALM),随机光学重建显微技术(STORM)等等。
光学分辨率极限
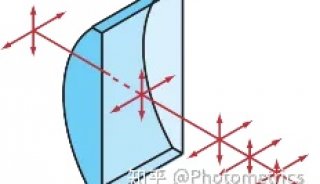
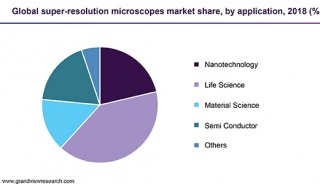
光以波的形式传播,当一个点光源通过透镜在成像面聚焦为一个小光点时,不管物镜有多好,成像光点都会比实际的发光点大。这是因为光波在物镜光阑的边缘会发生衍射,将波前向外扩散(图1)。
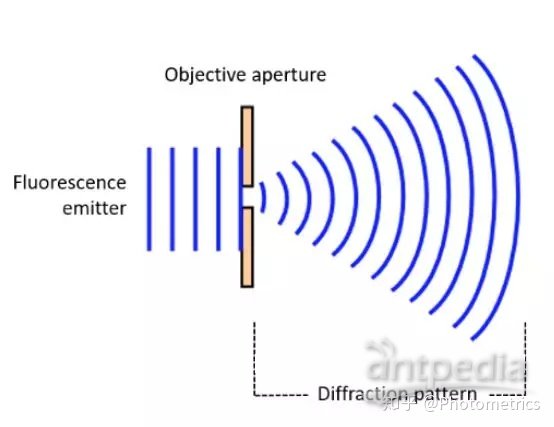
图1 发射荧光在物镜孔径边缘发生衍射
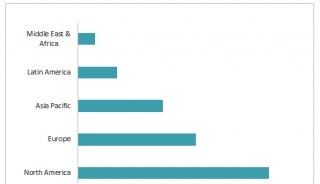
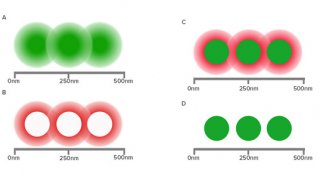
衍射光斑有一个明亮的中心点,以及环绕它的一系列亮度渐弱的衍射环,称为艾里斑。当两个点过于靠近,所成的像斑重叠在一起,就分辨不出是两个点的像了。因此,光学分辨率存在一个极限。根据瑞利判据,当一个艾里斑的中心与另一个艾里斑的第一级暗环重合时,刚好能分辨出是两个点所成的像(图2)。用公式表示如下:
d = 1.22λ / 2NA
d表示分辨率,λ是发射光的波长,NA是物镜数值孔径。

图2 光学分辨率极限示意
以GFP为例,λ=510nm,采用NA=1.4的物镜,光学分辨率的极限就是222nm。但是在生物系统中,膜蛋白,转运蛋白和核糖体等亚细胞结构往往小于50 nm,而且通常不会间隔足够远。这样一来,就不可能直接对其进行清晰的成像。
怎么办呢?科学研究的脚步当然不可能止步于此。为了克服衍射极限,研究人员开发了一系列超分辨成像方法。在本期中,我们将首先为大家介绍单分子定位超分辨显微成像技术~
这种超分辨显微技术都是通过选择性地打开和关闭单个荧光基团,确保成像区每次仅有少量、随机、离散的单个荧光分子发光,再通过高斯拟合,定位单个荧光分子(点扩散函数)的中心位置(图3),以实现高精度的空间定位,最后将系列图片叠加合成一幅超分辨图像。
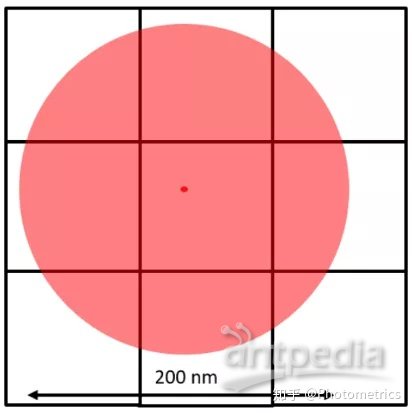
图3 通过高斯拟合进行定位
不同的技术所使用的“开关”荧光的方法不同,下面我们就来看看它们各自的原理和特点吧~
光激活定位显微技术(PALM)
PALM (Photoactivated Localization Microscopy)由Eric Betzig和Harald Hess于2006年首次发表于《Science》。它的原理其实非常简单,一句话概括就是通过“开关”与目标分子结合的光激活荧光蛋白,对其进行分批定位,确定中心光斑的位置。
光激活荧光蛋白PA-GFP是绿色荧光蛋白的变种,可以被适当波长(405nm)的激发光激活。实验时首先随机激活一部分荧光蛋白,再用488nm激发荧光,就可以只采集一部分目标分子的荧光信号。荧光蛋白被激活的概率与激发光的强度成正比,只要激活的蛋白足够少,相距足够远,就可以对其进行定位。完成数据采集后,对这些荧光蛋白进行光漂白直至完全失活,然后再激活另一些荧光蛋白进行定位。重复这个过程,就可以将样品中的所有目标分子定位,将这些原始数据合并,就能得到目标分子的超分辨图像。
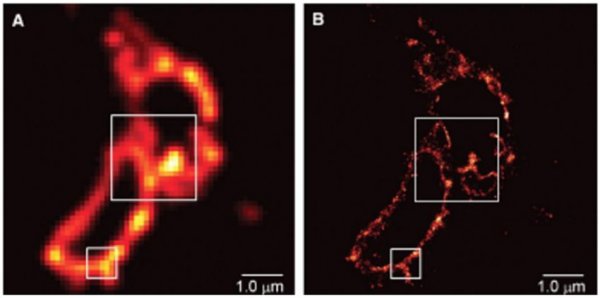
Betzig等人在文章中将PALM与全内反射荧光显微镜(TIRF)进行了比较(图4)。可以看到PALM对于荧光分子的定位精度远高于TIRF。
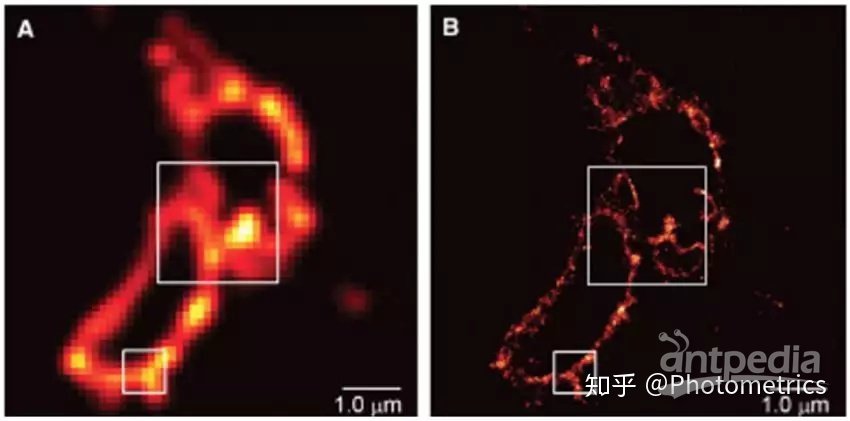
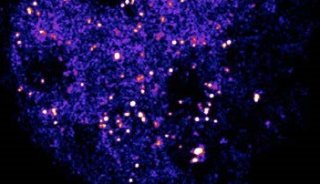
图4 TIRF(A)和PALM(B)对溶酶体膜的成像效果比较(Betzig et al., 2006)
随机光学重建显微技术(STORM)
STORM (Stochastic Optical Reconstruction Microscopy) 由华人科学家庄小威等人同样于2006年发表于《Science》杂志。它的基本原理与PALM类似,不同的是STORM使用合成的光转换荧光染料,而不是光激活荧光蛋白与目标分子结合。那么它是如何控制荧光的开关的呢?
荧光染料被激发进入发射态,之后会进入暗态(Dark state),在暗态中它们将与自由氧结合,进入漂白状态。在漂白状态下,染料不会再次发出荧光。如果不让染料与自由氧结合,它将无法进入漂白状态,一直维持在暗态。高功率的激发光可以使染料从暗态再次进入发射态。这种从亮到暗再到亮的状态切换看起来就像是染料在“闪烁”一样。
和PALM一样,STORM也是通过随机的分批“点亮”目标分子来进行超分辨定位(图5)。由于消除了光漂白步骤,STORM可以更快地采集数据。不过,STORM高度依赖于荧光染料的特性:既需要产生足够强的信号,同时又要有良好的闪烁密度。如果染料闪烁得太快,相邻的分子之间可能会有很多干扰,无法定位单个分子;但是如果它们闪烁得太慢,可能无法获得足够的图像来定位每个分子。STORM最初使用的染料是cy3和cy5,不过目前最流行的是Alexafluor 647。
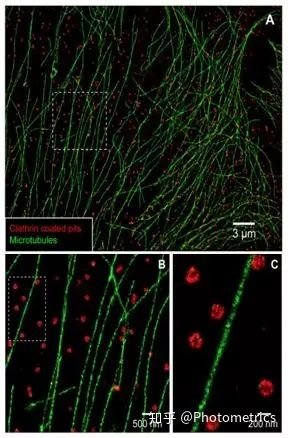
图5 哺乳动物细胞微管和CCP的双色STORM成像(Bates et al., 2007)
DNA-PAINT
DNA-PAINT (DNA-Based Point Accumulation for Imaging in Nanoscale Topography) 是一种比PALM和STORM更新的技术,由Ralf Jungmann等人于2014年发表。它的原理是利用DNA链的互补性产生类似于荧光分子闪烁的效果。
在DNA-PAINT中,双螺旋DNA链的其中一条与目标分子相连,作为“对接”链,另一条被荧光标记,作为“成像”链。由于DNA链彼此之间是高度特异性结合的,因此成像链和对接链在溶液中会自发结合,在焦平面上产生单分子荧光。通过调整成像链的结合强度和浓度,可以反复产生瞬时结合,获得类似STORM中荧光染料的最佳闪烁速率。对获得的数据进行重建,就可以得到最终的超分辨率图像(图6)。
除了可调控的结合速率外,DNA-PAINT的另一个显著优点是任何荧光染料都可以用于成像 链,便于进行多色成像。
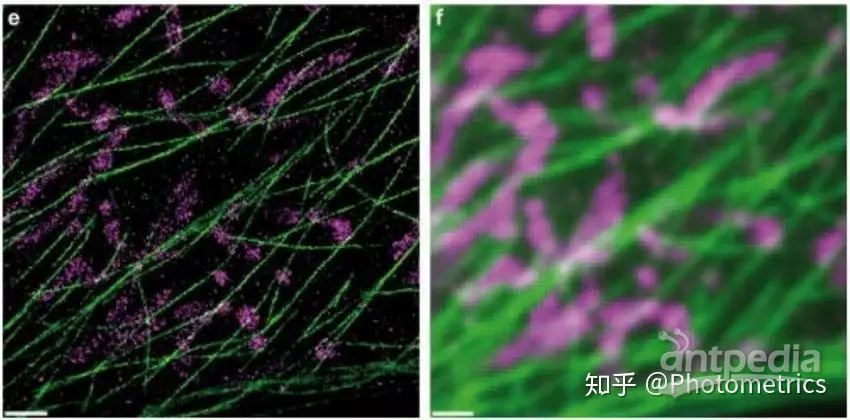
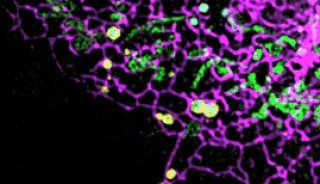
图6 DNA-PAINT获得的Hela细胞微管 (绿色) 和线粒体 (洋红色) 的双色超分辨率图像(e)和相同区域的衍射极限图像(f)效果对比(Jungmann et.al., 2014)
PALM,STORM等单分子定位超分辨显微成像技术自诞生以来,被广泛的应用到了各种研究领域,极大地增强了定位亚细胞结构与探索它们之间相互关系的能力。譬如对局部粘着复合物及其分布进行纳米尺度的成像,细胞骨架中细微结构的成像等。
那么,超分辨显微定位的分辨率是由什么决定的?获得的大量图像数据如何进行重构?……请听下回分解。
References
Bates, W. M., Huang, B., Dempsey, G. T., and Zhuang, X. (2007) Multicolor Super-resolution Imaging with Photoswitchable Fluorescent Probes. Science. 317(5845): 1749–1753. doi: 10.1126/science.1146598
Betzig, E., Patterson, G.H., Sougrat, R., Lindwasser, O. W., Olenych, S., Bonifacino, J. S., Davidson, M. W., LippincottSchwartz, J., Hess, H.F. (2006) Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313(5793): 1642-5; DOI: 10.1126/science.1127344
Jungmann, R., Avendaño, M. S., Woehrstein, J. B., Dai, M., Shih, W. M. & Yin, P. (2014) Multiplexed 3D cellular superresolution imaging with DNA-PAINT and Exchange-PAINT. Nat Methods. Mar; 11(3): 313–318. doi: 10.1038/nmeth.2835
Rust, M. J., Bates, M. & Zhuang, X. (2006) Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Methods. Oct;3(10):793-5. DOI: 10.1038/nmeth929
Schnitzbauer, J., Strauss, M. T., Schlichthaerle, T., Schueder, F. & Jungmann, R. (2017) Super-resolution microscopy with DNA-PAINT. Nat Protoc. Jun;12(6):1198-1228. doi: 10.1038/nprot.2017.024