新型CUT&RUN技术实现单细胞全基因组定位
在一项新的突破性研究中,来自美国匹兹堡大学和马萨诸塞大学医学院的研究人员对一种称为CUT&RUN(cleavage under targets and release using nuclease)的方法进行改进,使得在使用少量细胞(包括单细胞和单个植入前胚胎)的情形下,它适合用来研究转录因子和其他的DNA结合蛋白在染色质上的占据情况。相关研究结果于2019年4月4日在线发表在Cell期刊上,论文标题为“Profiling of Pluripotency Factors in Single Cells and Early Embryos”。论文通讯作者为匹兹堡大学的Sarah Hainer和马萨诸塞大学医学院的Thomas G. Fazzio。
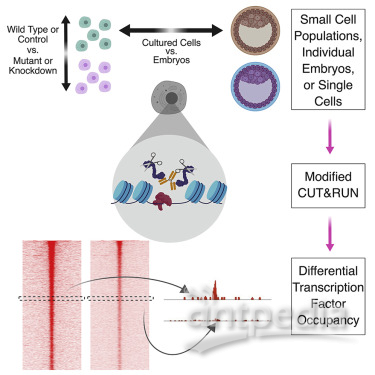
图片来自Cell, 2019, doi:doi:10.1016/j.cell.2019.03.014。
鉴于CUT&RUN可确定蛋白在染色质上的定位,它类似于广泛使用的染色质免疫沉淀(ChIP)技术。然而,目前可用的DNA结合蛋白全基因组定位方法需要数万至数百万个细胞。因此,DNA结合蛋白的体内定位方法受到严格限制,这是因为许多生物学上重要的细胞群体具有较低的细胞数量。
CUT&RUN最初于2017年经过改进,已成功应用于由1000多个细胞组成的细胞群体中。Hainer和Fazzio试图进一步改进这种技术,进一步改进的CUT&RUN称为超低输入CUT&RUN(ultra-low input CUT&RUN, uliCUT&RUN),并且在这项新的研究中,利用uliCUT&RUN技术首次描述了多能性因子在单细胞和单个植入前胚胎中的全基因组定位。
利用这种新技术进行的实验表明,在大多数细胞中,仅有一小部分转录因子结合位点被占据,这就证实了通过多细胞研究获得的测量结果。它还表明uliCUT&RUN可用于在细胞发育或疾病起关键作用的罕见细胞群体中研究转录因子结合情况。
Hainer说,“通过将定位研究推进到单细胞和单个胚胎水平,未来的研究可以着重关注细胞异质性和对有限生物样品的研究。通常,在组织样品中,细胞数量和细胞纯度之间的权衡阻止使用ChIP-seq分析纯化的组织特异性细胞群体。uliCUT&RUN能够从50个细胞中获得转录因子结合图谱,这些图谱与利用较高细胞数量获得的图谱高度重叠,这使得几乎可以从任何可用的样品中获得转录因子结合图谱。”