| 实验步骤 | 3.1 芯片背景
我们使用了 19846 个点,包含 9246 个 Unigene 序列的小麦 cDNA 芯片(http://www . cerealsdb. uk. net/index. htm) 用于详细的转录组研究 [3 ] 。重复的 unigene 集合阵列点到了 Codelink 活化芯片上(Amersham Biosciences Ltd, UK ) 。
芯片杂交用与 aa- dUTP- cDNA 样品荧光标记染料反向的染料——Alexa 荧光染料 555 和 647。杂交在转基因系 (B 102-1- 1、B1118-8-4 或 B1355-4-2 ) 和其原始非转基因系 ( L 88-31 或 Cadenza ) 之间的两个胚乳发育阶段(开花后 14 天和 28 天,dpa ) 和叶片 ( 发芽后 8 天,dpg ) 成对比较 [5]。每个材料取样 3 次,检测 2 次。
cDNA 芯片的优势是经济实惠,并允许使用者完全控制内容和设计(定制芯片)。相反,Affymetrix 寡核苷酸芯片更加灵活,它是单染料系统,使试验更简单、更特异(提高对类似的异构体和多基因家族成员的识别能力),提供更量化和易比较的数据。GeneCMp 基因组芯片,采用了一套能匹配任何转录序列的 25 碱基寡核苷酸“探针”。以小麦芯片为例,每组探针有 11 个,多数与组装的序列表达标签( EST ) 的公共保守域序列一致。因此,在 cDNA 芯片中,每一组探针对应的是许多 EST,而不是单个的 EST。为了识别不同的转录本,同时满足其他条件,如 GC 含量相对一致,探针通过程序自动化设计。对于一个目标转录本,设计了 一个完全匹配的探针(perfect matches,PM ) 、一个单碱基错配探针(mismatches,MM ) ,这样能对非特异杂交有一个估计和矫正。然而,对于 MM 探针的真正信号值存在争论,许多广泛使用的分析方法不用 MM 探针( 见 3.10节)。短 探针( 如 25-mer) 对于序列相似的转录本更有效,因为单个碱基错配足够使杂交不稳定,而且固定长度使得杂交条件可以标准化,以满足所有探针;相反,较长的可变长度的探针,比如那些 cDNA 芯片平台使用的探针,将不可避免与任何与其部分序列相似性探针杂交,所以其真实信号集成了几个不同的转录分子。
3.2 植物材料和生长条件
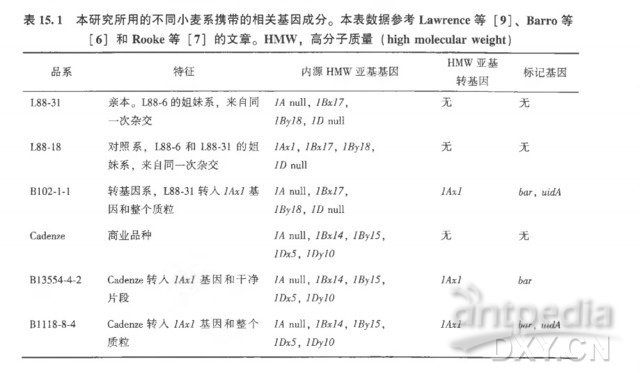
转录组比较研究使用了三个六倍体转基因面包小麦的胚乳和叶片。转基因小麦品系 B102 -1-1 ( L 88-31 背景) [ 6, 7 ] 和 B1118-8-4 ( Cadenz 北背景)[ 5 ] 由基因枪共转化两个质粒产生[ 14 ] 。一个质粒是 p1Axl 质粒 [ 13 ] ,含有由自身的胚乳特异启动子驱动的高分子质量谷蛋白亚基 1AX1 ( Glu-M ) 基因; 另一个质粒含有选择基因bar 和标记基因 uidA,由玉米泛素启动子驱动。转基因系 B1355-4-2 [ 5 ] 也来自 Cadenza,共转化获得只含 IAX1 基因和 bar 基因编码序列的“干净”片段。一个常规育种系 L88-18 [ 9 ] 是 L88- 31 的姐妹系,也用于转录组比较。两个常规育种系 ( L 88-31、L 88-18 ) 和转基因系(B 102-1-1 ) 的转录组两两比较是:B 102-1-1与 L 88-31、L 88-18与 L 88-31、 B 102-1-1与 L 88-18。转化方法的比较,即 “干净”片段与整个质粒的比较:B1355-4-2 与 Cadenza、 B1118-8-4 与 Cadenza、 B1355-4-2 与 B1118-8-4。本研究所用的不同面包小麦携带的相关基因成分的详细信息见表15 . 1 。
( 1 ) 植物种在平衡行列设计的盆钵中,每个处理(小麦生长发育阶段)有 3 个生物学重复。
( 2 ) 开花后 14 天和 28 天胚乳,取样的植物每盆种 2 株。每一株只保留 2 个分蘖。选中的盆钵(本试验为 3 个生物学重复)包括用于发芽后第 8 天取叶片的第三株植物。
( 3 ) 每天观察穗,一旦发现中央小穗开花就做标记。
( 4 ) 在无菌条件下,手工从颖果剥离种子胚乳;每盆只从 2 个穗子的中部分别取至少 24 枚胚乳为一个样品。
( 5 ) 样品都是一天中同一时间取样,以避免昼夜节律的影响。
3.3 SDS-PAGE
通过谷粒总蛋白 SDS-PAGE 凝胶电泳,检测所有小麦系的高分子质量亚基蛋白的表达(图 15. 1 ) ,使用 10% ( m/V) 丙烯酰胺凝胶和 Tris-硼酸缓冲液系统 [ 10 ] 。
3.4 RNA 提取
3.4.1 小麦胚乳总 RNA 提取
提取方法根据 Chang 等 [ 11 ] 改编而来。
( 1 ) 用预冷的研钵和杵(-70°C ) 在液氮里将 2~3 g 组织磨成粉末(见注 8 ) 。
( 2 ) 室温下迅速将磨碎组织转移到有 15 ml 提取缓冲液(加入 300 μl β-巯基乙醇 ) 的离心管中,充分颠倒混匀( 见注9) 。

( 3 ) 用等体积氯仿:异戊醇(终体积 15 ml ) 抽提两次,液相分离用 15000 g 室温离心 10 min。
( 4 ) 上清液加 0.25 倍体积 10 moI/L LiCl 混匀。4℃ 过夜沉淀 RNA,4℃、15000 g,离心 20 min 获取 RNA。
( 5 ) 500 ml SSTE 悬浮沉淀颗粒。
( 6 ) 用等体积氯仿:异戊醇抽提一次。
( 7 ) 上清液加两倍体积乙醇,-70℃ 沉淀 30 min 以上或者 -20℃ 沉淀 2 h。
( 8 ) 15000 g 离心 20 min 沉淀 RNA。
( 9 ) 75% 乙醇洗涤。
( 10 ) 干燥沉淀并溶解于无核酸降解酶水中。
3.4.2 发芽后 8 天幼苗 RNA 提取
提取方法根据 Cheng 等 [ 12 ] 改编而来。
( 1 ) 用预冷的研钵和杵(-70℃ ) 加液氮里将已知质量组织(大约 1 g 幼叶)磨成粉末(见注9) 。
( 2 ) 将冷冻的粉末快速转移到第二个有 10~15 ml 匀浆缓冲液的研钵中(见注10),继续研磨至均匀(见注 11)。
( 3 ) 匀浆液转移到 50 ml 带帽的圆底旋盖离心管中,60℃ 孵育 10 min ( 匀浆液终体积 5~10 ml )。
( 4 ) 离心管置冰上冷却,加 0.2 倍体积 pH 5.5 的 5 mol/L 乙酸钾(见注 12) ,轻轻地充分混匀,然后冰上静置 10~15 min。
( 5 ) 10000 g、4℃ 离心 15 min , 取上清液到新的离心管中。
( 6 ) 加等体积酚:氯仿(1 : 1,V/V ) 混匀,拧紧瓶盖并剧烈振荡 10 min。10000 g、21°C 离心 10 min。取上层水相于新离心管中(见注13)。
( 7 ) 水相重复上一步的酚:氯仿萃取和分层。
( 8 ) 往水相加 0~1 倍体积 3 mol/L pH 5.3 乙酸钠和 2.5 倍体积乙醇。混匀后 -20℃ 孵育过夜以提高核酸沉淀效率。
( 9 ) 10000 g 、4℃ 离心 30 min 沉淀核酸,弃上清。倒置离心管数分钟。
( 10 ) 少量无核酸降解酶水( 300~700 μl ) 溶解沉淀。核酸溶液转移到 Eppendorf 管,加 0.67 倍体积 10 mol/L 氯化锂沉淀 RNA。拌匀,冰上孵育 20~30 min ( 见注14)。
( 11 ) 离心(15000 g,室温,20 min ) 沉淀 RNA,弃上清。
( 12 ) 用尽可能小体积( 200~300 μl) 的无核酸酶水溶解沉淀,重复氯化锂沉淀,和上一步同样离心沉淀 RNA。
(13 ) 200 μl 无核酸酶水溶解沉淀,加入 15 μl 5 mol/L 乙酸钾(pH 5.5 ) 和 800 μl 乙醇。混匀,离心(15000 g 室温,20 min) 沉淀 RNA ( 见注15)。
( 14 ) 弃上清,加入 1.0 ml 80% ( V/V ) 乙醇,离心洗涤 RNA ( 15000 g,室温,10 min)。
( 15 ) 离心管开盖置于超净工作台,室温干燥沉淀不超过 10 min。溶解于 100~200 μl 无核酸酶水中。将每份样品分成等量小份以避免在反复冻融过程中污染或者降解。
3.4.3 总 RNA 样品除去基因组 DNA
RNA 提取之后用 DNA-free ( DNA 酶处理和清除试剂盒,Ambion) 按照使用说明处理 RNA 片段。该系统专为去除 RNA 样品中的 DNA 杂质和清除处理后的 DNA 降解酶 I,无需加热或苯酚提取。
3.4.4 RNA 定量分析和质量控制
RNA 浓度、完整性及质量检测使用 Nanodrop ND 1000 分光光度计(Labtech Int ,U K ) 和 Agilent 2100 生物分析仪 ( RNA 6000 Nano Assay, Agilent Technologies, PaloAlto, CA,USA ) ( 见注 16)。
3.4.5 样品保存
RNA 样品短期(最长 3 个月)保存在 -20℃,长期则在 -80℃。
3.5 cDNA 合成
( 1 ) 加 100 μg DNA 酶处理过的总 RNA ( 不超过 20 μl) 、8 μl oligo (dT)23 锚定引物和无核酸酶水至终体积 28 μl。
( 2 ) 将启动反应混合物在 70℃ 孵育 10 min,置于冰上 5 min。
( 3 ) 向启动反应混合物加入 cDNA 合成反应混合物:10 μl 5x 第一链缓冲液,10 μl 0.1 mol/L DTT,5 μl 50x aa-dNTP 混合物,2 μl Superscript 逆转录酶Ⅲ( 200 U/L ) ,加无核酸酶水至终体积 50 μl。
( 4 ) 42℃ 孵育 2~3 h。
( 5 ) 用微型离心柱(Qiagen) 参照使用说明纯化 aa-dUTP-dDNA 产物。
( 6 ) 收集最后的洗脱液 10 μl 作为样品。cDNA 的产品将用于准备芯片杂交的探针和实时 RT- PCR 技术。最后反应可不用 50x aa-dNTP 混合物进行(见注 17)。
3.6 cDNA 芯片标记
( 1 ) 为了 cDNA 与突光染料偶联反应,将需要反向染料标记的总 cDNA ( 见第3.5节步骤6 ) 分成两等份(5 μl ) ,并将不同梁料的反应放在单独的管。
( 2 ) 每管加入: 5 μl 氨基烯丙基纯化的 cDNA ( 见 3 . 5 节步骤 3 ) ,3 μl 1 mol/L NaHCO3 标记缓冲液,2 μl ALexa 荧光染料 555 或者 647,10 ml 无核酸酶水。
( 3 ) 移液器混匀,室温黑暗孵育 1 h。
( 4 ) 用 mini Elute columns 试剂盒(Qiagen) 去除没有与 aa-dUTP- cDNA 偶联的染料( 见注18)。
3.7 cDNA 芯片杂交
( 1 ) 取 20 μl 混合的标记 cDNA ( 来自 3. 6 节步骤 6 的两个 Alexa 染料)加入到 25 μl 2x 杂交缓冲液和 2 ml poly (dA ) 。
( 2 ) 探针在 95℃ 3 min 变性。
( 3 ) 标记的探针涂在盖片上,并将载片印有 Code Link 面朝下放置。
( 4 ) 将芯片杂交盒置于烘箱内 42℃ 过夜。
( 5 ) 将芯片置于含有溶液 A 的 Falcon 管(蓝帽)中(见 2. 6 节步骤 2 ) ,室温颠倒 15 min (见注 19)。
( 6 ) 将芯片转移到第二个含有溶液 A 的 Falcon 管中,室温颠倒 15 min。
( 7 ) 将芯片转移到第三个含有溶液 B 的 Falcon 管中( 见 2 . 6 节步骤3 ) ,室温颠倒 15 min。
( 8 ) 将芯片转移到第四个含有溶液 C 的 Falcon 管中(见 2 . 6 节步骤3 ) ,室温颠倒 15 min。
( 9 ) 将芯片放置干燥的 Falcon 管中,立刻 8000 g 离心干燥。
( 10 ) 用 Axon 仪器公司的 Gene-Pix 400B 型双激光扫描仪扫描杂交芯片。
3.8 cDNA 芯片数据分析
芯片做图像分析,检测每一个观测点的两种荧光信号的强度,以此来评估成对小麦系之间转录基因的表达差异。数据标准化后,作适合某个模型的统计分析,说明实验设计 ,检测差异表达的显著性。
3.8.1 图像分析和标准化
芯片上的点用 GenePix 软件扫描成图像(Gene Pix 第 5 版,美国 Axon 仪器公司),然后所有的点进行人工检测,排除杂交失败或者信号弱的。图像分析给出了每一点的所有像素,这些数据包含了两种荧光染料信号(647和 555 ) 的平均值和 Log2 比率值。这些数据专用于差异表达分析。将数据从 GenePix 导入 GeneSpring 软件包 (GeneSpring 6. 2 美国硅遗传公司),然后信号强度的 Log2 比率值进行标准化。本研究中,一个点的 Log2 比率值(差异表达的)( M ) 和 Log2 乘积(亮度的)( A ) 之间的比较表明,局部加权光滑描点技术( LOWESS ) 标准化处理,可用于消除数据不佳的趋势。换句话说,所有点的 Log2 比率值乘以 ( across ) 光 强 ( Log 乘积)应该在一个恒定范围内,但图像显示有一些会随着 A 増加而发生变化的趋势。因此,标准化处理的目的是消除 Log2 比率数据的系统变异( 比如由于实验过程)。局部加权光滑描点技术( LOWESS ) 标准化处理,是通过逐点检测 M 和 A 值之间的关系,然后相应地调整 M 值(adjusted M = MLOWESS- fittedM) 。由于有两个独立的实验, L 88-31、 L 88-18和 B 102-1-1的数据与 Cadenza的数据分开处理。
3.8.2 统计分析
参考 Kerr [ 15 ] 讨论的方法,使用 GenStat 统计系统( GenStat 第 7 版 ,GenStatProcedure Library Release PL15, Lawes Agricultural Trust, Rothamsted Research Harpenden, U K ) 分析规范化的数据( 进一步的详细信息见[ 5 ] 及其补充材料)。在估计基因的固定效应之前,用符合 Log2 比率的线性混合模型来估算实验设计( 生物学和技术重复)的随机效应。该模型通过模型计算用数据的整体残差( 噪声)估计标准误参 数(一个基因一个参数)。每个参数对其标准误的比率服从残留自由度的 t 统计分布,这使得从 0 (Log2 范围)开始的差异表达统计上显著性得到评估。在我们的研究中,通过表达量和拷贝数筛选显著差异表达基因( P <0.05 ),只有那些表达差异大于 1.5 倍并且存在多 2 次以上重复的基因被保留下来做进一步分析。
3.8.3 用实时 RT-qPCR 验证差异表达
挑选转录本做实时 RT- qPCR 验证芯片表达数据,见 3.13 节。
展开 |
|---|
