400-6699-117转1000
咨询列表
上海百趣生物医学科技有限公司
您好,欢迎您查看分析测试百科网,请问有什么帮助您的?



诚信认证:
工商注册信息已核实! 扫一扫即可访问手机版展台
扫一扫即可访问手机版展台
| 参考报价: | 10000 - 30000 RMB(人民币) | 型号: | 反向(lncRNA)靶基因预测 |
| 品牌: | 阿趣生物 | 产地: | 上海 |
| 关注度: | 543 | 信息完整度: | |
| 样本: | 典型用户: | 暂无 |
400-6699-117转1000
服务内容:
与常规的从lncRNA出发预测靶基因不同,反向long noncoding RNA(lncRNA)靶基因预测是从感兴趣的已知基因开始(例如p53,COX-2等与肿瘤等密切相关的基因,这些基因可以来自芯片分析结果、NLP结果或客户给定的任何基因列表),利用我们的long noncoding RNA(lncRNA)数据库和靶基因预测软件pipeline,找到潜在的调节这些基因的lncRNA。
研究方法:
我们的服务提供:
1.获取感兴趣的基因集合
这些基因可以来自芯片分析结果、NLP结果或客户给定的任何基因列表。如p53,COX-2。
2.建立lncRNA数据库
我们通过整合已知所有lncRNA数据库,形成一个全面的包含所有lncRNA序列的数据库
3.细胞组织特异高表达lncRNA的鉴定(可选)
根据客户提供的细胞组织信息,我们搜索已有的tiling芯片以及新一代测序得到的转录组信息,从全基因组已知lncRNA的中鉴定高表达的lncRNA。
4.cis靶基因预测
Cis作用[5]是lncRNA的一种靶基因作用方式。我们利用基因组注释和基因组浏览器鉴定lncRNA的可能的靶基因。一般在启动子区域同向转录的靶基因一般是促进表达作用,反向为抑制。而在3’-UTR区域时,部分情况下反向也为促进表达,见[14]。
5.trans靶基因预测
trans作用是lncRNA的另一种靶基因作用方式。我们利用RNAplex(http://www.tbi.univie.ac.at/~htafer/)等鉴定lncRNA的可能的靶基因,一个例子请见[15]。
综上,一个可能分析流程如下: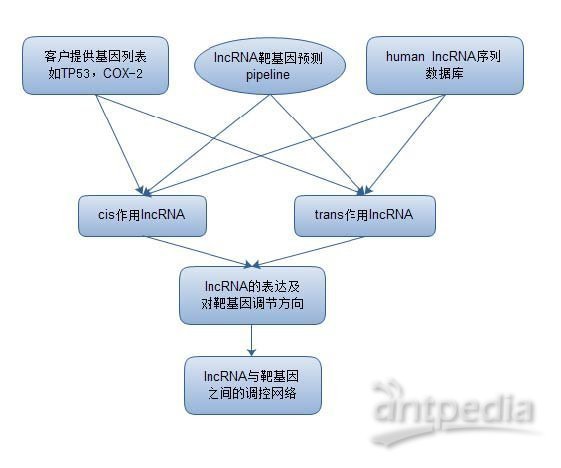
参考文献:
[1] Furuno M, Pang KC, Ninomiya N, Fukuda S, Frith MC, Bult C, Kai C, Kawai J, Carninci P, Hayashizaki Y, Mattick JS, Suzuki H. Clusters of internally primed transcripts reveal novel long noncoding RNAs. PLoS Genet. 2006 Apr;2(4):e37. Epub 2006 Apr 28.
[2] Khalil AM, Faghihi MA, Modarresi F, Brothers SP, Wahlestedt C. A novel RNA transcript with antiapoptotic function is silenced in fragile x syndrome. PLoS ONE. 2008 Jan 23;3(1):e1486.
[3] Mercer TR, Dinger ME, Sunkin SM, Mehler MF, Mattick JS. Specific expression of long noncoding RNAs in the mouse brain. Proc Natl Acad Sci U S A. 2008 Jan 9; [Epub ahead of print]
[4] Ponjavic J, Ponting CP, Lunter G. Functionality or transcriptional noise? Evidence for selection within long noncoding RNAs. Genome Res. 2007 May;17(5):556-65. Epub 2007 Mar 26
[5] Petruk S, Sedkov Y, Riley KM, Hodgson J, Schweisguth F, Hirose S, Jaynes JB, Brock HW, Mazo A. Transcription of bxd noncoding RNAs promoted by trithorax represses Ubx in cis by transcriptional interference. Cell. 2006 Dec 15;127(6):1209-21
[6] Pang KC, Frith MC, Mattick JS. Rapid evolution of noncoding RNAs: lack of conservation does not mean lack of function.Trends Genet. 2006 Jan;22(1):1-5. Epub 2005 Nov 10.
[7] Perez DS, Hoage TR, Pritchett JR, Ducharme-Smith AL, Halling ML, Ganapathiraju SC, Streng PS, Smith DI. Long, abundantly expressed non-coding transcripts are altered in cancer. Hum Mol Genet. 2008 Mar 1;17(5):642-55. Epub 2007 Nov 15.
[8] Franco-Zorrilla JM, Valli A, Todesco M, Mateos I, Puga MI, Rubio-Somoza I, Leyva A, Weigel D, García JA, Paz-Ares J. Target mimicry provides a new mechanism for regulation of microRNA activity. Nat Genet. 2007 Aug;39(8):1033-7. Epub 2007 Jul 22.
[9] Rinn JL, Kertesz M, Wang JK, Squazzo SL, Xu X, Brugmann SA, Goodnough LH, Helms JA, Farnham PJ, Segal E, Chang HY. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell. 2007 Jun 29;129(7):1311-23.
[10] Hüttenhofer A, Vogel J. Experimental approaches to identify non-coding RNAs. Nucleic Acids Res. 2006 Jan 25;34(2):635-46. Print 2006.
[11] Maeda N, Kasukawa T, Oyama R, Gough J, Frith M, Engström PG, Lenhard B, Aturaliya RN, Batalov S, Beisel KW, Bult CJ, Fletcher CF, Forrest AR, Furuno M, Hill D, Itoh M, Kanamori-Katayama M, Katayama S, Katoh M, Kawashima T, Quackenbush J, Ravasi T, Ring BZ,
Shibata K, Sugiura K, Takenaka Y, Teasdale RD, Wells CA, Zhu Y, Kai C, Kawai J, Hume DA, Carninci P, Hayashizaki Y. Transcript annotation in FANTOM3: mouse gene catalog based on physical cDNAs. PLoS Genet. 2006 Apr;2(4):e62.
[12] Ma XH, Hu SJ, Ni H, Zhao YC, Tian Z, Liu JL, Ren G, Liang XH, Yu H, Wan P, Yang ZM. Serial analysis of gene expression in mouse uterus at the implantation site. J Biol Chem. 2006 Apr 7;281(14):9351-60. Epub 2006 Jan 24.
[13] Khalil AM, Faghihi MA, Modarresi F, Brothers SP, Wahlestedt C. A novel RNA transcript with antiapoptotic function is silenced in fragile x syndrome. PLoS ONE. 2008 Jan 23;3(1):e1486.
[14] Mohammad ALI Faghihi et al. Evidence for natural antisense transcript-mediated inhibition of microRNA function. Genome Biology 2010, 11:R56
[15]Laura Poliseno et al. A coding-independent function of gene and pseudogene mRNAs regulates tumour biology. Nature, 24 June 2010.
反向long noncoding RNA(lncRNA)靶基因预测服务信息由上海百趣生物医学科技有限公司为您提供,如您想了解更多关于反向long noncoding RNA(lncRNA)靶基因预测服务报价、型号、参数等信息,欢迎来电或留言咨询。
注:该产品未在中华人民共和国食品药品监督管理部门申请医疗器械注册和备案,不可用于临床诊断或治疗等相关用途