检测DNA甲基化维持效率的方法研究中获新进展
3月8日,中国科学院生物物理研究所研究员朱冰课题组在Nature Protocols上,在线发表了以Simultaneously measuring the methylation of parent and daughter strands of replicated DNA at the single-molecule level by Hammer-seq为题的研究论文。
在以往工作中(Ming et al., Cell Research. 2020),朱冰课题组为研究复制叉过后DNA甲基化维持的动态过程以及调控机制,开发了Hammer-seq(Hairpin-assisted mapping of methylation of replicated DNA)。该方法结合了EdU(胸腺嘧啶类似物)标记新合成DNA链、点击化学反应在EdU位点添加biotin标签、Streptavidin亲和纯化和Hairpin全基因组DNA甲基化测序等技术,能够在DNA复制的不同时期,同时检测亲链和子链的DNA甲基化状态,进而可以研究DNA甲基化的维持动态及其调控机制。在Hammer-seq的开发过程中,该方法也在该课题组和清华大学教授颉伟课题组的一项合作工作中得到了应用(Wang et al., Nat Genet. 2020)。
为拓展Hammer-seq的应用范围,科研人员详细描述了Hammer-seq的设计思路、开发过程、实验步骤、优点和缺点等,展望了Hammer-seq潜在的应用场景,其中包括:解析复制叉过后新合成DNA链的甲基化维持速率;检测甲基化维持过程中伴随发生的从头甲基化事件;结合突变细胞系的构建,解析相应基因在调控甲基化动态维持过程的作用机制等。此外,简化版的Hammer-seq还可用于构建Hairpin全基因组DNA甲基化测序文库,且相比于原有方法更加地简洁和易于操作。
该工作得到科技部、国家自然科学基金委员会和中科院等的资助。生物物理所博士明轩为论文第一作者,研究员张珠强为论文通讯作者,朱冰为论文共同作者。
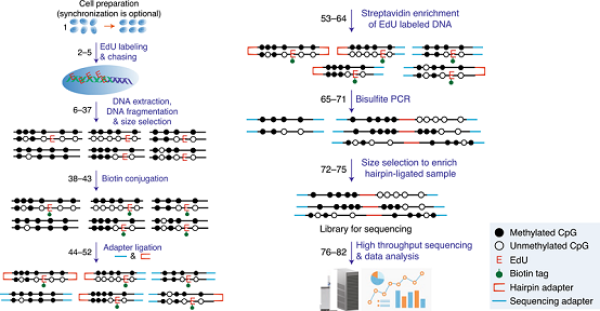
Hammer-seq建库和分析流程图