活细胞提取及应用——单个细胞级别的活细胞提取
由于细胞异质性的存在,单细胞层面的分析就变得十分重要。目前对于单细胞分析的方法主要还是通过化学、生物学的方法进行裂解后,提取内容物进行分析,然而这种方法往往会对样本造成一些损伤。直接提取活细胞具有诸多优点,但是操作苦难。如今一种全新使用FluidFM科技的技术新报道有望提供一种活细胞提取新型的简易方法。
1.为什么要进行活体单细胞提取
随着技术的发展,对于细胞的研究开始向单细胞领域的分析靠拢。随着细胞异质性的发现以来,人们开始更多地认识到即使基因型完全相同,但由于基因表达的不同,也会导致细胞的表型出现差异,从而使得每个细胞的功能、组成等各方面也不完全相同。而这种差异是在生物界广泛存在的,即使同源细胞群中也是存在的。因此对于单细胞层面的分子分析,成为了揭示病理特征,细胞应激等方面的重要手段。
在目前的单细胞分析手段中也面临着诸多挑战。目前主要的方法是使用流式细胞仪或者微流控芯片对细胞进行分选并隔离单细胞,然后通过裂解的方式对细胞进行分析。这种高通量分选的方式虽然快速,但是本身需要将细胞从原始的培养环境中取出,这样始终会导致细胞内部一些生物信息的丢失或损伤。而且由于细胞破坏方式使用的是化学或者生物手段裂解,所使用的裂解液也会对细胞之中的一些组分产生破坏。因此单细胞分析始终面临着重大的挑战。
因此,学者开始尝试从活细胞中直接提取其成分。目前已经有诸多不同的活细胞提取技术得到了建立,并且通过这类的方法所提取分析内容的方法所获得的结果往往要好于裂解的方式。因此活细胞提取技术是一种更加无损的获得细胞内组分的方式。
2.使用FluidFM设备提取细胞并不会影响细胞活力
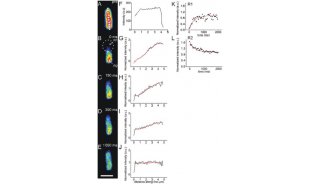
在本文中,作者对首先建立了一种使用FluidFM提取细胞的方法。他们尝试了各种提取量以确定使用FluidFM能够对细胞提取的最大量。在实验中他们发现使用FluidFM抽取4 pL的细胞质并观测,发现82%的细胞依然存活,如图1B所示。但是如果将抽取量加大到4.5 pL则无细胞能够存活。而对细胞核而言抽取0.6 pL后,有86%的细胞依旧存活,如图1A。而在0.7 pL或以上的体积提取则不可避免的导致细胞死亡,如图1B所示。随后他们对提取后细胞的存活状况进行了考察。而后他们继续对提取过的细胞进行观察,结果发现这些细胞的功能并没有受到影响。细胞能够在提取后约30小时后正常分裂,如图1E所示。因此作者认为使用FluidFM提取的最大量为细胞核0.6 pL和细胞质4pL。
3.使用FluidFM单细胞提取物的三种应用
3.1透射电镜负染色观测
对于细胞亚结构的观察,往往对于揭示细胞病变有着重要的意义。然而细胞裂解的传统手段往往会产生大量的碎片,因此对细胞器的观察造成了诸多困难。在本篇报道中,作者通过使用FluidFM设备提取细胞内容物,在低温环境下转移到透射电镜的铜网上,然后进行负染色和挥干,之后将片子放到透射电镜下观察,并使用传统裂解方法得到单细胞溶液同时铺设在铜网上进行对比。通过观察他们发现,使用FluidFM技术得到的细胞提取物能够观察到大泡状的结构、小球状的结构和长丝类的结构,如图2C所示。而相比之下细胞裂解法得到的结果却不尽人意,如图2D所示。
3.2酶活力的检测
酶活力的检测对于探寻细胞异质性有着十分重要的意义。因此作者也对FluidFM提取的细胞提取物进行了对比。首先作者通过β-半乳糖苷酶实验来测定提取蛋白的完整性。通过测定酶解底物产生的荧光素的荧光强度,他们成功观察到荧光强度随时间而增加,说明提取物中的蛋白没有被破坏,如图3C、D所示。随后作者又对不同细胞进行了不同酶活力的检验,结果显示无论是LacZ转基因HeLa细胞上还是在测定HeLa细胞的Caspase3酶上均取得了成功,如图3 E、F所示。
4.3单细胞级转录检测
单细胞层面的基因表达通常需要反转录或者PCR扩增,之后使用qPCR测定。而在此之前往往需要将细胞裂解,而作者他们采用了与传统方法不同的策略。他们首先创建了使用FluidFM直接从活细胞中提取大约0.01 pg RNA,并用普通PCR管合成cDNA并进行qPCR检测,如图4A所示。由于如此小的提取量是不能直接放到PCR管里面的,所以他们采取的策略是首先将提取液注入1uL水中,然后再转移到PCR管中,进行合成和检测如图6B所示。他们检测了两种管家基因beta-actin (ACTB)beta-2-microglobulin (B2M)以及GFP mRNA的表达量。在21个样本中有90%的样本成功检测到至少1中基因的表达,其中2/3的样本可以同时检测到三种基因的表达。而对细胞核的检测中,也能够检测到至少一种基因的表达,如图4C所示。在对比同一细胞同时提取细胞质(1.7 pL)和细胞核(1.3 pL)中这三种基因的表达时,可以发现两者基本相同,如图4D所示。
总结
随着生物研究越发趋于微观化,对于分析单个细胞的需求变得越来越大。但是由于单个细胞体积小,所能够提取出的物质相比以前细胞群落分析来说,难度显著提高。这不仅对检测仪器的灵敏度有了新的更高的要求,也同时对样本本身的质量也提出了更高的指标。本篇中使用FluidFM提取活细胞所取得的样本质量相比于传统裂解手段有了明显的提高,从而取得了令人满意的结果。另外这种方法控制提取量后,甚至能够做到不杀死细胞的情况下完成提取,这使得对于对单个细胞代谢测定的追踪成为了可能。
多功能单细胞显微操作系统--FluidFM BOT,是将原子力系统、微流控系统、细胞培养系统为一体的单细胞操作系统。主要功能包括单细胞注射、单细胞提取以及单细胞分离。
FluidFM BOT极大的方便了单细胞水平的研究,尤其适合应用于精准医疗、单细胞生物学、单细胞质谱、单细胞基因编辑、药物研发等领域。
注射、提取、分选一体化的单细胞操纵解决方案
参考文献:
1. Actis, P., Maalouf, M.M., Kim, H.J., Lohith, A., Vilozny, B., Seger,
R.A., and Pourmand, N. (2014). Compartmental genomics in living cells
revealed by single- cell nanobiopsy. ACS Nano 8, 546–553.
2. Amara, A., and Mercer, J. (2015). Viral apoptotic mimicry. Nat. Rev. Microbiol. 13, 461–469.
3. Bengtsson, M., Sta° hlberg, A., Rorsman, P., and Kubista, M. (2005).
Gene expression profiling in single cells from the pancreatic islets of
Langerhans reveals lognormal distribution of mRNA levels. Genome Res.
15, 1388–1392.
4. Bertrand, R., Solary, E., O’Connor, P., Kohn, K.W., and Pommier, Y.
(1994). Induction of a common pathway of apoptosis by staurosporine.
Exp. Cell Res. 211, 314–321.
5. Cai, X., Evrony, G.D., Lehmann, H.S., Elhosary, P.C., Mehta, B.K.,
Poduri, A., and Walsh, C.A. (2014). Single-cell, genome-wide sequencing
identifies clonal somatic copy-number variation in the human brain. Cell
Rep. 8, 1280–1289.
6. Grindberg, R.V., Yee-Greenbaum, J.L., McConnell, M.J., Novotny, M.,
O’Shaughnessy, A.L., Lambert, G.M., Arau´ zo-Bravo, M.J., Lee, J.,
Fishman, M., Robbins, G.E., et al. (2013). RNA-sequencing from single
nuclei. Proc. Natl. Acad. Sci. USA 110, 19802–19807.
7. Guillaume-Gentil, O., Potthoff, E., Ossola, D., Do¨ rig, P.,
Zambelli, T., and Vorholt, J.A. (2013). Force-controlled fluidic
injection into single cell nuclei. Small 9, 1904–1907.
8. Guillaume-Gentil, O., Potthoff, E., Ossola, D., Franz, C.M.,
Zambelli, T., and Vorholt, J.A. (2014). Force-controlled manipulation of
single cells: from AFM to FluidFM. Trends Biotechnol. 32, 381–388.
9. Hashimshony, T., Wagner, F., Sher, N., and Yanai, I. (2012). CEL-Seq:
singlecell RNA-Seq by multiplexed linear amplification. Cell Rep. 2,
666–673.
10. Jiang, L., Schlesinger, F., Davis, C.A., Zhang, Y., Li, R., Salit,
M., Gingeras, T.R., and Oliver, B. (2011). Synthetic spike-in standards
for RNA-seq experiments. Genome Res. 21, 1543–1551.
11. Kovarik, M.L., and Allbritton, N.L. (2011). Measuring enzyme activity in single cells. Trends Biotechnol. 29, 222–230.
12. Kuipers, M.A., Stasevich, T.J., Sasaki, T., Wilson, K.A., Hazelwood,
K.L., McNally, J.G., Davidson, M.W., and Gilbert, D.M. (2011). Highly
stable loading of Mcm proteins onto chromatin in living cells requires
replication to unload. J. Cell Biol. 192, 29–41.
13. Liebherr, R.B., Hutterer, A., Mickert, M.J., Vogl, F.C., Beutner,
A., Lechner, A., Hummel, H., and Gorris, H.H. (2015). Three-in-one
enzyme assay based on single molecule detection in femtoliter arrays.
Anal. Bioanal. Chem. 407, 7443–7452.
14. Lo, S.J., and Yao, D.J. (2015). Get to understand more from
single-cells: current studies of microfluidic-based techniques for
single-cell analysis. Int. J. Mol. Sci. 16, 16763–16777.
15. Meister, A., Gabi, M., Behr, P., Studer, P., Vo¨ ro¨ s, J.,
Niedermann, P., Bitterli, J., Polesel-Maris, J., Liley, M., Heinzelmann,
H., and Zambelli, T. (2009). FluidFM: combining atomic force microscopy
and nanofluidics in a universal liquid delivery system for single cell
applications and beyond. Nano Lett. 9, 2501–2507.
16. Nagaraj, N., Wisniewski, J.R., Geiger, T., Cox, J., Kircher, M.,
Kelso, J., Pa¨ a¨ bo, S., and Mann, M. (2011). Deep proteome and
transcriptome mapping of a human cancer cell line. Mol. Syst. Biol. 7,
548.
17. Nawarathna, D., Turan, T., and Wickramasinghe, H.K. (2009).
Selective probing of mRNA expression levels within a living cell. Appl.
Phys. Lett. 95, 83117. O’Huallachain, M., Karczewski, K.J., Weissman,
S.M., Urban, A.E., and Snyder, M.P. (2012). Extensive genetic variation
in somatic human tissues. Proc. Natl. Acad. Sci. USA 109, 18018–18023.
18. Osada, T., Uehara, H., Kim, H., and Ikai, A. (2003). mRNA analysis of single living cells. J. Nanobiotechnology 1, 2.
19. Pfeiffer-Guglielmi, B., Dombert, B., Jablonka, S., Hausherr, V., van
Thriel, C., Scho¨ bel, N., and Jansen, R.P. (2014). Axonal and
dendritic localization of mRNAs for glycogen-metabolizing enzymes in
cultured rodent neurons. BMC Neurosci. 15, 70.
20. Picelli, S., Faridani, O.R., Bjo¨ rklund, A.K., Winberg, G.,
Sagasser, S., and Sandberg, R. (2014). Full-length RNA-seq from single
cells using Smartseq2. Nat. Protoc. 9, 171–181.
21. Raj, A., Peskin, C.S., Tranchina, D., Vargas, D.Y., and Tyagi, S.
(2006). Stochastic mRNA synthesis in mammalian cells. PLoS Biol. 4,
e309.
22. Ramsko¨ ld, D., Luo, S., Wang, Y.C., Li, R., Deng, Q., Faridani,
O.R., Daniels, G.A., Khrebtukova, I., Loring, J.F., Laurent, L.C., et
al. (2012). Full-length mRNA-Seq from single-cell levels of RNA and
individual circulating tumor cells. Nat. Biotechnol. 30, 777–782.
23. Rissin, D.M., Kan, C.W., Campbell, T.G., Howes, S.C., Fournier,
D.R., Song, L., Piech, T., Patel, P.P., Chang, L., Rivnak, A.J., et al.
(2010). Single-molecule enzyme-linked immunosorbent assay detects serum
proteins at subfemtomolar concentrations. Nat. Biotechnol. 28, 595–599.
24. Rondelez, Y., Tresset, G., Tabata, K.V., Arata, H., Fujita, H.,
Takeuchi, S., and Noji, H. (2005). Microfabricated arrays of femtoliter
chambers allow single molecule enzymology. Nat. Biotechnol. 23, 361–365.
25. Saha-Shah, A., Weber, A.E., Karty, J.A., Ray, S.J., Hieftje, G.M.,
and Baker, L.A. (2015). Nanopipettes: probes for local sample analysis.
Chem. Sci. 6, 3334–3341.
26. Sarkar, A., Kolitz, S., Lauffenburger, D.A., and Han, J. (2014).
Microfluidic probe for single-cell analysis in adherent tissue culture.
Nat. Commun. 5, 3421.
27. Schmid, A., Kortmann, H., Dittrich, P.S., and Blank, L.M. (2010).
Chemical and biological single cell analysis. Curr. Opin. Biotechnol.
21, 12–20.
28. Tang, F., Barbacioru, C., Nordman, E., Li, B., Xu, N., Bashkirov,
V.I., Lao, K., and Surani, M.A. (2010). RNA-Seq analysis to capture the
transcriptome landscape of a single cell. Nat. Protoc. 5, 516–535.
29. Taniguchi, K., Kajiyama, T., and Kambara, H. (2009). Quantitative
analysis of gene expression in a single cell by qPCR. Nat. Methods 6,
503–506.
30. Van Gelder, R.N., von Zastrow, M.E., Yool, A., Dement, W.C.,
Barchas, J.D., and Eberwine, J.H. (1990). Amplified RNA synthesized from
limited quantities of heterogeneous cDNA. Proc. Natl. Acad. Sci. USA
87, 1663–1667.
31. Veyer, D.L., Maluquer de Motes, C., Sumner, R.P., Ludwig, L.,
Johnson, B.F., and Smith, G.L. (2014). Analysis of the anti-apoptotic
activity of four vaccinia virus proteins demonstrates that B13 is the
most potent inhibitor in isolation and during viral infection. J. Gen.
Virol. 95, 2757–2768.
32. Wachsmuth, M., Weidemann, T., Mu¨ ller, G., Hoffmann-Rohrer, U.W.,
Knoch, T.A., Waldeck, W., and Langowski, J. (2003). Analyzing
intracellular binding and diffusion with continuous fluorescence
photobleaching. Biophys. J. 84, 3353–3363.
33. Wang, D., and Bodovitz, S. (2010). Single cell analysis: the new frontier in ‘omics’. Trends Biotechnol. 28, 281–290.
34. Wasilenko, S.T., Stewart, T.L., Meyers, A.F., and Barry, M. (2003).
Vaccinia virus encodes a previously uncharacterized
mitochondrial-associated inhibitor of apoptosis. Proc. Natl. Acad. Sci.
USA 100, 14345–14350.
35. Weis, K. (2003). Regulating access to the genome: nucleocytoplasmic transport throughout the cell cycle. Cell 112, 441–451.
36. Wu, M., and Singh, A.K. (2012). Single-cell protein analysis. Curr. Opin. Biotechnol.23, 83–88.
37. Zhao, L., Kroenke, C.D., Song, J., Piwnica-Worms, D., Ackerman,
J.J., and Neil, J.J. (2008). Intracellular water-specific MR of
microbead-adherent cells: the HeLa cell intracellular water exchange
lifetime. NMR Biomed. 21, 159–164.