DNA甲基化预测
实验概要
本实验分别对DNA片段、基因、启动子和外显子进行了甲基化的计算预测,并且随机选择了1000甲基化的和1000未甲基化的个体进行预测。用于甲基化预测的特征有:GC相关特征、四联体频率、转录因子结合位点(TFBSs)。所有预测方法均采用Weka提供的软件进行。
实验步骤
1. DNA甲基化数据
本研究基于在拟南芥中使用Tiling芯片第一次进行全基因组水平的DNA甲基化分析的实验结果。实验数据包括26,852个具有显著DNA甲基化水平的DNA片段,这些片段覆盖了22,554,840 bp并且代表了完全测序的拟南芥核基因组的~18.9%。拟南芥25,423表达基因中,作者将它们分为三部分:内部甲基化基因(33.3%),启动子甲基化基因(5.2%)和未甲基化基因(61.5%)。利用这些数据,我们确定了所有拟南芥基因的启动子和外显子的甲基化状态。我们分别对DNA片段、基因、启动子和外显子进行了甲基化的计算预测,并且随机选择了1,000甲基化的和1,000未甲基化的个体进行预测。
2. 用于甲基化预测的特征
1) GC相关特征
CpG岛的传统定义需要三个参数:序列一长度,GC含量和CpG ratio。我们利用Wilcoxon秩检验比较了甲基化和未甲基化的DNA片段的GC含量和CpG ratio值,并且发现这两个参数在甲基化和未甲基化的DNA片段间具有非常显著的差异。所以这两个参数被用作预测的两个特征。
在植物和动物中,甲基化主要在CG sequence context中发现。在进化过程中双核营酸TpG(以及它的反义互补CpA)的分布推测起来也和mCpGs的去甲基化有关。我们分别计数了甲基化和未甲基化片段中的双核普酸CpG和TpG,并且利用Wilcoxon秩检验比较它们在两类片段间的分布。显著差异意味着CpG和TpG的分布与DNA甲基化间的联系。我们采用了每1 kb中的CpG和TpG的数目作为预测的另外两个特征。
2) 四联体频率
人类淋巴细胞CpG岛甲基化的研究显示CpG岛甲基化与DNA序列中的四联体是高度相关的。所以我们直接从DNA序列计数每个四联体的频率,并且发现256个四联体中的153个在甲基化和未甲基化片段间具有显著差异。所以四联体频率被作为预测的特征。
3) 转录因子结合位点(TFBSs)
甲基可能破坏转录因子的结合位点并导致转录的失败,所以DNA甲基化能够抑制转录。这意味着在TFBSs和DNA甲基化间存在着联系。而且以前的研究己经显示在人类大脑中TFBSs与CpG岛的甲基化是相关的。所以我们预测某些TFBSs的分布可能在拟南芥的甲基化和未甲基化片段间存在差异。我们从PlantCARE,PLACE和AGRIS上得到了拟南芥的105个己知的TFBSs的Position weight matrices (PWM)。这些TFBSs被用于pattern searchprogram MotifScanner来扫描和确定推定的TFBSs的位置,采用默认的标准:最大启动子大小3kb并且没有启动子序列和上游基因有重叠。我们在拟南芥基因组中得到了%个TFBSs类。然后我们利用Wilcoxon rank-sum test比较了甲基化和未甲基化片段间的96个TFBSs的存在与否并且发现66个TFBSs具有显著差异。所以我们把TFBSs的存在与否作为甲基化预测的特征。
3. 预测方法
我们用了多种方法来测验预测性能,这些方法包括alternating decision tree (ADTree),Bayes network (BayesNet),C4.5 decision tree (C4.STree),simple decision table majority classifier (DecisionTable),logistic model trees (LMT),multinomial logistic regression model with a ridge estimator (Logistic),decision tree with naive Bayes classifiers at the Leaves (NBTree),rules from partial decision trees built using C4.5 decision tree (PART),forest of random trees (RandomForest),normalized Gaussian radial basisbasis function network (RBFNetwork),linear logistic regression models (SimpleLogstic),support vector machine (SVM) and voted perceptron algorithm (VotedPerceptron)。这里使用的所有预测方法都是Waikato environment for knowledge analysis (Weka)提供的,Weka是一个可以提供执行大量机器学习和统计算法的环境的Java软件包。所有这些方法的数据被准备成属性相关文件格式(ARFF),这种格式由所有个体和相应个体的属性值(逗号分隔)的列表构成。
4. 性能评估
我们把所有的样本随机分成一个训练集(66%)和一个测试集。预测方法在训练集上进行训练然后在测试集上进行评估。我们使用特异性(SP),敏感性((SE)、精确性(ACC)和相关系数((CC)来评估预测方法的性能。将未甲基化的样本作为阳性类别,甲基化的样本作为阴性类别,我们使用下面的公式计算SP,SE,ACC和CC:
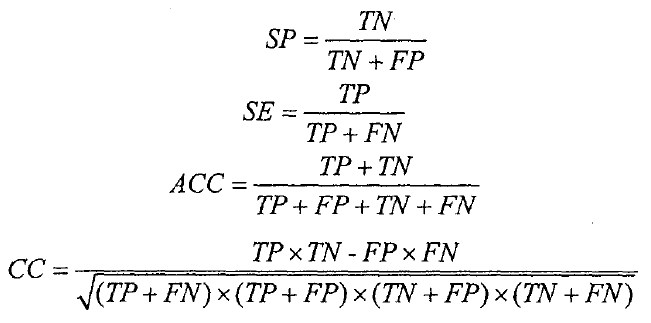
TP,TN,FP和FN分别代表真阳性、真阴性、假阳性和假阴性。这些参数比只使用正确预测的总百分率能够提供一个对分类性能的更精确的评估。
附 件 (共1个附件,占34KB)
![]()
1.jpg
34KB
查看