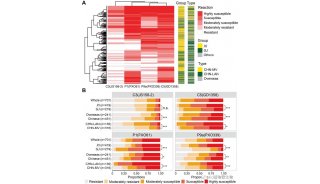
水稻RGA基因组规模的鉴定与分析
实验概要
本实验对粳稻蛋白质数据库进行BLASTP,获得所有粳稻的RGA序列,对RGA进行分类,分别统计了各类RGA在不同染色体上的分布情况,对两个亚种间RGA多态性进行了鉴定和开发。
实验步骤
1. 水稻RGA的搜索
搜集已报道的45个R基因,然后用它们对粳稻蛋白质数据库进行BLASTP (参数取E<10-10,最小长度为该基因的80%),获得所有粳稻的RGA序列。去除粳稻数据库中存在的重复克隆,建立一个粳稻RGA蛋白数据库。同时,根据每个BLASTP搜索中匹配最好的结果,得到这些粳稻RGA的核普酸序列。此外,去除粳稻数据库中存在由于的克隆重复造成的R基因的重复。
2. 水稻RGA的结构分类及其在染色体上的分布
利用Hmmer程序中的hmmsearch部分,采用前面建立的功能域列表,在新建立的数据库中搜索基因序列所包含的功能域。利用pepcoil程序分析序列中CC结构的可能性,数值大于90%的认为具备该结构。运用TM-HMMer分析TM转膜功能域。依据功能域分布的情况,对RGA进行分类,分别统计各类RGA在不同染色体上的分布情况。
3. 两个亚种间RGA多态性的鉴定和开发
将所有粳稻的RGA序列分别与粕稻基因组数据库进行TBLASTN联配,以确定釉稻中对应的等位基因。为消除非等位联配,在TBLASTN搜索中采用了严格的判别标准,将E值设为10-20。对初筛到的粕、粳稻RGA等位基因,进一步用sim4程序进行联配分析,去除匹配率≤85%且两条序列同时间断200bp以上的结果。接着运用diffseq程序分析SNP和InDel在RGA的基因组序列中的分布情况,将存在InDel的作为候选的RGA标记。以粳稻的基因组序列为模板,在INDEL位置的两侧各取100bp的序列,利用ePrimer3程序在取得的序列的两端进行引物设计。ePrimer3程序一般给出五对候选引物,我们取其设计最合理的一条(考虑到目标片段最大长度不大于1000bp),并进行电子PCR(e-PCR)验证。对得到的水稻RGA标记进行命名,规则为以OSR开头,后跟4个数字,例如:OSR0255。上述步骤主要通过编写pert脚本程序来实现。
-
焦点事件

-
焦点事件

-
科技前沿

-
科技前沿

-
科技前沿

-
科技前沿
-
焦点事件

-
焦点事件

-
焦点事件