杨辉组/高彩霞组发现单碱基编辑系统存在严重脱靶效应
2016年,David Liu团队在 Nature 期刊上首次报道了基于胞嘧啶脱氨酶APOBEC1(能催化C脱氨基变成U,而U在DNA复制过程中会被识别成T)和尿嘧啶糖基化酶抑制剂UGI(能防止尿嘧啶糖基化酶将U糖基化引起碱基切除修复)的单碱基编辑工具(BE3)首次实现可以在不引入DNA双链断裂的情况下对单碱基进行替换【1】,2017年该团队在Nature上发表论文报道了另一种单碱基基因编辑工具——腺嘌呤碱基编辑器 (ABE)【2】,它可以将A•T碱基对转换成G•C碱基对,加之2016报道的将G•C碱基对转换成T•A 碱基对的成果,从此研究人员首次实现了不依赖于DNA断裂而能够将DNA四种碱基A、T、G、C进行替换的单碱基基因编辑技术(目前上述两篇Nature论文总被引次数已超千次)。
单碱基编辑器的开发,为定向编辑和修正基因组中的关键核苷酸变异提供了重要工具,由于该技术不引入DNA双链断裂,因此展现了其在遗传疾病治疗与动植物新品种培育等方面潜在的重大应用价值。然而值得注意的是,CRISPR/Cas9从问世以来,其脱靶风险一直备受关注,如果将CRISPR/Cas9及其衍生工具贸然用于临床的话,脱靶效应可能会引起包括癌症在内的很多种副作用。尽管此前人们推出过多种检测脱靶的方案,然而由于过去的方法或者依赖于计算机软件预测,或者依赖于高通量测序检测DSB产生,还有体外检测的方法。这些方法都存在一些局限性,不能高灵敏性检测到脱靶突变,尤其是单核苷酸突变。因此关于CRISPR/Cas9及其衍生工具的真实脱靶率一直存在争议。那么一种能够突破之前限制的脱靶检测技术,将会成为CRISPR/Cas9及其衍生工具是否能最终走上临床的关键。人们迫切希望可以找到一种既能够不依赖于脱靶位点预测,又能有足够信噪比的精密脱靶检测手段。
北京时间2019年3月1日凌晨,Science杂志“背靠背”在线发表了两篇来自中国科学家完成的研究论文,分别用不同的方法在不同的物种上首次发现了单碱基编辑系统存在严重的脱靶效应,这给当前如火如荼开展的相关临床试验蒙上了一层阴影。
题为Cytosine base editor generates substantial off-target single nucleotide variants in mouse embryos的研究论文由中科院神经科学研究所(中国科学院脑科学与智能技术卓越创新中心)、上海脑科学与类脑研究中心杨辉研究组与中科院上海营养与健康研究所隶属的计算生物学研究所(中国科学院-马普学会计算生物学伙伴研究所)李亦学研究组、斯坦福大学遗传学系Lars M. Steinmetz研究组以及中国农业科学院深圳农业基因组研究所的研究人员合作完成。

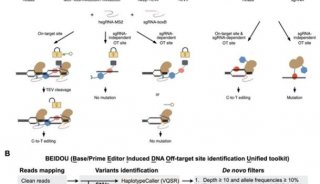
该研究探索了全基因组范围内基因编辑技术可能造成的脱靶效应,建立了一种被命名为GOTI(Genome-wide Off-target analysis by Two-cell embryo Injection)的新型脱靶检测技术,并使用该技术发现: 近年来兴起的单碱基编辑技术有可能导致大量无法预测的脱靶,因而存在严重的安全风险。此研究显著提高了基因编辑技术脱靶检测的敏感性,并且可以在不借助于任何脱靶位点预测技术的情况下发现之前的脱靶检测手段无法发现的完全随机的脱靶位点,为基因编辑工具的安全性评估带来了突破性的新工具,有望成为新的行业检测标准。
如果要提升检测脱靶效应的精度,就必须彻底颠覆原有的脱靶检测手段。首先,为了实现不借助于脱靶位点预测,这就要求必须找到非常严格的对照组来确定基因突变的位点;同时为了检测不依赖于sgRNA的随机突变,最好使用基于单细胞全基因组测序。为了实现以上目标,杨辉研究组与合作者建立了一种名叫“GOTI”的脱靶检测技术。研究者们在小鼠受精卵分裂到二细胞期的时候,编辑一个卵裂球,并使用红色荧光蛋白将其标记。小鼠胚胎发育到14.5天时,将整个小鼠胚胎消化成为单细胞,利用流式细胞分选技术基于红色荧光蛋白,分选出基因编辑细胞和没有基因编辑细胞,再进行全基因组测序比较两组差异(下图),这样就避免了单细胞体外扩增带来的噪音问题。而且由于实验组和对照组来自同一枚受精卵,理论上基因背景完全一致,因此直接比对两组细胞的基因组,其中的差异基本就可以认为是基因编辑工具造成的。

图片来自Science论文
在杨辉实验室全体成员与合作单位的共同努力下,GOTI系统被成功建立了起来。借助于这个系统,团队成员先是检测了最经典的CRISPR/Cas9系统。结果发现,设计良好的CRISPR/Cas9并没有明显的脱靶效应,这个结果结束了之前对于CRISPR/Cas9脱靶率的争议。然而团队还检测了另一个同样被给予厚望的CRISPR/Cas9衍生技术单碱基编辑系统BE3和ABE,这个系统可以精确引入点突变,在之前的研究中从未发现过有明显的脱靶问题。在GOTI的检测下发现,ABE系统同样表现出非常高的特异性,然而BE3存在非常严重的脱靶,而且这些脱靶大多出现在传统脱靶预测认为不太可能出现脱靶的位点,因此之前方法一直没有发现其脱靶问题。
在BE3编辑的胚胎中,平均有283个 SNVs被检测到,相比于Cre或CRISPR/Cas9处理组增加了20倍以上。而这些突变90%以上都呈现出G到A或者C到T的模式,进一步表明这些突变不是胚胎发育过程中随机产生的突变,而是由于BE3编辑所导致。这些突变随机分布于基因组中,有许多会影响蛋白的编码,甚至有一些位于原癌基因(Pin1)和抑癌基因(Rsu1, Stk4, Tsc1, Park7, Rbpj, Ing3, Hipk2, Phlpp2, Yeasts4, Ptch1, Stk3, Smchd1)上的突变,这意味着这些脱靶突变可能会进而引起癌症的发生,具有潜在风险,也就是说BE3系统不适合在临床上使用。
总的来说,上述重要发现,证实了以BE3为代表的部分基因编辑技术存在无法预测的脱靶风险,让世人重新审视了这些新兴技术的风险。更重要的是,此工作建立了一种在精度、广度和准确性上远超越之前的基因编辑脱靶检测技术,有望由此开发精度更高、安全性更大的新一代基因编辑工具,建立行业的新标准。
值得一提的是,上述工作曾在“基因编辑婴儿事件中”发生后的第二天就上传到生命科学预印本杂志BioRxiv上

据悉,该工作从实验设计到全部完成历时一年半左右,中科院神经所博士后左二伟(现任中国农业科学院深圳农业基因组研究所研究员)、计算生物学所博士研究生孙怡迪和魏武研究员、中国农业科学院深圳农业基因组研究所助理研究员袁堂龙为本文共同第一作者,杨辉研究员、李亦学研究员以及斯坦福大学Lars M. Steinmetz教授为本文共同通讯作者。
题为Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice的研究论文由中科院遗传与发育研究所高彩霞研究组完成。该研究在水稻中对BE3 (基于融合rAPOBEC1胞嘧啶脱氨酶的CBE系统)、HF1 -BE3(高保真版本BE3)与ABE单碱基编辑系统的特异性进行了全基因组水平评估,首次在体内利用全基因组测序技术全面分析和比较了这三种单碱基编辑系统在基因组水平上的脱靶效应。

该研究对经过不同单碱基编辑系统转化的的56棵T0代水稻植株与21株对照植株进行全基因组测序。进一步序列统计分析发现,经过单碱基编辑系统处理后,基因组内的插入或删除(indels)突变的数量与对照组相比没有显著变化,但是BE3与HF1-BE3,无论是在有无sgRNA的情况下,均可在水稻基因组中造成大量的单核苷酸变异(SNVs),且大部分为C>T类型的碱基突变。与经过农杆菌转化但不含任何碱基编辑系统的对照植株相比,BE3系统和HF1-BE3系统处理的植株在基因组范围内的C>T SNVs分别增加了94.5%和231.9%,额外产生了98个C>T SNVs和242个C>T SNVs。重要的是,与Cas-OFFinder软件预测结果比较发现,这些额外增加的C>T SNVs绝大多数发生在不是现有软件可以预测到的脱靶位点。此外,这些C>T变异在染色体间均匀分布,但呈现出在转录活跃区富集的趋势。这些区域倾向于释放单链DNA为胞嘧啶脱氨酶提供合适的底物。研究还发现,与CBE系统相反,ABE系统表现出非常高的特异性。ABE处理的植株与对照植株在全基因组范围内的SNV数量基本一致。
总的来说,该研究表明现有BE3和HF1-BE3系统,而非ABE系统,可在植物体内造成难以预测的脱靶突变,因此,需要进一步优化提高特异性。该工作创新性地利用相似遗传背景的克隆植物及全基因组重测序解决了以前大量异质细胞序列分析的复杂性。
据悉,高彩霞研究组博士生靳帅、宗媛以及梁承志研究组工作人员高强为本文的共同第一作者,高彩霞研究员为本文的通讯作者。
原文链接:
http://science.sciencemag.org/content/early/2019/02/27/science.aav9973
http://science.sciencemag.org/content/early/2019/02/27/science.aaw7166
参考文献
1、 A. C. Komor, Y. B. Kim, M. S. Packer, J. A. Zuris, D. R. Liu, Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420-424 (2016).
2、Gaudelli, N. M., Komor, A. C., Rees, H. A., Packer, M. S., Badran, A. H., Bryson, D. I., & Liu, D. R. (2017). Programmable base editing of A• T to G• C in genomic DNA without DNA cleavage. Nature, 551(7681), 464.
-
仪器推荐

-
仪器推荐
-
仪器推荐
 询底价 Tel:400-6699-117 转 6746
询底价 Tel:400-6699-117 转 6746 -
仪器推荐

-
仪器推荐

-
焦点事件

-
焦点事件

-
科技前沿
-
项目成果
-
项目成果
-
项目成果
-
科技前沿