DeepDEP:深度学习构建肿瘤依赖性图谱
大家好呀!今天给大家介绍一篇2021年发表在Science Advances上的文章。全基因组功能缺失筛查揭示了对癌细胞增殖十分重要的基因,称为肿瘤依赖性。然而将肿瘤依赖性关系与癌细胞的分子组成联系起来并进一步与肿瘤联系起来还是一个巨大的挑战。本研究,作者提出了DeepDEP,基于深度学习模型和基因组数据可以预测癌症依赖性。该模型对无标签的肿瘤基因组进行无监督预训练来改进癌症依赖性学习。作者使用三个独立数据集验证DeepDEP的性能。通过系统的模型解释,作者扩展了当前的癌症依赖性图谱。将DeepDEP应用于泛癌的肿瘤基因组数据并首次构建了具有临床相关性的泛癌依赖性图谱。总的来说,DeepDEP作为一种新的工具可以用于研究癌症依赖性。
Predicting and characterizing a cancer dependency map of tumors with deep learning
使用深度学习预测和描述肿瘤异型性图谱
1.模型设计
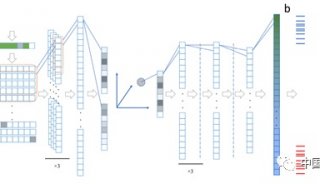
基因依赖性或必要性是指基因对细胞增殖和生存的重要程度。癌细胞中的基因依赖性与遗传环境有关。本研究,作者提出了DeepDEP,通过学习肿瘤和细胞系样本的高维基因组数据来预测基因依赖性。DeepDEP使用转移学习对无标记的肿瘤样本进行无监督的预训练,随后对有标记的CCL样本进行参数微调,用于捕获基因组学和基因依赖之间的关系(图1A)。该模型包括以下几个部分:1.对DNA突变,基因表达,DNA甲基化和CAN数据进行降维编码神经网络;2.构建用于提取DepOI的编码网络;3.构建预测网络用于将学习到的特征转化为依赖性打分(图1B)。使用自编码器对8238个TCGA肿瘤样本的每种组学数据进行降维,将构建的自编码器的结构和参数转移到DeepDEP的四个相应编码器上,随后对整个网络进行训练生成一个基因依赖的特征空间。

原始依赖性打分是CERES以接近与零的模式估计和校正的基因效应打分图2A)。通过SGRNA,筛选复制和CCL质量控制计算打分,其负值越大,依赖性越强即重要性越强。作者基于高度可选依赖性或COSMIC选择1298个癌症中候选DepOIs,每个DepOI涉及33.2个分子特征(图2B)。共获得360844(278CCL*1298DepOIs)个标记样本。将CCLs随机分为训练集/验证集(90%)和测试集(10%)(图2C)。训练集和测试集之间没有肿瘤大小和细胞类型等的显著差异。
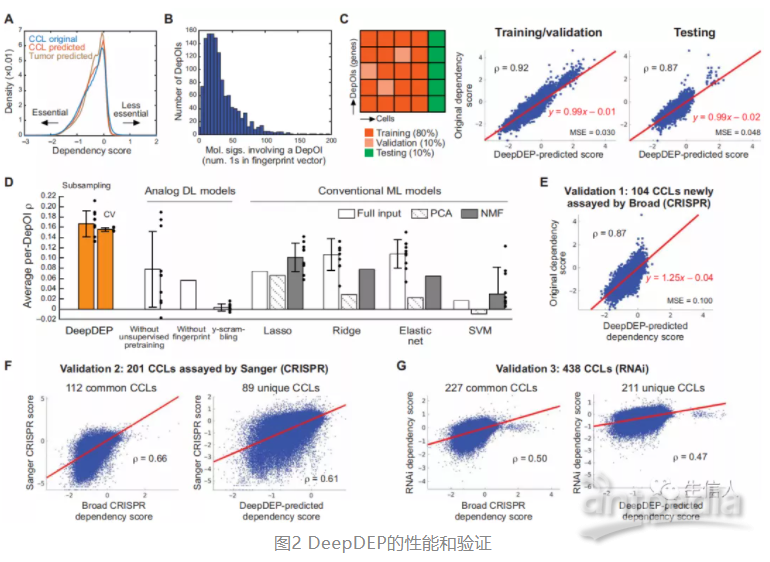
2.模型性能和与其他方法比较
测试集的预测准确率较高(图2C),性能稳定。总的来说,作者的模型在1298个DepOIs中的平均DepOIs为0.18(图2D)。随后,作者重点关注两个高度变异的DepOIs亚集。这些DepOIs更可能是癌症相关基因,例如TP53是高度变量依赖性的。这两个亚集的平均DepOI分别为0.34和0.28,TP53的DepOIs为0.62。随后,作者将DeepDEP与六种ML方法进行比较。与DeepDEP相比,所有ML方法在10轮抽样中均会显著降低DepOI的相关系数(图2D)。
3.模型验证
为避免模型的过拟合,作者使用下采样和10折交叉验证的方法验证模型性能。作者使用三个独立数据集验证模型,DeepDEP的预测打分与实际打分一致(图2E),尽管对这些数据使用了不同的筛选机制和算法,作者鉴定到了一些共有的CCLs(图2F和2G)。
4.使用基因表达数据描述基因依赖性的模型解释
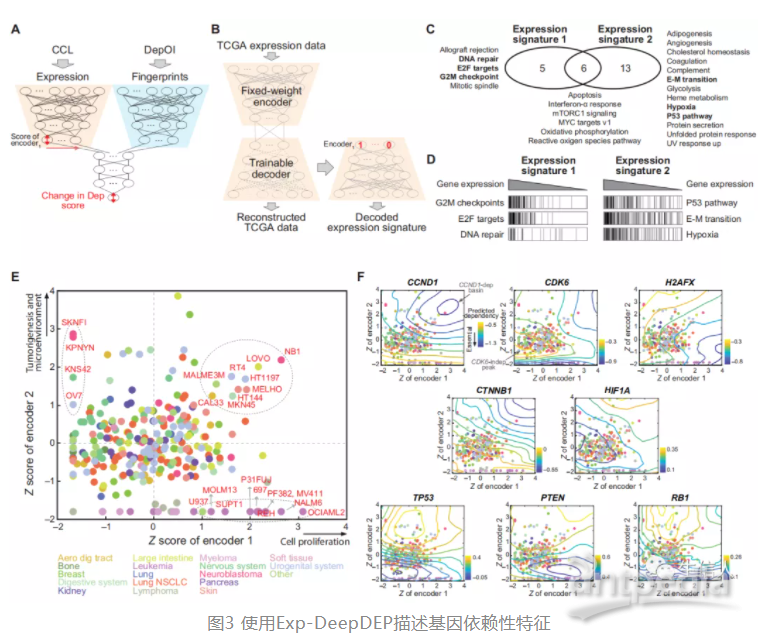
作者构建了仅使用一种或两种组学数据的简化模型,例如Mut-DeepDEP使用突变数据,Mut/Exp-DeepDEP使用突变数据和表达数据。Mut/Exp-DeepDEP和Exp-DeepDEP的性能与DeepDEP的性能相当,但仅使用突变或CNA数据的模型性能较差。作者研究Exp-DeepDEP来了解单一组学模型学习的信息,即依赖性和基因表达之间的关系。其编码层的输出有50个神经元,在使用CCL依赖性数据进行最终训练后仅有两个神经元为非零值。作者人为干预这两个神经元并检查预测依赖性打分的变化(图3A)。通过解码层重构6016个基因的表达水平,从而解码这两个神经元的表达特征(图3B)并进行GSEA分析。特征1与细胞增殖相关通路正相关(图3C和3D),特征2与肿瘤发生和肿瘤微环境等过程有关。作者通过编码器映射了278个CCLs,将每个CCL转化为特征打分。作者鉴定到24个CCL中有20个在特征2中活性最低而在特征1中高度变化(图3E)。作者通过组合不同的特征打分预测基因依赖性并使用等高线图进行可视化(图3F)。
5.使用Mut-DeepDEP研究SE进行模型解释
作者使用Mut-DeepDEP预测与CCL中与诱导或移除基因突变有关的基因依赖性变化,或突变和基因敲除之间的SE。简单地说,作者一次干扰一个突变,0变1表示为野生型变为突变体(图4A)。对CCL中的突变DepOI对,通过比较突变和未突变的DepOI的预测依赖性计算SE打分。SE打分负值表明CCL中的突变越重要。总的来说,SE打分是适中的(图4B),独立于细胞系(图4C)。作者分析了2个最常见的SE对,PTEN突变/CHD1删失和BRCA1/PARP1。CCLs中PTEN/CHD1的评价SE显著低于CHD1与其他基因突变的平均SE打分(图4D),BRCA1/PARP1的结果类似。作者研究与KRAS突变有关的SE基因,结果表明EGFR和KRAS之间存在SE相互作用(图4E)。
6.预测肿瘤依赖性
肿瘤和CCL之间的整体依赖性特征的相关性与CCL之间的相关性类似(图5A)。肿瘤之间的依赖性变化是适中的,细胞周期调节因子CDND1和CDK6是选择性最高的基因之一。
7.预测肿瘤依赖性与基因组学的相关性
上述结果与基因组表达和甲基化数据中癌症类型的特异性形成对比(图5B)。肿瘤的依赖性与突变负担较高,CNA和基因表达和甲基化水平升高有关(图5C)。接下来,作者研究基因组在基因依赖性中的作用,作者鉴定到1.6M的M-Dep,2.1M的E-Dep,1.3M的Me-Dep和1.7M的C-Dep事件(图5D)。对于每个DepOI,作者计算这四类事件的百分比,大多数DepOI为E-Dep,M-Dep和C-Dep事件(图5E)。
8.利用临床和临床前数据验证肿瘤依赖性
作者首先研究乳腺癌,其临床数据来自TCGA数据库。预测的ER+对ESR1有更强的依赖性(图6A)。作者还评价了靶向分子治疗的药物反应数据,曲妥珠单抗对ERBB2的依赖性较强(图6B)。与22个进展性疾病的PDXs相比,预测达到CR的PDXs对FGFR2/FGFR4的依赖性较高(图6C)。在TCGA的微卫星不稳定性(MSI)易发癌中,预测MSI高的肿瘤对WRN的依赖性较强(图6D)。
9.预测肿瘤依赖性与化疗耐药和生存相关性
作者分析BRCA与化疗耐药相关的依赖性,比较化疗后达到CR和PD的患者之间的预测相关性。共有71个基因在两组中存在显著差异(图7A),大多数依赖性与化疗耐药性正相关,依赖性打分越负,化疗反应越差。NDUFS5的依赖性最大(图7B)。化疗反应相关DepOIs显著富集在线粒体和OXPHOS的GO terms上(图7C)。随后,作者分析不同谱系癌症患者的基因依赖性和OS的关系,有34例DepOIs可用于8种癌症的预后(图7D和7E)。例如IL2的依赖性打分越负,在7种癌症中的OS较好在2种癌症中的OS较差(图7F)。作者的研究表明,IL的依赖性越强,UVM(图7G),KICH,KIRP的OS越好。SMAD4是一个肿瘤抑制因子,SMAD4依赖性越强,七种癌症的OS越好(图7H和7I)。作者的研究表明基因依赖性打分在化疗反应和预测预后方面具有重要的临床意义。
本研究基于深度学习和基因组多组学数据构建DeepDEP,可以预测癌症依赖性,构建癌症依赖性图谱。作者使用三个额外数据验证DeepDEP的性能。此外,作者全面分析了基因依赖性与基因组学和临床特征的相关性。