Nat. Methods | 色谱-质谱联用助力蛋白质相互作用网络解析

前言
蛋白质复合体是细胞中非常重要的大分子装置,如何描述蛋白质复合体各种组分之间相互作用关系一直都是科学家们想要解决的难点问题之一。系统性地解析多蛋白复合物对于了解其在细胞内的功能非常关键。
基本信息
2019年7月15日,来自多伦多大学的Gary D. Bader与波士顿大学的Andrew Emili研究组合作在Nature Methods杂志上发表长文EPIC: software toolkit for elution profile-based inference of protein complexes,搭建了通过色谱-质谱联用(Chromatographic fractionation–mass spectrometry,CF–MS)的方式解析蛋白质相互作用网络的软件包,并且免费向所有人开放使用(https://hub.docker.com/r/baderlab/bio-epic/)。

结果及分析
1.色谱-质谱联用分析
亲和纯化质谱分析(Affinity purification–mass spectrometry,AP-MS)是鉴定蛋白质复合物广泛使用的有力工具之一【1】。但是亲和纯化质谱分析的问题是很难大规模地分析蛋白质复合物中组分的相互作用关系,另外也很难用于非模式生物之中。而色谱-质谱联用分析方式对于大规模分析大分子更加适用【2,3】。色谱-质谱联用的方法是从细胞提取混合物(例如组织裂解物)将稳定结合的蛋白复合物根据其生物物理特性共纯化。

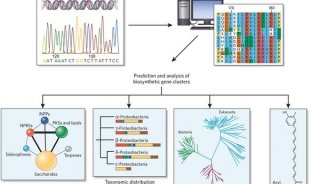
图1 | EPIC中色谱-质谱实验准备过程
2.建立相互作用网络
根据EPIC检测蛋白-蛋白相互作用并建立相互作用网络的步骤最基本的是基于色谱-质谱联用在非变形条件下对天然大分子混合物的广泛实验分离。离子交换高效液相色谱在解析复合物成分等方面非常有效(图1)。为了提高实验效率可以添加非离子型洗涤剂以溶解疏水性络合物,使用化学交联剂可以用于提高不稳定成分的稳定性。色谱-质谱的实验主要包括三个步骤:生物化学分馏、质谱分析以及最后得到的蛋白质表达谱分析(图1)。而使用EPIC自动分析来自色谱-质谱联用测得的数据主要通过以下三个步骤:(1)使用相关性分析计算共洗脱蛋白质图谱相似性;(2)使用现有的数据库中实验以及功能性证据训练机器学习,并对复合体中蛋白-蛋白相互作用进行计算;(3)对可能的蛋白质复合物进行预测、分类以及标准化(图2)。

图2 | EPIC数据分析过程
根据已有的多个蛋白质数据库对EPIC进行优化之后,考虑到“实践是检验真理的唯一标准”,作者们将在线虫中获得的16组共洗脱得到的蛋白质图谱导入到EPIC,EPIC成功预测了3855个线虫蛋白质(约占线虫蛋白质组的25%)中16,098组高度可信的蛋白质相互作用(图3)。并且其中13,547组蛋白质相互作用是完全新颖的相互作用关系。为以后研究全蛋白质组中的相互作用提供了新颖的方法和角度。

图3 | EPIC分析线虫蛋白质相互作用网络
总结
目前我们对于细胞和组织中的蛋白质相互作用了解的范围还极小极有限,对于非模式动物更是知之甚少。色谱-质谱联用是解决这一问题的理想实验技术,可以用于任何来源的生物样品。但是色谱-质谱联用分析方式所得到的数据复杂且难以处理。因此,Bader与Emili研究组开发的EPIC软件对于不同生物来源的天然大分子组装的分析有极大的促进作用,操作界面简单并且易于在不同的系统中使用,同时该软件也是开源的,让软件能够与大家的实验共同进步。
好的研究真正重要的是技术共享、知识共享,而不是固步自封、抱紧自己的一亩三分地。越来越多的科学家正将最新的研究成果发表在预刊印网站供大家开放讨论,将实验操作视频上传到网络供大家参考,将技术涉及的详细代码与大家分享。永远对热烈的思想碰撞、积极的知识分享、不分国界与地界的全开放的科研环境抱有憧憬,相信科学研究的前途每一天都会变得更好。
原文链接:https://doi.org/10.1038/s41592-019-0461-4
参考文献
1. Rigaut, G. et al. A generic protein purificationmethod for protein complex characterization and proteome exploration. Nature biotechnology 17, 1030-1032, doi:10.1038/13732(1999).
2. Havugimana,P. C. et al. A census of humansoluble protein complexes. Cell 150, 1068-1081,doi:10.1016/j.cell.2012.08.011 (2012).
3. Wan,C. et al. Panorama of ancientmetazoan macromolecular complexes. Nature525, 339-344,doi:10.1038/nature14877 (2015).

鹿明生物 · 易明学院第32期多组学技术理论与蛋白生信实践培训开班在即...
心动不如行动,名额不多啦,赶紧报名
扫描二维码 报名抢座吧
举办地点:中国 北京
(北京市石景山区石景山路23号)
培训时间:2019年8月22-23日
主办单位:上海鹿明生物 · 易明学院

END
(转载自BioArt) 
-
仪器推荐

-
仪器推荐

-
仪器推荐
 询底价 Tel:400-6699-117 转 7875
询底价 Tel:400-6699-117 转 7875 -
仪器推荐

-
仪器推荐

-
厂商文章

-
厂商文章
-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章
-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章

-
厂商文章

















