转录组+代谢组专题 | 转录组+代谢组到底分析什么?

转录组+代谢组到底分析什么?
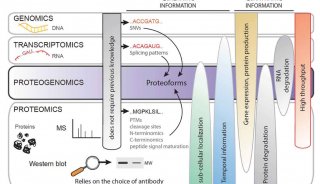
基因组,转录组,蛋白质组,代谢组等单一组学分析方法为动植物生长发育过程提供丰富的数据信息。但是生命系统是多组分互相联系的有机体,是高度复杂的庞大系统,单一组学的研究只能识别单一的变化,不能发现导致变化的原因,也无法研究复杂生物过程的相互联系。需要从多层次和多因素相互作用的全局性角度进行整合研究,才能完整地认识和揭示生命的复杂生理活动。

不同层次的组学数据联合分析,一方面可以相互验证,另一方面为我们提供了一个全景式了解生物活动过程的窗口。通过建立不同层次分子间数据关系,同时结合功能分析、代谢通路富集等,系统全面地解析生物分子功能和调控机制,最终实现对生物变化大趋势与方向的综合了解,提出分子生物学变化机制模型,并筛选出重点代谢通路或者基因、代谢产物进行后续深入实验分析与应用。
转录组+代谢组,基于代谢组和转录组数据,开展表达基因(mRNA),代谢物积累的相关分析。差异积累的代谢物信息可辅助时序表达的众多基因进行“共表达”分析,构建核心调控网络,找出关键候选基因,阐述生物学现象。那么转录组+代谢组的多组学关联,可以做哪些分析内容?
转录组+代谢组关联分析整体可以分为三部分,基于KEGG通路的kegg注释和富集分析;基于两组学的建模关联分析;基于相关性的关联分析。
一 基于KEGG的关联分析
1. kegg通路注释
KEGG数据库的特点是,将基因和代谢物以图形化的形式显现出来,以便研究者了解生物体内基因与代谢的合成调控过程。对转录组与代谢组的数据进行关联分析时,选择比较组中的差异基因和差异代谢物进行KEGG通路注释。图中框框代表基因,点代表代谢物。上下调信息,以颜色来表征。红色代表基因或者代谢物是上调的,绿色代表基因或者代谢物是下调的。蓝色代表注释到该酶上的基因既有上调又有下调。

KEGG通路注释图
2. kegg富集分析
富集分析是将功能相似的基因集通过统计学检验算法富集到一起,从而方便研究具有某一类功能的基因。通过KEGG注释分析将差异基因进行了分类,但是在每个差异分组中会有多则几十个通路。所以需要通过富集分析的方法,去筛选出更可能被激活的通路。富集分析原理是基于超几何分布。分别对差异基因和差异代谢物进行kegg富集分析,展示共有的,且以转录组pvalue排序,取前25的通路进行条形图展示。不同颜色代表不同组学。纵坐标代表KEGG通路名称。

KEGG富集分析条形图
3. kegg富集气泡图
利用两组学共同富集到的KEGG pathway绘制气泡图,气泡图是一个五维图,即通过横纵坐标、气泡颜色渐变、形状、大小体现不同组学共同富集到的KEGG pathway。其中,横坐标代表该通路在不同组学的富集因子(Diff/Background),纵坐标代表KEGG pathway名称;红-黄-绿色的渐变代表富集的显著程度由高-中-低的变化,用Pvalue表示;气泡的形状代表不同的组学,气泡的大小代表差异代谢物或基因的数目,数目越大,点越大。
.

KEGG富集气泡图
二. 基于相关性的关联分析
利用基因和代谢物在所有样本中的定量值进行相关性分析,相关性方法是使用R中的cor函数计算基因和代谢物的皮尔逊相关性系数,选取相关性系数大于0.80且pvalue小于0.05相关性结果进行后续分析。
1. 九象限图
九象限图可以用来展示每个差异分组里皮尔逊相关系数大于0.80且pvalue小于0.05的物质的差异倍数情况,用黑色虚线,从左至右、从上至下,依次分为 1-9 个象限。横坐标代表基因差异倍数的log2值,小于1代表基因下调,大于1代表基因上调。纵坐标代表代谢物的差异倍数的log2值,小于1代表代谢物下调,大于1代表代谢物上调。每个象限的具体含义,以第一象限为例,代表该象限对应点代表的基因和代谢物,相关性大于0.8,且基因下调,代谢物上调。这一部分基因可能是负调控代谢物的。

相关性九象限图
2. 相关性聚类热图
筛选差异分组的差异基因和差异代谢物的相关性结果绘制相关性聚类热图。图中每行为一个基因,每列为一个代谢物,红色代表基因和代谢物正相关,绿色代表基因和代谢物负相关。

相关性聚类热图
3. 相关性网络图
网络图来表示代谢物和基因之间的相关关系,通常通过相关性计算得出来的相关性结果数量会非常大,如果直接用来绘制相关性网络图,绘制结果会无法查看。所以相关性网络图作为一个展示性分析结果,一般是选择最终要展示的数据进行绘制。

相关性网络图
4. 典型相关分析
典型相关分析(canonical correlation analysis, CCA),是利用综合变量对之间的相关关系来反映两组指标之间的整体相关性的多元统计分析方法。对每个通路中的差异基因及差异代谢物进行CCA分析。图中以十字区分出四个区域,在同一个区域内,距原点越远,相互距离越近,关联性越高。代谢物标注为紫色,基因标注为红色。

CCA图
三. 基于建模的关联分析
1.O2PLS分析
O2PLS 模型用于两个数据组间的整合分析,包括系统生物学组学间关联、分子调控机制-表型间关联等各种大数据组的内在联系都可通过此模型进行整合分析。该模型一方面可反映不同数据组间的整体影响,另一方面可直接体现不同变量在模型中的权重(权重越大,意味着该变量的变化对另一个组学的扰动更剧烈),从而更加精准地发现关键调节现象。O2PLS 为非监督建模,可客观描述两数据组间是否存在关联趋势,尽可能从源头上避免假阳性关联。由于 O2PLS 模型可灵活进行组学数据挖掘,发现不同层面的调节信息,从而有助于建立系统生物学调节网络。
选取所有的差异基因、差异代谢物建立 O2PLS 模型,通过载荷图初步判断不同数据组中相关性和权重都比较高的变量,筛选出影响另一组学的重要变量。图中每个点到原点的距离高度代表基因过着代谢物和另外一个组学相关性的大小,且颜色越深表示相关性越大,图中标示出对另一个组学影响较大的前 10 个基因/代谢物。


基因与代谢物的loadings图
2. PCA分析
PCA是一种无监督模式识别的多元数据统计分析方法,它通过正交变换将一组可能存在相关性的变量转换为一组线性不相关的变量,转换后的这个变量叫主成分。简单理解PCA的数据处理原理就是:将原始数据压缩成n个主成分来描述原始数据集的特征,PC1表示能描述多维数据矩阵中最明显的特征,PC2表示除PC1之外的所能描述数据矩阵中最显著的特征,PC3……PCn以此类推。
这个分析方法常用来研究如何通过少数几个主成分来揭示多个变量间的内部结构,即从原始变量中导出少数几个主成分,使它们尽可能多地保留原始变量的信息,且彼此间互不相关,通常数学上的处理就是将原来多个指标作线性组合,作为新的综合指标。
分别对转录组以及代谢组进行PCA分析,直观展示各样品分组间是否在两组学间存在差异。


转录组与代谢组PCA分析
以上就是转录组和代谢组联合分析的主要内容,由于生物过程具有复杂性和整体性,多种物质共同影响生命系统的表型和性状。通过转录组+代谢组的关联分析,从因到果进行分析,发现分子机制的变化机理。

客服微信:metware888
咨询电话:027-62433042
邮箱:support@metware.cn
网址:www.metware.cn
我就知道你“在看”