空间代谢组 | 最全分析内容解析(数据挖掘必看)
空间多组学频繁引爆科研圈,其中不乏空间代谢组的身影。空间代谢组学(Spatial Metabolomics)将代谢组学信息扩展到了三维的水平,研究小分子在组织切片中的空间分布,告诉我们变化“在哪里发生”,从而极大拓展了对代谢组学样品信息的认知。
空间代谢组数据复杂,如何挖掘有效信息成为一大难点,此时需要尽可能完善的生物信息学分析流程协助数据挖掘,以筛选与研究目的相关的核心代谢物,为此小迈调研了近40篇文献,对空间代谢组学分析内容进行汇总,最终汇总了9条分析内容,并在下文中进行一一介绍。
PART
01
代谢物空间分布图
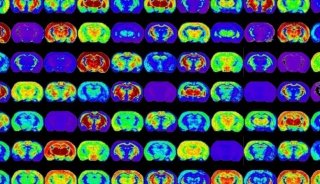
代谢物空间分布图作为空间代谢组的基础分析内容,文章中出现概率为100%。对原始数据进行平滑处理及Root Mean Square归一化后得到每个空间样品点的质荷比(m/z)信息,并转化为成像热图上的像素点,从而得到代谢物空间分布图。代谢物空间分布图主要展示代谢物在组织不同部位分布情况,可直接与H&E染色结果比对,以此了解代谢物积累部位,对疾病机理研究具有一定意义。

■ ■ ■ ■ ■
代谢物空间分布图示例
注:颜色从黑色到蓝、青、红色,代表目标物质在区域内含量依次增多。
PART
02
代谢物3D空间分布图
代谢物3D空间分布图是基本空间分布图的升级版本,除以颜色代表代谢物在不同部位的含量外,还加以高度展示,最终形成了3D图谱。同理代谢物3D空间分布图可了解代谢物积累部位,对疾病机理研究具有一定意义。

■ ■ ■ ■ ■
肝脏组织代谢物3D空间分布图(Zhiyuan Yuan et al,2021)
注:不同颜色代表代谢物含量高低。
PART
03
Segmentation分析
segmentation分析,原理是每个成像点都是一张质谱图(质谱图中是50-1300Da内所有的质谱峰),把这些质谱峰的m/z和对应的强度信息进行分析,相近的就用同一种颜色表示,最终形成了segmentationmap,可以参考segmentationmap结果将一个或多个颜色区域(cluster)作为感兴趣区域。segmentationmap分析可展示在病理过程中,代谢区域性变化,从而探索疾病发展过程,并利于区域特性对疾病分型、分级,对精准医疗具有一定意义。

■ ■ ■ ■ ■
脑组织Segmentation分析图(Alexandrovet al,2010)
注:相同颜色带别代谢特征相似区域。
PART
04
不同cluster相关性分析
对不同cluster进行相关性分析,以衡量不同cluster之间的距离,进一步了解生物组织不同区域变化过程中,区域之间的相互关系,以便阐释致病机理。

■ ■ ■ ■ ■
脑组织不同cluster矩阵距离分析(Alexandrovet al,2010)
PART
05
T-SNE分析
T-SNE(T-Distributed Stochastic Neighbor Embedding)是一种降维技术,将空间数据点的欧氏距离转化为条件概率来表征点间相似度(pairwisesimilarity),再使用梯度下降算法来使低维分布学习、拟合高维分布。T-SNE分析与Segmentation分析功能类似,主要将代谢趋势一致区域划分为一类,从而对组织进行空间分割。

■ ■ ■ ■ ■
T-SNE分析(Xiaowei Song et al,2019)
PART
06
UMAP分析
UMAP(Uniform Manifold Approximation and Projection for Dimension Reduction)是一种非线性降维技术,相对于T-SNE,其建立在黎曼几何和代数拓扑理论框架上的一种非常有效的可伸缩降维算法,特别对特高维度数据降维,UMAP算法更加快速。UMAP主要也是对组织进行空间分割,可将UMAP、T-SNE、Segmentation结果互相验证。

■ ■ ■ ■ ■
代谢物UMAP分析
PART
07
区域特征代谢物筛选
区域特征代谢筛选使用的方法为t-statisticsand representative single ion images分析,其核心为t统计量分析,可以准确地告知每段m/z与全局平均谱之间的关系。可根据此分析筛选区域内贡献度较高代谢物,并进行空间分布展示。筛选的代谢物可作为区域biomarker,并应用疾病或者癌症的诊断。

■ ■ ■ ■ ■
t-statistics and representative single ionimages分析展示
注:上图为segmentation分割区域,中间图为光谱特征收缩t 统计量,下图为特征代谢物空间分布图。
PART
08
代谢物与区域共定位分析
通过代谢物与区域共定位分析,可筛选与区域表达趋势一致代谢物,更利于区域biomarker筛选,结果以表格形式展示。该分析与t-statisticsand representative single ion images分析有异趣同工之妙。
PART
09
代谢物共定位分析
代谢物共定位分析(Colocalization)是一种发现与目标代谢物在空间上表达分布趋势一致的代谢物的分析方法。通过代谢物共定位分析挖掘代谢物之间的联系,以便解释致病机理,或寻找标志物panel。
■ ■ ■ ■ ■
代谢物共定位分析结果展示
PART
10
ROC分析
ROC(Receiver Operation Characteristic)分析是把灵敏度和特异度结合起来综合评价诊断准确度或判别效果的一种方法,可用ROC分析筛选判别不同区域标志物,ROC指标AUC值越大,对区域差异判别效果越好。
■ ■ ■ ■ ■
肺癌组织区域标志物空间分布图及箱型图(AUC>0.75)(YanyanChen et al,2021)
PART
11
空间代谢组与H&E染色关联分析
可使用t-SNE分析将空间代谢组数据投影到H&E染色图片上,以更好区分组织各区域,有利于病理学家诊断,以及确认外科手术成功与否。此分析未公开,如有需求需联系软件开发者。

■ ■ ■ ■ ■ t-SNE可视化生成组织学和代谢物空间分布合成图(SankhaS. Basu et al,2019)
★
迈维代谢提供优质的空间代谢组服务
1. 行业首个MALDI数据库:物质定性更准确

2. 业界最全数据库:鉴定物质更多
迈维代谢除拥有空间质谱成像数据库外,还拥有业界最全医学、植物本地高分辨数据库。医学(动物)本地数据库升级后,达到10000+种代谢物。
3. 物质数量达1000+:有效信息多
研发人员对空间代谢组实验方法进行优化,并结合空间代谢组数据库,单个项目检出高达1000+物质,二级定性物质可达300+,大大提高了定性物质数量及准确度,其中二级定性物质来源于MALDI数据库占比高达20%,由此可见空间代谢组数据库对物质鉴定数量及准确性很重要。
4. 数据分析和服务更专业
迈维代谢使用SCiLS™Lab专业成像软件进行离子图像和谱图的可视化,通过无限制样本数据的先进机器学习算法,实现空间分割的自动注释。同时支持手动划分区域,完成感兴趣区域代谢特性的个性化分析。如利用Segmentation、t-SNE、UMAP分析了解代谢空间分割详情,利用代谢物共定位分析挖掘代谢物之间的关系,利用代谢物与空间位置共定位分析筛选区域标志物。

■ ■ ■ ■ ■
部分分析内容展示
(TheodoreAlexandrov et al,2010;HuHet al,2021;XiaoweiSong,2019;Yanyan Chen et al,2021)
为帮助更好的理解空间代谢组技术及其应用,迈维代谢撰写了广受欢迎的空间代谢组书籍,包含空间代谢组技术介绍、实验流程、分析内容及应用方向,如有需求可联系迈维代谢当地销售领取,没有联系方式可加小迈微信:metware888,进行咨询。
迈维代谢空间代谢组书籍展示
技术介绍/实验流程/分析内容/应用方向
彩蛋时刻
迈维代谢空间代谢组产品除物质定性更准确、鉴定数量更多、数据分析和服务更专业外,特推出7周年限时活动,全场5折如有需求可联系当地销售工程师或者添加咨询微信(metware888)。

客服微信:metware888
咨询电话:027-62433042
邮箱:support@metware.cn
网址:www.metware.cn