生信分析系列干货 | 蛋白质组学数据挖掘神器 — 功能富集分析
生信系列干货强势来袭

随着对人类等生物体全基因组测序的完成,大家逐渐意识到与基因组学相比,动态变化的蛋白质组学才是系统阐述不同状态下生物体功能的关键,为众多疾病机理的阐明及治疗提供理论依据和解决途径。
随着蛋白质组学的研究逐渐增多,生信分析的作用也越来越大。景杰生物作为蛋白质组学研究的引领者,特此创建了蛋白质组学生信分析系列干货福利分享。我们的生信部门专业人员将手把手教你进行蛋白组学数据分析,打造一站式生信学习平台!
本次的干货主题为:蛋白质组学数据的富集分析,后续会定期推出蛋白质组学生信分析系列其他干货内容,记得关注收藏哦~
富集分析概念介绍
一般来说,我们通过常规的差异分析之后会得到差异蛋白列表,少则几十,多则成百上千。如何将这些差异蛋白与功能和表型相关联,锁定差异蛋白参与的关键功能,减少验证的工作量呢?这时候就需要我们的富集分析出场了。
富集分析通过比较差异蛋白在某种功能中占比富集程度来筛选关键功能。举个例子:如果实验组与对照组两组中共鉴定到2000个蛋白,其中有200个蛋白属于功能A,则蛋白占比为10%;在实验组与对照组两组间共筛选出100个差异蛋白,其中有90个蛋白属于功能A,则蛋白占比为90%。上述数据经过富集分析(以fisher精确检验为例)后,功能A的富集倍数是90%/10% = 9,fisher精确检验p<2.2e-16,则功能A显著富集。
富集分析手把手教学
那么作为代码小白,我该如何进行差异蛋白的功能富集分析呢?
今天我们会推荐两个常用的在线进行富集分析的网站:Metascape和DAVID,并对其中的操作和结果解读进行详细说明,现在就让我们开始这趟生信之旅吧~

Metascape


Metascape官网
网址:https://metascape.org/gp/index.html#/main/step1

Step1. 数据上传
I. 上传需要分析的蛋白列表(xls,xlsx,csv和txt格式文件均可;或直接输入蛋白名称列表,以逗号、冒号、空格、制表符或分行隔开;蛋白名称支持Gene Symbol、Entrez Gene ID、RefSeq、Ensembl、UniProt和UCSC等)II. 以蛋白组常用的Uniprot为例,直接输入一个蛋白集合,点击Submit



Step2. 物种选择
选择样本对应的物种名称(网站一般会根据输入的蛋白名自动匹配物种)


Step3. 选择数据分析模式
I. Express Analysis:直接按照默认设置进行分析,不需要任何自定义
II. Custom Analysis: 可以自定义一些参数


Step3.1. Express Analysis分析模式
I. 点击Express Analysis默认模式II. 完成后点击Analysis Report PageIII. 查看分析结果

IV. 分析结果主要包含:富集分析结果柱状图和富集分析表格柱状图:展示显著富集的功能通路,柱子长度和颜色代表-log10转换后的富集p value值,柱子越长,颜色越深,代表该功能富集越显著。分析表格:Count代表在这条功能中输入蛋白的数目,% 代表输入蛋白中属于这条功能蛋白的百分比,Log10(P)和Log10(q)分别为Log10转换后的富集分析P值和多重检验矫正后的q值。



Step3.2. Custom Analysis模式
I.进行富集分析的自定义设置II.设置功能富集的参数III.修改富集背景IV.选择进行富集分析的功能V.开始富集分析VI.输出结果与Express Analysis类似,不再详述。


DAVID


Step1. 开始数据分析


Step 2. 数据上传


Step3. 富集分析


Step4. 结果展示


景杰生信结果展示
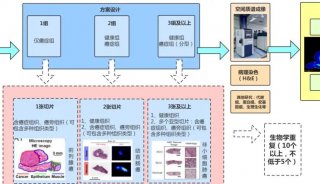
虽然上面介绍的两个在线网站可以完成我们需要的富集分析,但是如果我想在自己的文章里展示富集分析的结果,只有一个简单的柱状图似乎并不能吸引审稿人的眼球!如何才能用更高大上的图形展示我们的富集结果呢?景杰生信提供的功能富集分析结果,除了完整的富集分析excel表格,还会提供多张个性又美观的可视化图片,让你的富集分析与众不同。
 图1 显著富集功能通路气泡图
图1 显著富集功能通路气泡图
图2 显著富集功能通路弦图
 图3 显著富集功能通路Circos图
图3 显著富集功能通路Circos图 图4 差异表达蛋白显著富集KEGG功能通路图
图4 差异表达蛋白显著富集KEGG功能通路图 图5 景杰生信产品金字塔
图5 景杰生信产品金字塔 图6 景杰生信产品订购二维码
图6 景杰生信产品订购二维码想要获得这些分析图,可以扫描图6的二维码填写表单直接联系我们订购生信分析产品,也可以使用景杰生信云平台小工具自己动手免费分析哦,其中的一些图片还可以根据自己课题关注的相关功能进行个性化修改,具体如何操作,请期待我们下一期的推文吧!
参考文献:1. Zhou, Yingyao et al. 2019. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nature Communications.2. Huang, Da Wei et al. 2009. Systematic and integrative analysis of large gene lists using DAVID Bioinformatics Resources. Nature Protocols. 3. Sherman, Brad T et al. 2022. DAVID: a web server for functional enrichment analysis and functional annotation of gene lists (2021 update). Nucleic Acids Research. 4. Kanehisa, M., & Goto, S. 2000. KEGG: kyoto encyclopedia of genes and genomes. Nucleic Acids Research.
本文由景杰生物团队报道,欢迎转发到朋友圈。如有转载、投稿等其他合作需求,请文章下方留言,或添加微信ptm-market咨询。
景杰生物

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章
-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章
-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章

-
微信文章







