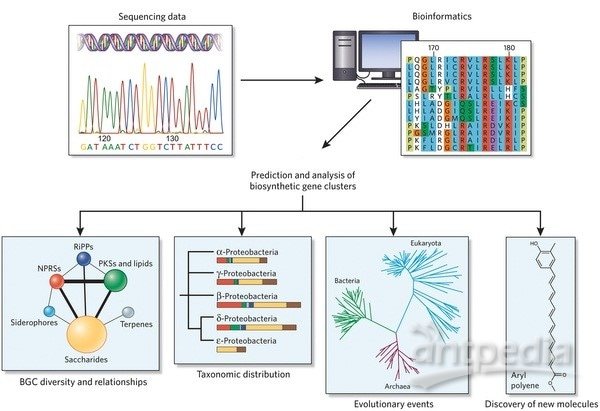
基因组学常规流程中SPIA通路分析方法
基因组学常规流程中通过实验样本组与组之间的比较得到成千上百的差异基因,再通过通路分析,从生物学角度探究差异基因的功能。但是常规的ORA、FCS方法往往忽视了基因在通路中复杂的相互作用关系,所以给大家介绍一种考虑更周全的分析方法SPIA。
SPIA整合了差异倍数、基因集显著性和拓扑分析,为每条通路计算一个总体概率值PG。其与过表达分析(ORA)以及功能集打分(FCS)分析相比,具有以下优点:
1. 不仅关注通路中的基因集,而且更重视基因在通路中的位置信息。
2. 充分考虑到拥有多种功能并且以不同角色参与到多条通路中的基因。
3. 除此之外,该方法还利用了通路的拓扑结构进行分析。
使用 PNDE 和 PPERT 分别代表富集分析(ORA)和拓扑分析(PT)的p值,假设有一条涉及6个基因的通路(如下图),图a和图b均有两个基因映射到该通路上,图1a是基因B与基因F,图1b中是基因A与基因B;显然图1a中的两个差异基因对这个通路的影响没有图1b中的大,但是使用富集分析计算出来的p值却相等。SPIA分析出的结果能很好的刻画这种不同(下图PPERT值的差异)。
 图1 PPERT值的差异
图1 PPERT值的差异
SPIA选择域直观展示
如图2,左上角x轴是PNDE,y轴是PPERT,如果单独有两个维度选择显著通路,PNDE选择区域2、3、6;PPERT选择区域1,2,4;SPIA选择1、2、3、5。
图2 SPIA选择域直观展示
SPIA与GSEA相比更有敏感性;与ORA相比有更好的特异性以及更好的通路排名。
历年被引用数:
组学应用参考案例:
1. Hoyles L, Fernández-Real JM, Federici M, Serino M, Abbott J, Charpentier J, Heymes C, Latorre Luque J, Anthony E, Barton RH, Chilloux J, Myridakis A, Martinez- Gili L, Moreno-Navarrete JM, Benhamed F, Azalbert V, Blasco-Baque V, Puig J, Xifra G, Ricart W, Tomlinson C, Woodbridge M, Cardellini M, Davato F, Cardolini I, Porzio O, Gentileschi P, Lopez F, Foufelle F, Butcher SA, Holmes E, Nicholson JK, Postic C, Burcelin R, Dumas ME (2018) Molecular phenomics and metagenomics of hepatic steatosis in non-diabetic obese women. Nat Med 24(7):1070–1080 (IF=30.641)
2. Riikka H., Juan C. Landoni, Kati J. Ahlqvist, et al. Defects in mtDNA replication challenge nuclear genome stability through nucleotide depletion and provide a unifying mechanism for mouse progerias, Nature Metabolism (2019). DOI:10.1038/s42255-019-0120-1 (2019年Nature的新刊)