Nature Plants文章:精准高通量检测R-loop分布的新方法
来自清华大学生科院,清华-北大生命科学联合中心的研究人员发表了题为“The R-loop is a common chromatin feature of the Arabidopsis genome”的文章,首次报道了精准、高效、高通量检测R-loop的方法,并利用此方法首次揭示模式植物拟南芥基因组中R-loop的分布特征。
这一研究成果公布在Nature Plants杂志上,文章的通讯作者为清华大学生科院孙前文和杨雪瑞,第一作者为徐炜博士。Nature Plants杂志专门邀请加州大学戴维斯分校Frederic Chedin教授为本研究撰写评论文章,彰显此项研究在领域内的重要性。
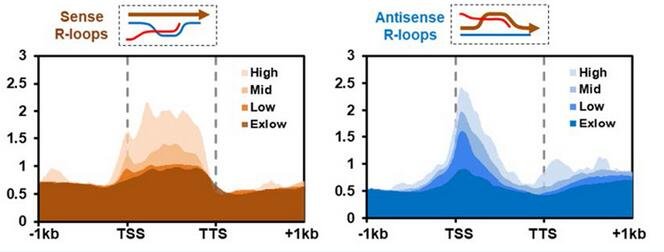
Metaplot分析显示拟南芥基因组中既存在正义R-loops(sense R-loops,由基因自身转录本与其模板链形成的R-loop),也存在反义R-loop(antisense R-loops,大多是由基因的反义转录本在正义链转录起始位点形成的R-loop)
R-loop是一种特殊的染色体结构,由一条RNA:DNA杂合链和一条单链DNA所组成。目前越来越多的证据发现这种独特的染色质结构在原核和真核生物中普遍存在,在很多关键的生物学过程中发挥重要功能,包括染色质修饰、转录调控、DNA损伤修复以及基因组稳定性等。DRIP-seq(DNA:RNA hybrid immunoprecipitation and sequencing)是一种基于特异识别DNA:RNA杂合链抗体的免疫共沉淀及高通量测序分析技术,已被用来检测人、小鼠、酵母等模式生物全基因组水平的R-loop分布。但已有的R-loop全基因组检测方法存在严重的技术缺陷,对数据的质量有不可忽视的影响。此外,植物全基因组水平的R-loop分布特征尚不清楚。
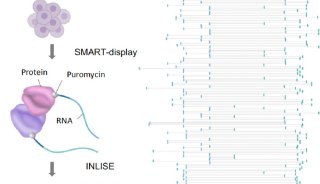
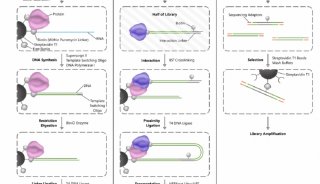
这项研究以模式植物拟南芥为材料,在传统R-loop全基因组检测技术基础上开发出一种全新的检测方法:基于单链DNA建库的DNA:RNA杂合链免疫共沉淀及高通量测序技术(简称ssDRIP-seq, single-strand DNA ligation-based library construction of DNA:RNA hybrid immunoprecipitation and sequencing)。
基于单链DNA建库的ssDRIP-seq原理图
与已报道的全基因组R-loop检测方法相比,ssDRIP-seq表现出易操作、精准、高效、高度灵敏、可重复性好等优点,并且可获得DNA链特异性信息。基于ssDRIP-seq,该研究获得了拟南芥全基因组水平R-loop的精准分布特征,如R-loop在拟南芥基因组中分布广泛且相对稳定,与基因表达、DNA甲基化、染色质修饰、GC/AT skew等存在着紧密关系。不同于哺乳动物基因组,拟南芥中广泛存在一类反义长非编码RNA在正义链转录起始位点形成的R-loop,推测这类特别的R-loop在调节基因转录起始或控制基因转录过程中发挥重要作用。该研究还基于所获得的数据对R-loop在基因沉默和基因组构成等中的功能进行了展望。
孙前文实验室成立于2014年秋,重点关注植物R-loop的形成、稳定和解除的调节机制及相应的生物学功能。本研究是孙前文实验室继报导R-loop调节长非编码RNA表达并影响拟南芥开花(Science, 2013)和R-loop调控水稻根的向地性(Molecular Plant, 2017)之后的又一重要成果。ssDRIP-seq这种精准、高效、高通量R-loop检测技术的建立为随后的功能研究提供了重要手段,将成为全基因组R-loop分析的标准,并全面打开全基因组水平分析R-loop的分布及后续功能研究的大门。