单链Oligo合成与DNA组装技术 (二)
2.2 Golden Gate组装
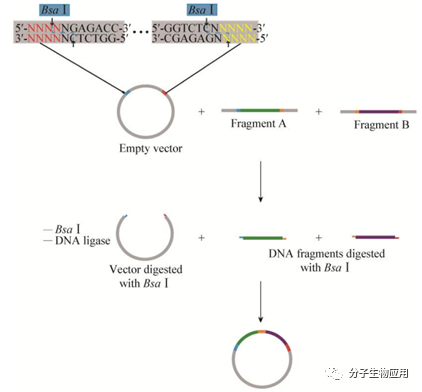
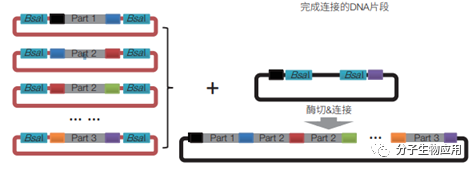
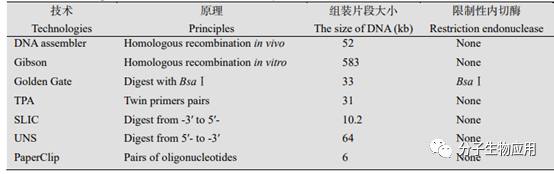
Golden Gate组装技术是基于非同源重组的代表性技术,其利用Ⅱ型限制性酶 BsaⅠ在识别位点外部切割的特性,设计特异的突出序列同时组装多个片段,且酶切和酶连可以同时进行,原理如图3所示。首先,扩增目的片段,在两端加上BsaⅠ识别序列,同时在识别序列内侧加上不同的4 nt突出部分,相邻片段衎接处的4 nt反向互补配对,然后将片段分别插入酶切前的中间载体,因此一共可以设计256个突出的末端。利用E.coli富集含有不同目的片段的中间载体,将这些载体与最终的载体(含有2个相邻的BsaⅠ酶切位点)混合,加入BsaⅠ和DNA连接酶,同时进行酶切和酶连组装多达9个DNA片段。
2011年,Cermak等人利用Golden Gate高效组装了TALEN敲除系统中的重复序列,整个过程只需要两步:第一步先将最多10个RVDs按顺序组装,每个RVDs可识别1个核苷酸;第二步再将这些RVDs和TALEN系统的其它功能元件如NLS、AD等组装成最终的敲除质粒。2015年,Lauressergues等人使用Golden Gate构建microRNAs表达质粒,探究了苜蓿、拟南芥中2条 microRNAs的转录调控作用及其它5条microRNAs编码的具有调控作用的短肽对植物主根生长的影响。
图3 基于Bsa I内切酶的Golden Gate组装
图3 Golden Gate组装
2.3 同尾酶BioBrick、iBrick 和 C-Brick
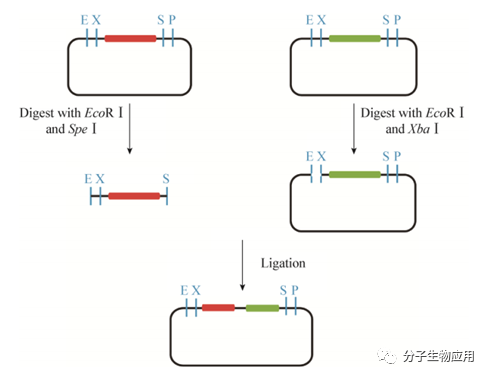
BioBrick是合成生物学领域一个经典的基因组装技术,BioBrick的原理如图4所示。同尾酶可以识别不同的DNA序列但是能切出相同黏性末端的一类限制性内切酶,但其切开的末端相互连接后不会再形成原酶切位点。其利用同尾酶Xba Ⅰ和 Spe Ⅰ酶切产生相同的粘性末端,酶连后片段连接处会产生86p的疤痕,Xba Ⅰ和Spe Ⅰ都不再识别连接处的疤痕,这样即可在不损失酶切位点的情况下不断组装DNA片段。2014年,国内学者利用归位内切酶I-Sce Ⅰ和PI-Psp Ⅰ可产生相同粘性末端的特点,模仿BioBrick原理设计出一种名为iBrick的DNA 组装方法。归位内切酶识别的序列比普通的限制性内切酶长,在DNA中出现的频 率极低,因此iBrick相比于BioBrick对DNA片段序列几乎没有限制。但较长的识别序列在酶切酶连组装的同时也产生了更长的疤痕,为了解决这个问题,科学家利用一种应用了CRISPR-Cas系统的DNA组装方法C-Brick。C-Brick利用Ⅴ型CRISPR/Cas 系统蛋白Cpf1核酸内切酶,在PAM位点切割产生5nt的粘性末端。该方法利用sgRNA识别长序列的特点避免了对待组装片段序列的要求,同时可灵活设计sgRNA以识别不同的序列,只产生5 bp的疤痕,完美地规避了 BioBrick 的缺点。
图4 BioBrick技术的组装原理
2.4 基于DNA聚合酶的策略。
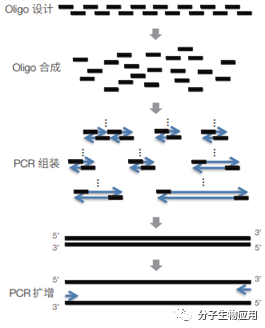
该方法首先合成有互补配对重叠区的Oligo,利用PCR扩增的原理,可以对有互补重叠区的Oligo进行延伸连接获得小DNA片段,然后小DNA片段以重叠PCR的方式进行逐步连接获得目的DNA片段,以其作为模板,进一步PCR扩增可以大量获得目的片段(图5)。此方法(polymerase cycling assembly,PCA)无须依赖额外的DNA连接酶,直接从人工合成的Oligo开始组装,操作简单、快速。Stemmer 等人曾用此法实现基因及质粒的一步拼装。Smith等人则 通过增加 Taq 连接酶的步骤用此法合成了 ФX174 噬菌体 的基因组。
图5 基于DNA聚合酶合成法
3. 其他DNA组装技术
除了以上最常用的基因组装技术外,近年来发展起来很多其他DNA组装方法,如:SLIC、UNS、 PaperClip和 MASTER等方法。