多样化C to T植物碱基编辑器被开发
近日,电子科技大学张勇教授、马里兰大学YiPing Qi博士、扬州大学张韬教授课题组合作于《Plant Biotechnology Journal》发表了题名《Improved plant cytosine base editors with high editing activity, purity, and specificity》的研究论文。该研究针对7种胞嘧啶脱氨酶、4种不同的碱基编辑策略构建了21个不同的碱基编辑系统,对这21种胞嘧啶碱基编辑系统的编辑效率、编辑范围、编辑纯度及特异性等编辑特性进行了细致比较,并将胞嘧啶碱基编辑器应用于miRNA调控相关基因、籽粒相关基因、除草剂抗性基因编辑,为碱基编辑器在植物功能基因组研究及分子育种实践中的有效应用提供了多样化分子工具及应用实例。
碱基编辑系统将脱氨酶与失去双链剪切活性的Cas蛋白(dCas9或者nCas9)融合作用于单链DNA(ssDNA),以实现靶位点的碱基替换。碱基编辑器不需要产生DNA双链断裂(DSB),也不需要提供供体DNA,且不会产生细胞毒性,因此在动植物基因碱基编辑上有广泛应用潜力。自2016年胞嘧啶碱基编辑器(CBE)首次在哺乳动物细胞中被开发、证明,CBEs在扩大编辑范围、优化编辑窗口、减少编辑副产物和增强特异性等方面得到了快速发展。迄今为止,rAPOBEC1、hAID、PmCDA1、human APOBEC3A及APOBEC3B等多种胞嘧啶脱氨酶结构域被应用于植物CBE碱基编辑器中。但相对研究及应用需求,现有CBE仍有较多待改进处,如何有效提升CBE编辑效率及特异性、如何进一步拓展编辑窗口、如何提升C to T编辑纯度等关键问题亟待明确研究数据回答。
电子科技大学张勇教授课题组及其合作者,基于水稻原生质体及通量测序策略,明确评估了7种不同脱氨酶结构域在CBE应用中的编辑效率、C to T纯度、活性窗口、序列背景依赖性以及全基因组编辑特异性等关键碱基编辑器活性及特性,并在此基础上,开发了高效植物CBE碱基编辑新工具,有效提升了编辑效率、编辑纯度、编辑窗口,并降低了非特异性编辑。同时,研究者进一步应用构建的高效CBE工具在水稻中快速、高效且精准的创制了重要农艺性状相关基因修饰材料,并鉴定了新的水稻OsALS、OsEPSPS抗除草剂变体,为CBE碱基编辑器在植物功能基因组研究及分子育种实践中的有效应用提供了进一步的可行例证。全文研究结果简述如下:
1、构建7种不同脱氨酶胞嘧啶碱基编辑系统CBE_V01,基于水稻及拟南芥原生质体转化、通量测序,针对水稻内源多GC位点及拟南芥甲基化位点检测到有效的定向碱基编辑事件。研究结果表明:在水稻原生质体中,A3A/Y130F-CBE_V01的C-T转化效率最高,其次是PmCDA1-CBE_V01, hAID-CBE_V01 , A3A-CBE_V01; rAPOBEC1-CBE_V01和eA3A-CBE_V01碱基编辑活性较低,eA3A/Y130F-CBE_V01没有碱基编辑活性;A3A/Y130F-CBE_V01碱基编辑窗口为远离PAM端4-15位,PmCDA1-CBE_V01碱基编辑窗口为远离PAM端1-6位;A3A/Y130F-CBE_V01在拟南芥原生质体甲基化位点显示出最高的编辑效率,编辑窗口与水稻一致(图 1);
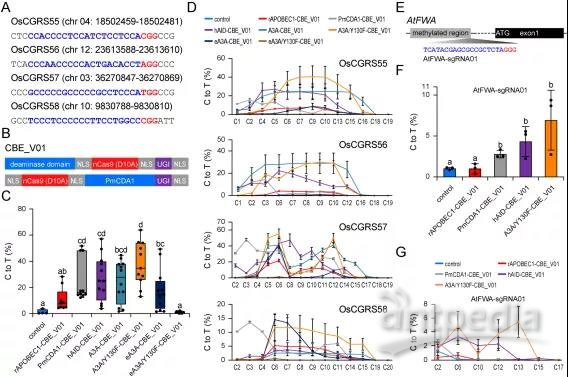
图 1. 基于植物原生质体瞬时表达及高通量测序的7个CBE_V01系统基因组碱基编辑特性分析
2、基于农杆菌介导的遗传转化,将高效的A3A/Y130F-CBE_V01应用于水稻重要性状基因修饰工作,在稳定转化再生植株中检测到有效的单基因、多基因编辑事件,获得了对应农艺性状改良再生植株(图2),且在水稻中筛选了新的OsALS、OsEPSPS抗除草剂变体;
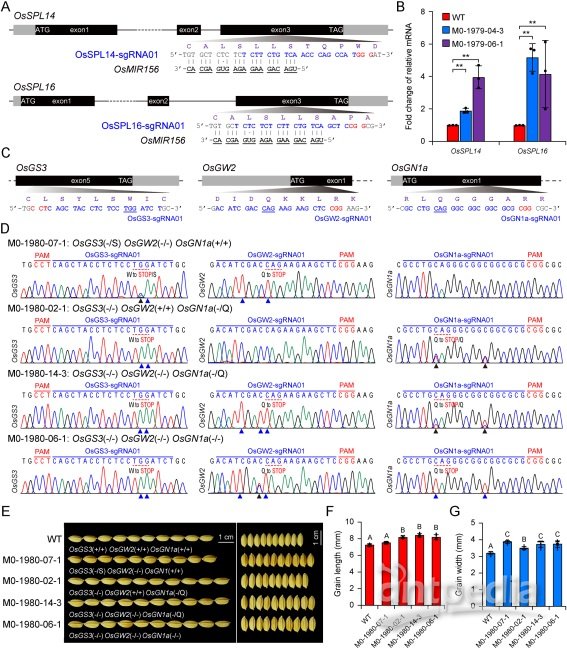
图 2. 基于农杆菌遗传转化的A3A/Y130F-CBE_V01水稻单基因、多基因编辑
3、基于上述工作进一步开发CBE系统,应用MS2-MCP招募UGI的策略,高度富集UGI,构建CBE_V04系统,基于水稻原生质体转化及高通量测序,针对水稻内源基因检测到有效的定向碱基编辑事件。结果表明:与CBE_V01相比,CBE_V04系统的碱基编辑效率相当;CBE_V04系统的碱基编辑窗口与CBE_V01一致;A3A/Y130F-CBE_V04的C-T转化效率最高,与CBE_V01一致;A3A/Y130F-CBE_V04的indel效率比A3A/Y130F-CBE_V01低,其编辑产物纯度高,且不改变编辑效率及窗口(图3);

图 3. 基于植物原生质体瞬时表达及高通量测序的CBE_V04系统基因组碱基编辑特性分析
4、运用WGS全基因组评估水稻10种CBE系统的脱靶效应,选择6个CBE_V01和4个CBE_V04碱基编辑器,59个水稻独立单株进行全基因组测序及分析。结果表明:不同CBE系统的SNV随机分布,不依赖于sgRNA,为全基因组脱靶效应;PmCDA1和rAPOBEC1的CBE_V01及CBE_V04系统与对照相比SNV数量略有增加;hAID-CBE_V01产生的SNV最多,其次为A3A/Y130F-CBE_V01;与其它胞嘧啶脱氨酶相比,PmCDA1导致的C-T转化倾向最小(图 4);

Figure 4. 通过WGS全基因组评估水稻10种CBE系统的脱靶效应
-
焦点事件

-
焦点事件
-
焦点事件

-
企业风采

-
项目成果
-
科技前沿


















