Oligo芯片的构建及数据分析
实验概要
本实验在生物素标记cRNA片段化的基础上,提供了小鼠全基因组Oligo芯片的构建及数据分析流程。
实验步骤
1. 芯片杂交
使用Affmetrix Hybridization Oven 640,先进行Test芯片的杂交、清洗染色、扫描和分析,根据Test芯片的结果再杂交Real芯片。
1) 根据表1配制杂交液(己将杂交过程中会损失部分(10-20ul)考虑在内,例如芯片是49/64 Format,需要配制杂交液300ul)。

2) 轻弹管壁数次使其充分混匀,瞬时离心(约5 sec)收集溶液于管底。
3) 将杂交液置于加热块上,99℃温育5 min。
4) 其间,用一次性吸头通过芯片上septa注入相应体积1×Hybridization Buffer,将芯片置于杂交炉中,45oC温育10 min,60 rpm旋转。
5) 将99℃已温育5 min的杂交液转移到另一加热块上,45oC温育5min。
6) 从加热块上取出杂交液,微型离心机最大转速离心5min,以除去杂交混合液中的不溶性杂质。
7) 从杂交炉中取出芯片,吸出1 XHybridization Buffer注入200ul澄清杂交液,不要吸取管底的不溶性物质。
8) 将芯片平衡放置于杂交炉中,45℃,60 rpm旋转杂交16h,进行下一步。
2. 芯片清洗染色
使用A伪metrix Fluidics Station 450进行芯片洗染:
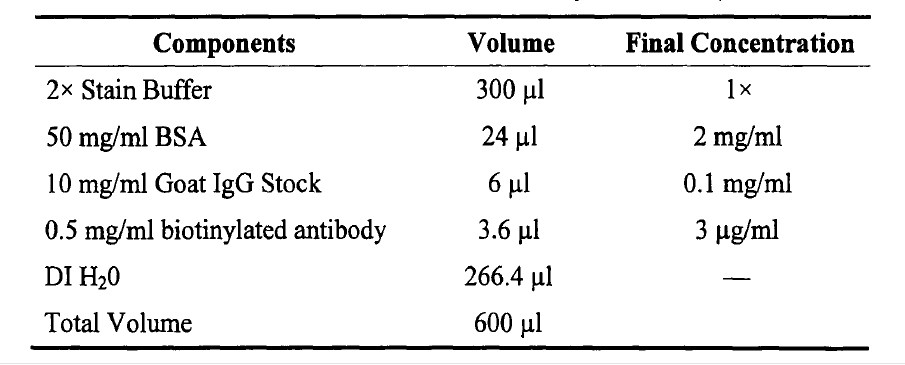
1) 取1.5 ml EP管,按照表2配制SAPE Stain Solution,充分混匀,瞬时离心(约5 sec)收集溶液于管底,取600ul到另一1:5 ml EP管中。

2) 取1.5 ml EP管,按照表3配制Antibody Solution,充分混匀,瞬时离心(约5 sec)收集溶液于管底。
3) 杂交结束后,吸走杂交液,注入200ul Non-Stringent Wash Buffer(Wash Buffer A)。
4) 在GCOS软件中运行程序‘`EukGE-WS2v5",开始芯片清洗染色。
3. 芯片扫描
使用Affymetrix Scanner 3000对芯片进行扫描,得到荧光信号图像。
4. 数据分析
使用Affymetrix软件系统GeneChip Operating software (GCOS 4.0)对芯片图像进行数据提取,得到.DAT, .CEL, .CHP和.RPT文件。
1) 数据预处理
通过扣除背景值和噪声修正,对数据进行预处理得到信号值。
2) 基因表达的判别
基因是否表达主要是根据PM和MM强度差异的相对指标予以判定的。对于第i个probe pair:
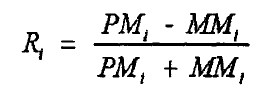
Ri分布在0-1之间,当Ri越接近于1, PM和MM的差异越显著。对于每一个probe
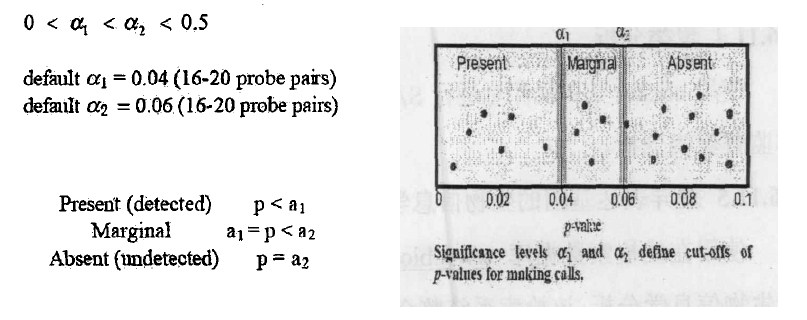
set的11对probe的Ri值,采用(单侧)Wilcoxon's ranktest计算p值,根据p值判断基因是否表达。当p<0.04,
PM和MM探针有显著性差异,基因表达,判定为“P";当p>0.06,
PM和MM探针没有显著性差异,基因不表达,判定为“A",如果0.04印<0.06,基因表达的判定处于临界状态,判定为“M"(如图所示):
3) 差异表达基因的筛选
将GeneChip Operating software (GCOS 1.4)得到的原始信号值,用dChip
software进行不变集非线性归一化;用SAM软件分别筛选MT /
小鼠和MT-/-小鼠DOX给药前后的变化基因,按照q-value(%)<5%的标准,把ratio
>2的基因认为是DOX给药后发生上调改变的基因,ratio<0.5的基因认为是DOX给药后发生下调改变的基因;筛选出差异表达基因用于生物信息学分析。各组间相互比较示意图见图,箭头的末端为每个两两分析中的参照。
4) 聚类分析
使用cluster 3.0软件对进行SAM软件筛选后的基因的信号值进行非监督聚类分析。
5) 差异表达基因的生物信息学分析
使用在线检索数据库对筛选出的差异表达基因进行生物信息学分析。该检索系统整合了包括Unigene, Locus link, OMIM,NCBI dbEST, NCBI CDD在内的多种公开数据库,并且提供Gene Ontology和Pathways (KEGG} Genmapp and Biocarta)分析。
-
项目成果