Cell Research 肠道菌群调控宿主RNA甲基化和基因表达新机制
肠道菌群微生物组学是近年来研究热点,肠道菌群在维持宿主生理平衡和健康中发挥着重要作用,在人和动物疾病治疗方面具有极大的应用前景。研究表明,肠道菌群及其代谢产物可调节宿主基因表达。随着研究的深入, 肠道菌群和宿主之间的相互作用机理也越来越多被发现,特别是通过表观遗传影响宿主的基因表达。如最新研究发现肠道菌群可调控宿主DNA甲基化修饰和组蛋白修饰【1】。
近年来,mRNA上的化学修饰被发现可调控基因表达的多个层面,参与细胞分化、生物个体发育及癌症疾病等重要生命过程。该领域被称为RNA表观遗传学或表观转录组学,开启了表观遗传领域新的研究热潮。美国芝加哥大学华人科学家何川教授在其中做出了卓越的贡献。N6-methyladenosine (m6A)修饰是真核生物mRNA上发生最为广泛的化学修饰,参与调控mRNA稳定性、剪接加工、转运以及翻译等一系列mRNA代谢过程,对mRNA的命运决定发挥重要作用【2】。哺乳动物细胞内m6A甲基化修饰主要是由METTL3/METTL14/WTAP复合物催化形成,同时m6A修饰可被去甲基化酶FTO/ALKBH5可去除,宿主体内m6A修饰调控基因表达是动态可逆的【3】。目前以m6A修饰为引擎的RNA表观转录组学研究成为国际研究热点,已广泛应用于各类疾病模型研究。然而肠道菌群和宿主的互作过程中,宿主RNA上的修饰如m6A是否会受到微生物的影响,以及有没有可能微生物通过m6A来与宿主对话,都是尚无答案却又引人入胜的问题。
12月17日, Cell Research杂志在线发表了美国芝加哥大学潘涛课题组和中山大学骆观正课题组最新研究成果Transcriptome-wide reprogramming of N6-methyladenosine modification by the mouse microbiome,该论文首次报道了哺乳动物肠道菌群调控宿主RNA甲基化修饰特征【4】。

为了探讨肠道菌群与宿主相互作用的RNA表观遗传学分子机制,研究人员建立无菌小鼠和正常菌群小鼠平行对照试验,通过高分辨质谱检测和m6A-seq RNA测序分析,结果发现无菌小鼠与正常小鼠m6A水平在多个器官组织都存在显著差异,肠道菌群可抑制宿主mRNA甲基化修饰。与之相一致的是,肠道菌群引起的宿主m6A变化直接导致宿主多器官组织mRNA转录组变化。更为有趣的是,相比其他器官组织,肠道菌群对宿主不同年龄阶段脑组织m6A水平调控最为明显,并且对mRNA上m6A位点调控主要集中于CDS基因编码区,这意味着肠道菌群对宿主RNA甲基化修饰调控具有器官特异性,肠道菌群对宿主m6A水平负向调控可能与m6A调节转录本稳定性和基因剪接有关,用于维持宿主重要基因稳定表达【5】。
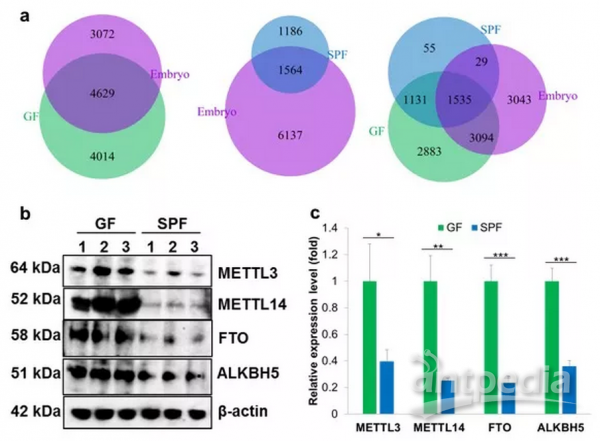
研究人员进一步分析发现,无菌小鼠脑组织m6A模式与胚胎期小鼠脑组织更为相似,提示m6A甲基化修饰对胚胎早期发育的重要性。哺乳动物出生后暴露于外界微生物环境中,宿主体内m6A水平发生相应的变化调控基因表达以适应外界环境【6】。研究人员比较了m6A甲基化酶和去甲基化酶在无菌小鼠和正常小鼠体内表达水平,发现多种酶的表达水平在两组动物之间存在明显差异(下图),为肠道菌群调控宿主m6A的分子机制提供了重要证据。肠道菌群对宿主脑组织等器官的RNA甲基化调控可能是通过其代谢产物与宿主细胞相互作用实现的,具体分子机制有待于深入研究。
这项研究是继病毒等病原体对宿主m6A影响的报道以来【7, 8】,首次将m6A与肠道微生物-宿主相互作用联系起来,揭示了肠道菌群与宿主相互作用的RNA表观遗传学分子机制,为今后深入理解微生物组影响人类健康的机理开启了新的一扇门,研究成果在肠道菌群相关的疾病治疗和人类健康中具有潜在的应用价值。
据悉,芝加哥大学汪肖云博士和中山大学博士生李言是论文共同第一作者,中山大学骆观正教授和芝加哥大学潘涛教授为论文共同通讯作者。
论文链接:https://www.nature.com/articles/s41422-018-0127-2
参考文献
1. Romano, K. A. et al. Cell Host. Microbe 22, 279–290 (2017).
2. Roundtree, I. A., Evans, M. E., Pan, T. & He, C. Cell 169, 1187–1200 (2017).
3. Yang, Y., Hsu, P. J., Chen, Y. S. & Yang, Y. G. Cell Res. 28, 616–624 (2018).
4. Wang, X.Y. et al. Cell Res. 0:1–4 (2018).
5. Wang, X. et al. Nature 505, 117–120 (2014).
6. Diaz Heijtz, R. et al. Proc. Natl. Acad. Sci. USA 108, 3047–3052 (2011).
7. Gokhale N.S. et al. Cell Host Microbe 20, 654-665 (2016).
8. Kennedy E.M. et al. Cell Host Microbe 19, 675-685 (2016).