三篇Nature子刊:如何克服CRISPR的脱靶效应
规律成簇的间隔短回文重复CRISPR与内切酶Cas9的组合,原本是细菌抵御病毒的重要武器,现在这一组合已经成为了一个通用工具,被用于在真核生物中进行位点特异性的基因组修饰。
最近Nature Biotechnology杂志上发表了三个研究团队的研究成果,研究人员深入分析了CRISPR的脱靶效应,并提出了非常可行的解决之道。
CRISPR系统修改目的基因的简便性,让几乎所有实验室都有可能随心所欲的进行基因组编辑。你需要做的只是,在自己感兴趣的细胞或生物中,表达Cas9内切酶和引导RNA(gRNA)。引导RNA是一段与目标DNA片段匹配的短RNA,它指导着CRISPR-Cas9的DNA剪切活性。
不过困扰其他定向核酸酶的老问题——脱靶效应,也同样影响着CRISPR系统。人们发现,Cas9会在基因组的一些脱靶位点进行剪切。弗吉尼亚大学Mazhar Adli等人的研究显示,在人类基因组中Cas9除了结合目的基因以外,还殃及了许多其他的位点。
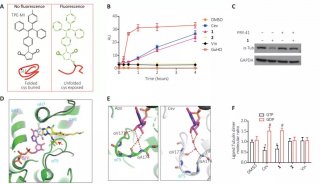
Adli及其同事构建了失去酶活性的Cas9(dCas9),并将其与12种不同的gRNA搭配。随后他们通过ChIP-seq技术,在HEK293T细胞中检验了dCas9在全基因组的结合情况。研究人员发现,脱靶结合的多少主要取决于gRNA。对于绝大多数gRNA而言,dCas9能结合几十到几百个脱靶位点(Kuscu et al., 2014)。研究显示,具有酶促活性的Cas9的确会切割这些ChIP-seq鉴定的脱靶位点(尽管不是全部)。
这项研究告诉我们,CRISPR系统的特异性可能无法满足某些应用的需求。应该怎么办呢?Adli等人认为,可以用Cas9 nickase代替野生型Cas9进行基因组编辑。Cas9 nickase是一种突变型Cas9,只切割一条DNA链。
然而,哈佛大学David Liu研究组和麻省总医院Keith Joung研究组找到了更好的办法(Guilinger et al., 2014 and Tsai et al., 2014)。他们的策略是对Cas9进行基因工程改造,让其依赖二聚化才能酶切,就像锌指核酸酶(ZFN)和TALEN那样。二聚化酶有着更严格的序列要求,理论上应该能够大大减少脱靶位点的数量。
这两个研究团队不约而同地将Fok1核酸酶融合到了dCas9的N端,与ZFN和TALEN中的方向相反。他们发现,融合而成的二聚酶与Cas9的正确切割效率相差不多。Liu和Joung两个团队还通过深度测序,检验了野生型Cas9的那些脱靶位点,发现二聚酶显著减少了可检测到的脱靶事件。