【指南】Cytoscape之stringAPP蛋白互作分析详解
覆盖蛋白互作网络分析一直以来是蛋白质组学数据分析的重要部分,现在常用的如String网络数据库,它能提供两千多个物种的互作信息,展示的网络系统较小。2019年在J Proteome Res. 杂志上介绍了Cytoscape 其中一款插件StringApp(Cytoscape StringApp: Network Analysis and Visualization of Proteomics Data),更适合用于展示大网络。并且在网络分析,数据导入与可视化方面有更大的灵活性,为科研人员提供更加自主化个性化的分析工具。下面小编特地为做蛋白质组学数据分析的老师梳理了StringApp的用法指南。
StringApp优势
● 可展示更大的网络系统;
● 数据导入分析,可视化更灵活;
● 可与Cytoscape软件中的其他功能插件结合使用,同一数据多层次分析;
● 可集成String数据库创建一个工作流;
● 免费使用;
操作流程
1.安装string APP:打开Cytoscape,点击菜单栏中的Apps--选择App Manage--在搜索框中输入“String APP”,安装该插件。


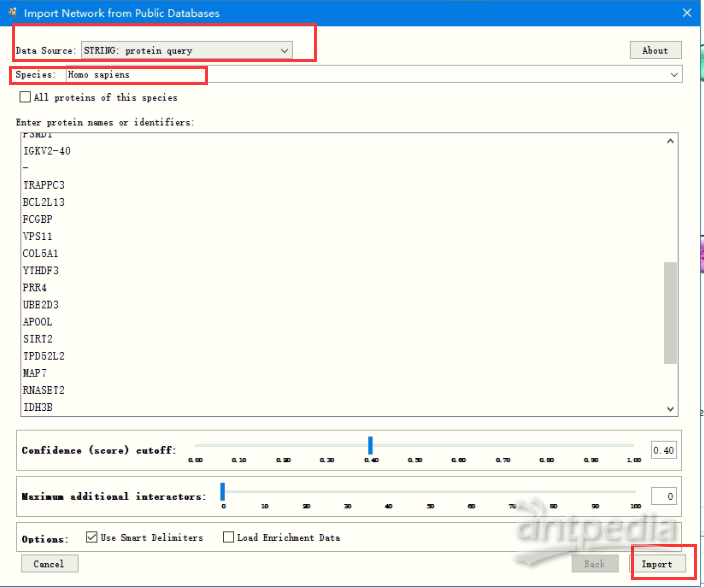
2. 导入互作网络数据库:安装完成后,导入互作网络数据库。选择菜单栏中的File--Import--Network--Public database。

3. 属性选择:弹出的属性选择框中,Data Source = String:protein query,输入要分析的蛋白/基因名。根据需要选择Confidence Score,代表蛋白间相互作用强弱的值,Cut-off值设定默认0.4(medium)。这样就会得到一个初步的蛋白网络互作图。
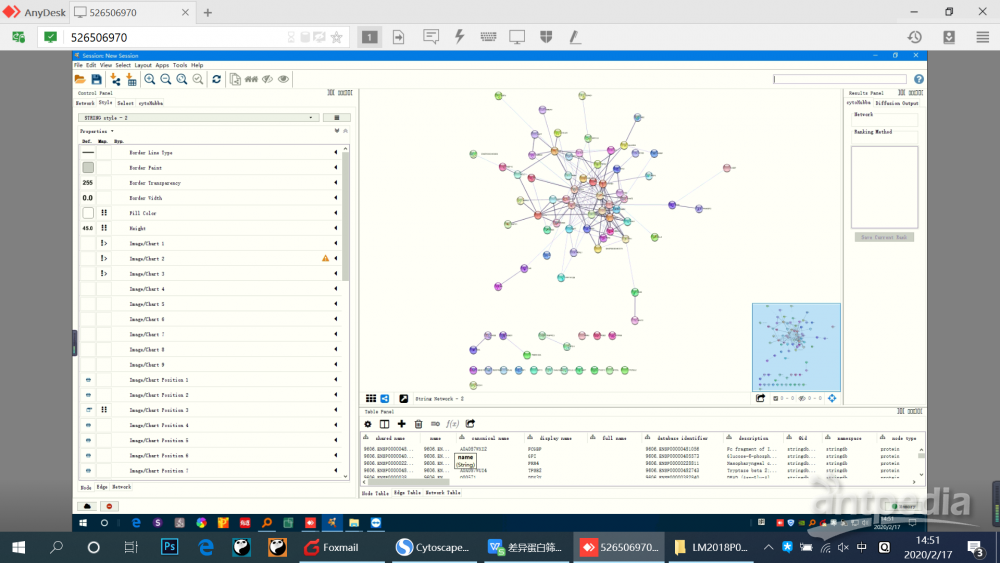
4. 个性化调整:对初始图进行个性化调整,应用到左边工具栏中string style工具栏下节点,线的多种属性选项。

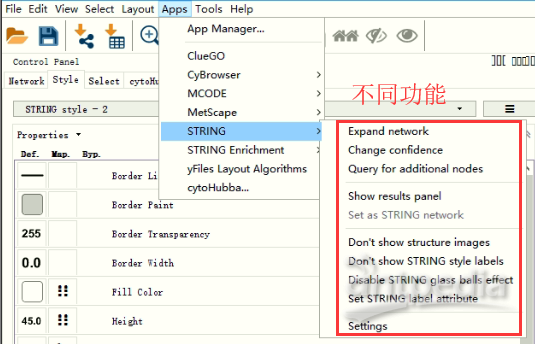
5. 内容更改:如对网络图的内容进行更改,点击菜单栏Apps--String下修改。
6. 通路富集分析:值得推荐的是KEGG通路富集功能,在菜单栏Apps下还有一个被自动安装的STRING Enrichment插件,该插件的功能主要是对互作网络图中蛋白进行通路富集分析。设定FDR value cutoff值后(默认为0.05),即可得到目标蛋白/基因的KEGG富集分析结果。


接着,我们可以选中自己感兴趣的一条通路,网络图中显示黄色的点就是该通路的蛋白。然后就可以对这条通路下的蛋白显示风格进行个性化调整,如形状、颜色、大小等。
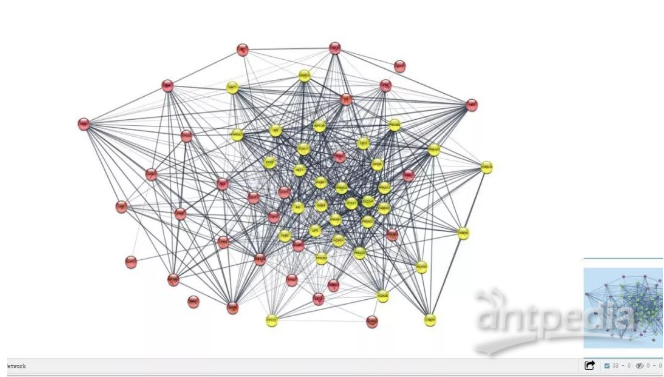
众所周知,蛋白质是细胞功能的主要执行者。目前随着可获得的大规模蛋白质相互作用数据的迅速增长,研究者可以从系统水平上整体的对细胞生命活动的基本组件和结构进行研究、认识和理解。但是,随之而来,分析此类复杂的时空相互作用数据来反映细胞、组织、生命活动以及其功能成为一大挑战。本文推荐的这款Cytoscape插件:StringApp使得研究蛋白之间相互作用及功能变得容易起来!相信大家看完本篇应用指南也能从实操上具体落地画出蛋白互作图啦~~
掌握新技能后,赶快实际操练一下吧!
猜你还想看
◆组学研究,R语言实用技巧—热图,运用pheatmap包简单易懂快速汇图方法来袭~