河北工业大学李建伟等开发强大miRNA-疾病关联预测工具
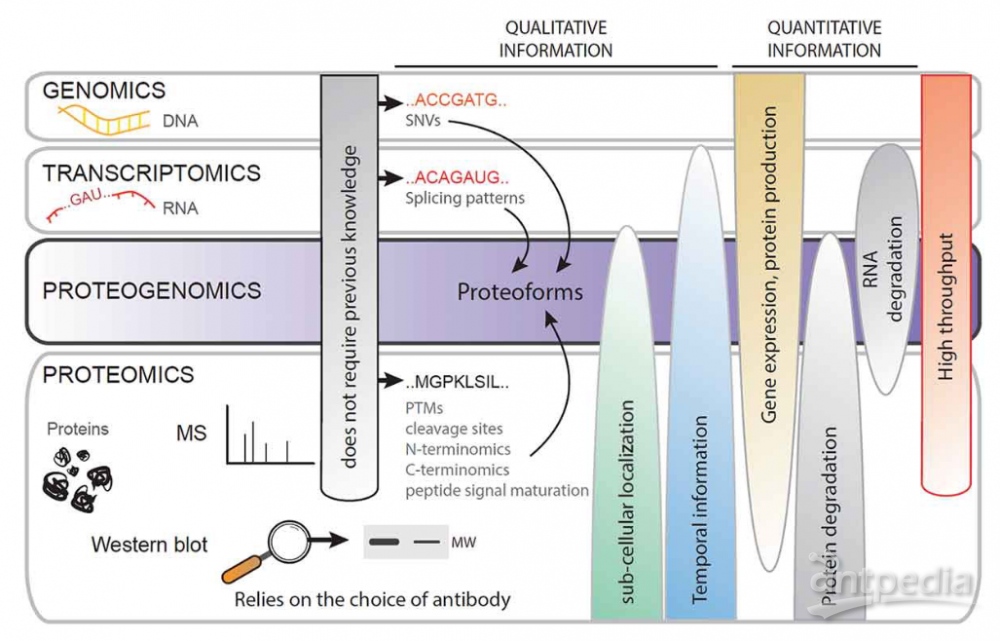
MicroRNA(miRNA)是22nt左右的RNA,主要通过靶向mRNA的3'UTR区来调节基因表达。这些小的非编码RNA广泛参与重要的生物学过程,例如细胞分裂,分化,凋亡,细胞周期调节,炎症和应激反应。因此,miRNA的失调,包括表达失调,功能获得或丧失的突变以及表观遗传沉默,通常在许多疾病的发作和发展中起重要作用,包括但不限于癌症,心血管疾病和神经退行性疾病疾病。迄今为止,有一些流行的miRNA疾病关联数据库,其中HMDD和miR2Disease手动从文献中挑选出已知的miRNA疾病关联,而dbDEMC通过在观察到的疾病(癌症)中鉴定差异表达的miRNA来推断miRNA疾病关联。这些数据库不仅可以用于生物医学科学家了解miRNA在疾病中的作用,还可以用于生物信息学开发人员建立新颖的miRNA-疾病关联预测工具。
确实,鉴于仍然有很大一部分潜在的miRNA-疾病关联尚待探索,因此计算方法构成了实验分析的必要补充。例如,最新的miRBase记录了1917个人类miRNA基因,而根据当前的DO命名法则有9000多个疾病术语。相比之下,HMDD v3.1是目前最新的miRNA-疾病关联数据集,仅涵盖893种疾病和1206个miRNA基因之间的35547个miRNA-疾病关联。这些统计数据表明,尚未通过实验研究报道约30%和约80%的人类miRNA和疾病。考虑到实验的时间和人工成本,有效和准确的计算预测工具是必要的,并保证社区筛选主要目标进行进一步研究。

基准数据集上36种miRNA-疾病关联预测因子的整体表现
在这里,基于来自最新HMDD v3.1数据库的8000多种新颖的miRNA疾病关联,研究人员对36种容易获得的预测方法进行了系统比较。通过严格的精确召回曲线分析对它们的整体性能进行了评估,其中有13种方法显示出可接受的准确性(AUPRC> 000.200),而前两种方法的AUPRC则有希望达到0.300以上,并且在仅考虑因果关系时,其中大多数方法的排名也很高miRNA疾病关联为阳性样品。通过组合不同的预测变量或采用更新的miRNA相似度矩阵证明了性能提高的潜力,与最佳的单个预测变量和使用先前相似性矩阵的预测变量相比,分别可提高AUPRC的16%和46% 。该研究分析提出了可用方法的一个普遍问题,即预测结果严重偏向于带有许多已知miRNA的注释良好的疾病,并且无法通过与一般miRNA-区别因果性miRNA-疾病关联来进一步对阳性样本进行分层。
总之,该基准测试结果不仅为生物医学研究人员选择合适的miRNA-疾病关联预测因子提供了参考,还为开发更强大的miRNA-疾病关联预测因子提供了未来的方向。
-
仪器推荐

-
仪器推荐

-
仪器推荐

-
仪器推荐
 询底价 Tel:400-6699-117 转 5224
询底价 Tel:400-6699-117 转 5224 -
仪器推荐
-
科技前沿

-
焦点事件

-
焦点事件