分子对接计算中如何确定对接口袋?(一)
在一般的分子对接计算中,一个不可或缺的步骤是定义配体分子(通常为有机小分子)的结合位置,即对接口袋。对于蛋白-小分子复合物X-ray晶体结构,口袋内就有一个配体,它为我们指示了对接口袋的位置。但还有很多X-ray晶体结构、NMR解析的结构没有配体结构,我们该如何确定对接口袋呢?更一般地,对于核酸、多肽以及主客体中的主体分子,又该如何定义对接口袋呢?
对接口袋这个概念存在于分子对接计算中,是受体中配体结合的可能区域。通过设定足够大的盒子把口袋囊括起来来告知对接程序它的位置。如果对接口袋被设定在真正的活性结合位点上,则有更大概率找到配体正确的活性构象与结合模式。对接口袋,顾名思义,通常呈口袋状(开口小、肚子大、能容纳一定体积的分子结构),也有其他形状,比如管道状、凹槽状和浅洼状,而以口袋形状最为典型。对于蛋白-配体复合物而言,大且深的疏水性空腔对于配体结合至关重要。对于蛋白结构,这一特点便成为各种算法寻找对接口袋/识别结合位点的重要依据和原则。
识别蛋白口袋/配体结合位点的方法
下面针对蛋白讲述确定对接口袋的常用策略。由于核酸、多肽以及主体分子的口袋特点千差万别,难以一概而论,但若能灵活变通,这些策略也经常适用,甚至操作上更为简单。
一、 文献/数据库调研法
最可靠的信息,莫过于实验数据。我们可通过文献调研,从他人/前人的实验结果中获悉该蛋白的主要功能和所属家族,找到它的活性位点信息。比如,胰凝乳蛋白酶的His-Asp-Ser催化三联体、锌指蛋白的Zn2+结合位点。通过对已有较多研究的其他种属的同源蛋白进行比较研究,找到对应的口袋信息。UniprotKB数据库(https://www.uniprot.org/)整合了丰富的蛋白结构-功能信息,有的还包含突变位点信息。这为我们识别蛋白口袋/配体结合位点提供可靠的依据。
二、 实验筛查法
如果文献和数据库上缺少有用信息,而实验手段又相当方便且便宜时,那就自己做实验吧,比如定点突变(通常用在对计算结果的验证,而非相反)、荧光探针标记[1]。
三、 软件预测法
这是最简单、最直接的办法。目前已有众多软件/算法可以帮助人们预测蛋白的活性口袋或者识别配体的结合位点。下图总结了部分预测程序的基本情况。
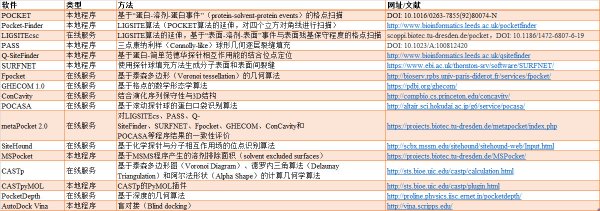
(蛋白口袋/配体结合位点预测本地程序或在线服务,点击此处跳转相关链接页面)
各程序对输入内容的要求大同小异,输出结果却千差万别。感兴趣的读者,可进行一番探索。这里以在线服务POCASA为例,讲解如何预测蛋白受体1UWH的口袋。该晶体结构为蛋白-配体复合物,配体分子指示了结合位点,正好用来检验POCASA能否正确预测口袋位置。
1、登录POCASA网站:http://altair.sci.hokudai.ac.jp/g6/service/pocasa/
2、在PDB ID栏输入1uwh,其他参数保持默认值,点击Get Pockets and Cavities按钮
等待片刻即返回结果。Chain ID默认为NULL,表示选择蛋白文件的第一条链。该蛋白晶体结构包含A、B两条链,均有配体小分子,我们预测A链上的口袋位置。

(POCASA提交任务界面)