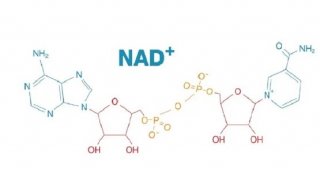
基于 Orbitrap GC-MS 的非靶向代谢组学(二)
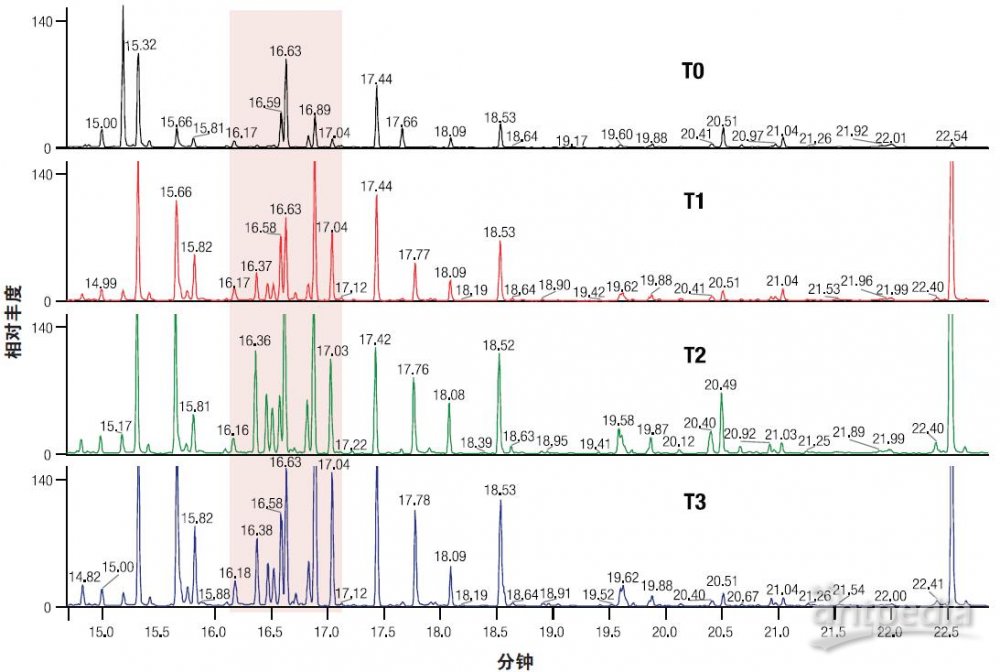
图 2. 大鼠肌肉组织样品代谢 0–3 天后的 GC-MS 色谱图(自上而下天数增加),X 轴为保留时间,Y 轴为强度(固定为1E9 计数)。例如,记下高亮区域中峰强度随时间的变化。
化合物发现阶段
Q Exactive GC 系统获取了每个样品的全扫描色谱图(图 2)。该系统在较宽的动态范围内采集不同浓度水平代谢物的全扫描色谱图,不会丢失任何精确质量数信息。使用自动峰选取(结合使用 XCMS 和 MzMatch.R)功能提取每个 EI 峰簇。在该步骤中,已检测到 1193 个明显峰簇并进行了初步定量,强度阈值为 100,000。已解卷积峰簇的示例见图 3。

图 3. 已解卷积峰簇被推断识别为酪氨酸。
定量阶段
首先使用单变量统计方法分析结果。使用 Student's t 检验对比每个时间点和时间零点。将每个代谢物的平均 T0 强度设为 1,倍数变化显示为 1 的倍数值,以方便对比强度明显不同的代谢物(图 4 和图 5)。已检测到 272 个代谢物的平均强度发生显著性变化(P 值小于 0.05),包含已检测峰簇的定量矩阵示例见表 3。
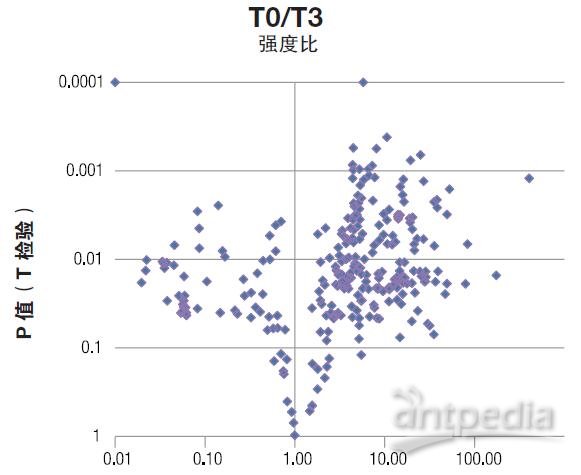
图 4. 发生显著性变化代谢物的火山图。每个代谢物以深蓝色菱形表示。强度比(X 轴)是指 T0/T3 时代谢物的倍数变化,Y 轴显示代谢物的 P 值。因此,右上方和左上角的代谢物是倍数变化最大且最具有统计学意义的代谢物。

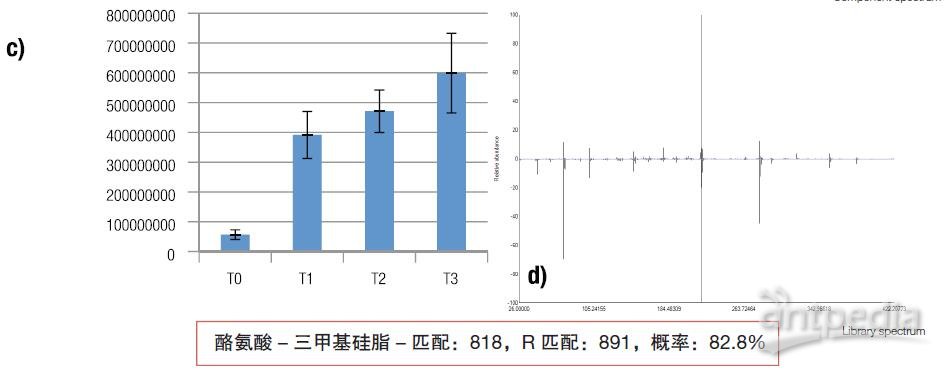
图 5. 数据分析流程的总结。对原始数据进行峰检测和解卷积(a),然后将其以基峰的形式显示在 IDEOM 代谢组学软件中(b),含定量信息和定量统计方法。IDEOM 软件也提供定量图形信息(c),该图显示了每种条件下的平均强度和标准偏差,本例中每种条件下含四份相同的生物样品。使用 NIST 库归属目标峰,(d)证实了质谱数据的匹配的结果,图下方含已归属衍生化氨基酸的Nist分数值。

表 3. 已检测峰簇的定量矩阵。基峰(峰簇中强度最大的峰)、保留时间(RT)和检测到的最大强度见第 1–3 列。将 T0 的强度归一化为 1,其他时间点的强度与 T0 强度相比并视情况以不同颜色显示,见第 4–7 列。第 8–11 列包含每项比值的 T 检验 P 值。
-
精英视角

-
焦点事件

-
精英视角