测序入门:接头家族大揭秘
在高通量测序技术日益成熟的今天,相信大家或多或少都对文库制备实验有所了解,而文库制备的目的,就是为了在目标DNA序列两端连接上特异的接头,从而能够在测序平台上机测序。那今天,我们就来扒一扒关于Illumina平台文库接头的那些事儿吧。

更多测序研究等你探索
接头结构
接头作为文库的必要组成部分,包括P5/P7、Index以及R1
SP/R2
SP序列。其中P5/P7序列能够跟测序芯片上的P5/P7序列互补和相同,只有这样才能将待测片段固定在Flowcell上进行桥式PCR扩增;Index又称为barcode,目的是给文库加上特定的标签,用于文库混合测序时区分不同的文库样本;R1
SP/R2
SP是Read1和Read2测序引物结合的区域,在dNTP和DNA聚合酶的作用下能够进行碱基的延伸。下图是接头的一般结构,呈“Y”字型。
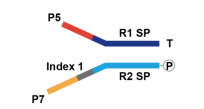
图1:接头的一般结构
“Y”型接头保证了每条单序列两端均为不同的测序引物,从而可以通过后续的PCR扩增形成两端带有不同核苷酸序列(P5/P7)的文库。
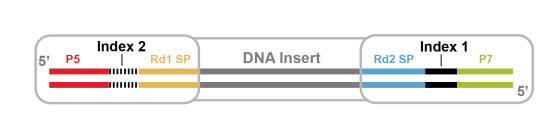
图2:文库的一般结构(中间灰色部分为待测序列)
接头如何分类
01
单/双端Index接头
根据Index位置的不同,接头可以分为单端和双端Index接头。单端Index接头只在P7端存在Index序列,双端Index接头在P5和P7两端均存在Index序列。一般而言,Index序列的碱基数为6
nt或8
nt。以8碱基Index序列为例,单端Index组合理论上可以有48种,双端Index组合理论上可以有416种。但是,考虑到测序时Index组合要满足碱基平衡和激光平衡原则,实际可供选择的Index种类为数不多。目前双端Index组合最多高达3840种,这直接影响最终文库混合上机能够混合的样本数。
目前,测序仪的测序能力远大于测试样本数据量,例如Hiseq、Novaseq等,为避免仪器浪费,就需要更多样的文库标签来区分不同的文库样本,因此双端Index接头的应用更为广泛。
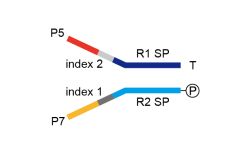
图3:双端Index结构
注:由于Illumina不同机型双Index
PE测序策略不同,i5端Index 2的读取方式也有所不同。如测序平台为Miniseq,Nextseq,Hiseq
3000/4000,Hiseq X等机型时,Index 2序列需要进行反向互补后再输入到sample sheet中。
02
长接头和短接头
根据能否匹配PCR-free文库,接头又可以分为长接头(完整的Y型)和短接头(不完整的Y型接头)。长接头通过TA连接的方式连接到待测DNA片段两端,在文库产量足够的情况下,可不进行PCR扩增直接上机测序;而短接头通过TA连接的方式连接到待测DNA片段两端后,必须使用与短接头互补的Indexing
Primers进行PCR扩增成为完整接头后,才能上机测序。造成上述区别的主要原因是长短接头引入Index序列的方式有所不同。
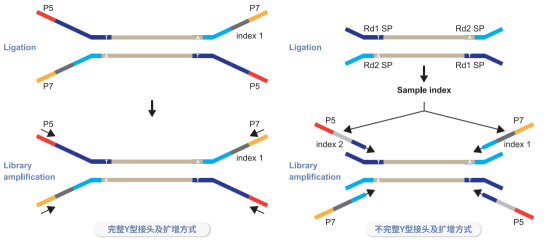
图4:长短接头Index序列不同引入方式
注:长接头连接产物扩增时,引物序列只需要与P5/P7互补即可,而短接头连接产物扩增时,引物序列还需要包含有Index。
03
UDI & UMI接头
2017年,Illumina公司推出了“测序洗衣机”型号——NovaSeq
6000,这款仪器的特点在于运用了ExAmp扩增方式和Patterned flow cell样品槽,2天能产出高达6
Tb的数据,为更大规模的全基因组测序带来了希望。新技术的应用虽然大大增加了扩增效率,使得通量更高、成本更低,但同时也加大了游离接头的干扰,导至出现标签跳跃(Index
hopping)问题。
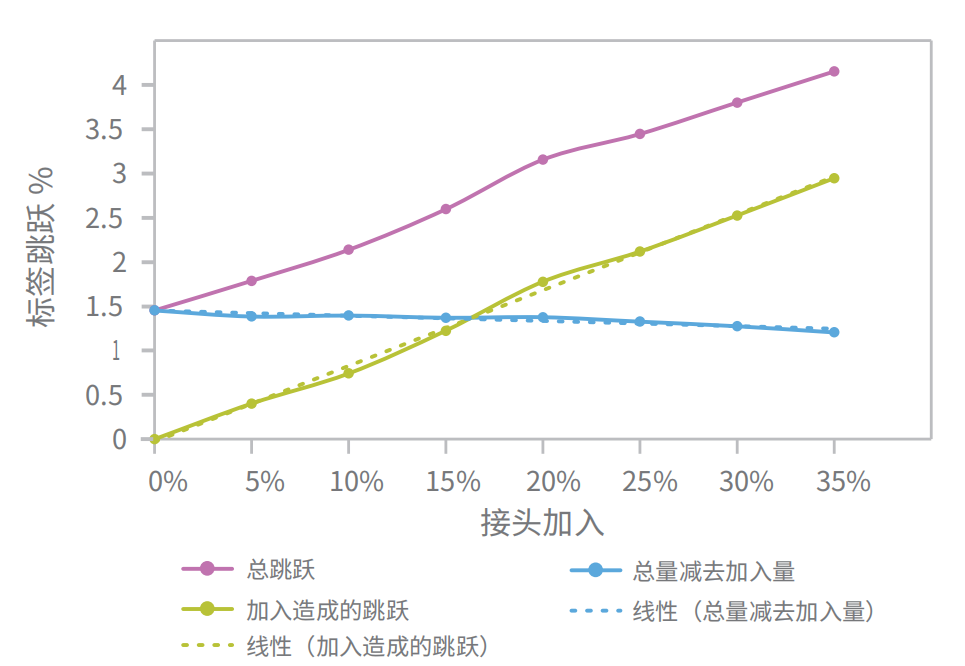
图5:标签跳跃百分比与加入的接头水平的曲线[1]
为了解决Patterned
Flow Cell测序平台(Hiseq系列、Novaseq等)凸显的标签跳跃问题,Illumina提出了“Prepare dual
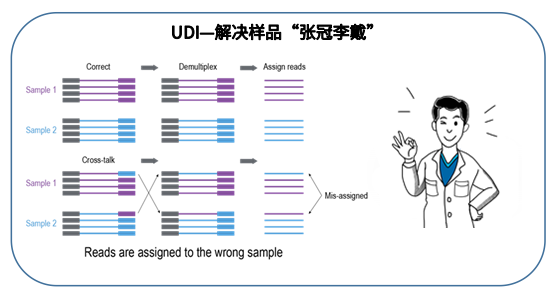
Indexed libraries with unique Indexes”的策略,即在文库的P5和P7端带上独特的标签—UDI(Unique
dual Index),通过P5 Index 2和P7 Index
1成组设计,两端Index交叉验证,从而能够有效降低Index错误分配的现象。
此外,在极低频突变检测中,避免假阳性是液体活检格外关心的重要一环。应用分子标签技术,能够分辨出检测到的突变是真实的低频突变还是来源于测序错误和PCR扩增错误,从而过滤掉背景噪音,实现低频突变的准确检测。
而分子标签UMI(Unique
Molecular
Identifiers)通常设计为完全随机的核苷酸链(如NNNNNN)、部分简并核苷酸链(如NNNRNYN)或者固定核苷酸链(当模板分子有限的情况下)。NGS文库构建过程中,在UDI接头结构的基础上引入UMI标签,通过对原始DNA分子片段进行标记,能够验证测序结果在原始DNA序列上的一致性,从而剔除假阳性突变。
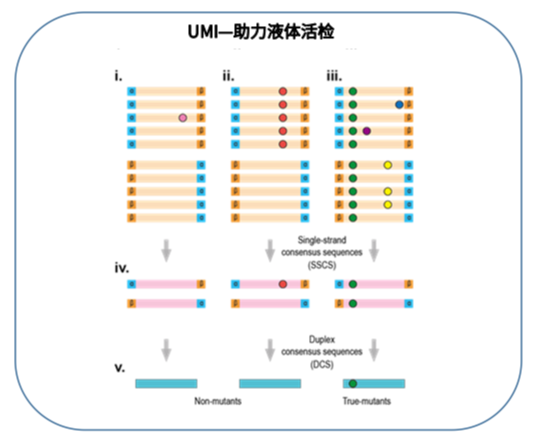
如上图所示,三个分子被测了5次,其中左侧和右侧均有测序错误,但该错误并没有在每个测序数据中出现,所以在后续合成一个分子的时候,测序错误被修正,只保留了真正的突变。
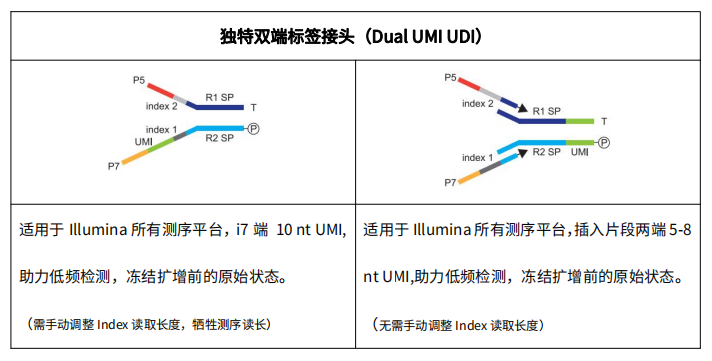
目前这种新型的UMI接头包含三种元件,即P5 Index、P7 Index以及UMI元件。根据UMI标签引入接头的位置—紧随P7端Index或位于插入片段两端,又分为两种不同的接头类型:
注1:转座酶文库不是通过TA连接的方式连接接头,其引入Index的方式与非完整接头类似,即转座酶切的过程中引入R1 SP和R2 SP,再经过PCR扩增引入Index和P5/P7序列,最终形成完整的文库。
注2:Small RNA文库亦不是通过TA连接的方式连接接头,而是将Small RNA的3’和5’端分别与通用接头连接,经过逆转录、PCR扩增得到Small RNA文库。
参考文献:
[1]
https://www.illumina.com/content/dam/illumina-marketing/documents/products/whitepapers/Index-hopping-white-paper-770-2017-004.pdf
-
仪器推荐

-
仪器推荐

-
仪器推荐
 询底价 Tel:400-6699-117 转 5942
询底价 Tel:400-6699-117 转 5942 -
仪器推荐

-
仪器推荐

-
焦点事件


























