Agilent SureSelectQXT WGS 文库制备的质量控制(二)
gDNA 的数量和完整性
SureSelectQXT WGS 方案需要高质量的 DNA 样品,以获得最佳性能和 gDNA 起始材料的精确定量结果。按照该方案使用配有 dsDNA BR 分析试剂盒的 Qubit 仪器进行系列定量分析。将相同的样品在 4200 TapeStation
系统和 NanoDrop 中均进行6次重复基因组 DNA ScreenTape 分析。图2显示了获自 4200 TapeStation
系统、Qubit 和 NanoDrop 的数据,说明了基因组 DNA ScreenTape 分析法适用于对 gDNA
起始材料进行定量分析。采用UV 光谱法测定 gDNA 所得的量常会过高,这是由于其他缓冲液成分在 UV 光谱中也可能有吸收[4]。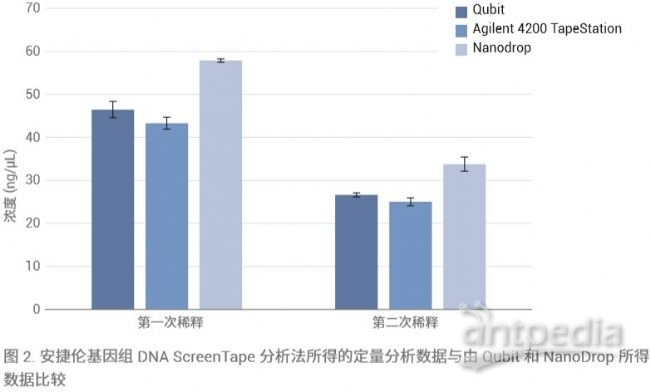
此外,基因组 DNA ScreenTape 分析法可在同一 QC 步骤中客观评价样品完整性。样品完整性由 TapeStation 分析软件的 DNA 完整值 (DIN) 计算功能自动测定(图 3)。
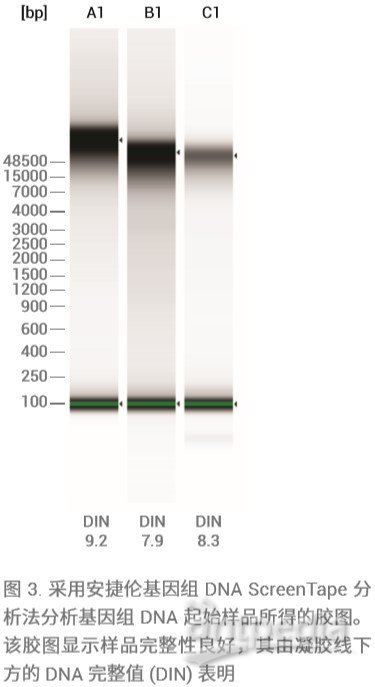
与其他系统不同的是,基因组 DNA ScreenTape 分析方法及 4200 TapeStation 系统只需 1 µL 样品即可在一步分析中同时对质量和数量进行评价。
Agilent SureSelectQXT 全基因组文库的片段分子量测定
除了 gDNA 起始材料的初始质量控制外,SureSelectQXT 方案还建议对扩增文库进行质量控制,从而确保在测序前显示整个片段的分子量范围。片段平均分子量显著小于 600 bp 可能表明碎裂反应中的 gDNA 过少,或者可能与测序数据的重复分析次数增加有关。相反,文库中片段平均分子量过大(超过 1000 bp)可能表明碎裂反应中的 gDNA 过多,并且可能需要更高的 DNA 浓度以在测序反应中获得最佳的群集密度。
为了证明这一点,制备起始量为 20、50 和 80 ng 的文库,用 50 ng gDNA 样品生成的文库作为标准文库。图 4显示了与 2100 生物分析仪系统相比,4200 TapeStation 系统在评估分子量测定方面的性能,通过用上述起始量对文库进行三次重复区域分析来评估。图中的数据展现了两个系统所得的文库分子量有很好的相关性。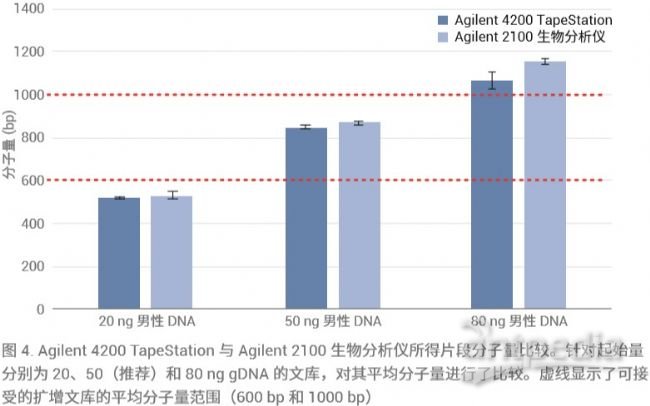
-
产品技术









