crystal数字PCR如何实现的核酸绝对定量?
crystal数字PCR技术是一种核酸分子的绝对定量技术,原理是将PCR反应体系分配到大量微小的反应器中,在每个微反应器中包含或不包含
1 个或多个拷贝的目标核酸分子 (DNA 模板) ,进行"单分子模板"PCR
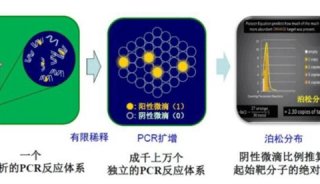
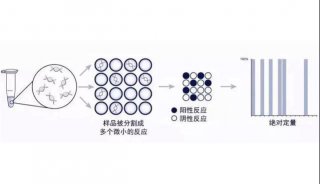
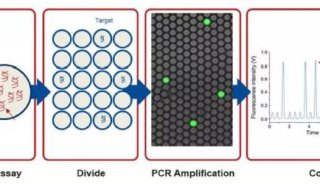
扩增。扩增结束后,通过阳性反应单元(通过终点荧光信号判断)的数目和统计学方法计算原始样本中靶基因的拷贝数。工作流程包含4个主要环节,即样本DNA/RNA的PCR/RT-PCR反应预混、反应预混液的分散或分区、PCR扩增、荧光信号的采集与数据分析(如下图所示)。
Crystal数字PCR将样本DNA分散到25000-30000万个微滴中,使每个微滴中包含或者不包含1个或多个拷贝的目标分子(DNA模板),所有微滴以2D阵列的形式单层随机平铺在sapphire芯片中,进行PCR扩增,扩增结束后读取各个反应单元的阴性或阳性荧光信号并进行统计学的分析,就可以计算出原始样本的模板拷贝数。那么我们如何保证每个微滴中只有一个DNA分子呢?
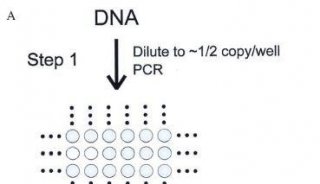
举例说明:将100个DNA分子分散到100个微滴里,理想情况下,我们希望100个DNA分子平均分布100个微滴中如下图中A图所示,但是实际情况是100个DNA分子以随机的状态分布在100个微滴中,如下图中B图所示。
A.理想情况下的平均分布
B.实际情况下的随机分布

图:DNA分子的平均分布(理想情况,A图)和随机分布(实际情况,B图)
如上由于目标DNA分子随机分布于阳性反应单元中,直接进行对阳性反应单元进行计数统计,不是真实的目标DNA分子拷贝数,每个反应单元中可能含有两个或两个以上的目标分子,这时可以使用泊松概率分布公式(1)进行计算。
 (1)
(1)
上述公式(1)中,λ为每个反应单元中含有的起始 DNA 分子平均拷贝数,p为在一定的λ条件下,每个反应单元中包含k拷贝目标分子的概率。λ由样品溶液的稀释系数m(或者分区数)决定,即λ=cm,其中c为样品的原始拷贝数。当k=0时,即不含目标DNA分子时,p即可为无荧光信号反应单元数与反应单元总数的比值,也就是阴性反应单元的比例,公式(1)可简化为公式(2):
 (2)
(2)
通过终点法检测,可以的到总反应单元数n和有荧光信号的阳性反应单元数f,所以阴性反应单元的比例即为公式(3)。
 (3)
(3)
因此得到下面等式:
 (4)
(4)
对上式两边同时以e为底取对数,得到下面等式:
 (5)
(5)
也就是说利用数字PCR方法进行核酸绝对定量时,只需要通过阴性反应单元的比例和样品的稀释系数(或分区数)即可确定反应单元的平均核酸拷贝数,从而实现DNA的精确定量。这就是数字PCR技术如何实现核酸绝对定量的本质。
泊松分布应用与数字PCR结果校正的前提是需要事件均匀一致,包含微滴的体积、几何参数、PCR反应条件。Crystal 数字PCR通过自动化微滴阵列的方式,实现了微滴的均匀一致和反应条件一致的同时,通过软件自动对微滴的几何参数进行质控,并可溯源到单个微滴,给出最可靠的数据结果。

微滴单点溯源