HiFi测序:单碱基分辨率的高通量端粒长度测量方法
端粒是线状染色体末端的特殊核蛋白结构,包含约5-15 kb的富含TTAGGG的双链重复序列和保护染色体末端的shelterin蛋白复合物,3'末端为富含G的单链悬突,对于维持人类基因组的稳定和完整复制至关重要。
端粒主要依赖端粒酶进行合成,长度也并非一成不变。除了生殖细胞和干细胞,体细胞由于端粒酶活性的缺乏会导致每次细胞分裂时端粒逐渐缩短(约50-200 bp)。研究表明外周血单核细胞(PBMCs)中与年龄相关的端粒生命早期每年缩短1190 bp,儿童期每年缩短126 bp,成年期每年缩短43 bp。随着年龄的增长,端粒的缩短从生命早期到成年期逐渐缓慢,当端粒缩到非常短时就会发生功能失调,诱发DNA损伤反应、导致细胞衰老并引发一系列疾病。因此正常人类体细胞中端粒长度的逐渐缩短成为年龄相关疾病的很有希望的生物标志物。但是目前多种端粒检测技术仍然达不到在单碱基分辨率的情况下实现端粒的高通量检测或者无法准确定量端粒的长度。因此,在实验和临床研究中都迫切需要一种能够及时纵向检测个体端粒磨损的高灵敏度方法。
在这里,新加坡国立大学Shang Li和Motomi Osato课题组在nature communications上发表了题为“High-throughput telomere length measurement at nucleotide resolution using the PacBio high fidelity sequencing platform”的文章,描述了使用新设计的端粒-DNA序列(称为"Telobaits")在细胞系和临床患者样本中捕获端粒的工作流程,并使用PacBio HiFi测序以单碱基分辨率实现端粒长度的高通量、准确测定。还揭示了分布在整个端粒重复序列区域的端粒变异序列(TVS)的极端异质性。TVS的存在破坏了典型端粒重复序列TTAGGG的连续性,从而影响了染色体末端shelterin蛋白复合物与端粒的结合和对端粒的保护。这些发现可能对人类衰老和疾病有深远的影响。

目前的技术限制
末端限制性片段(TRF)分析、实时定量聚合酶链反应(qPCR)、高通量单端粒长度分析(HT-STELA)和高通量荧光原位杂交(HT-QFISH)等只能提供端粒的平均长度或相对长度,缺乏单碱基的分辨率。最重要的是,它们缺乏检测可能对人类衰老和疾病有深远影响的端粒变异序列(TVS)的能力。
长读长测序平台可以实现端粒基因组DNA片段的直接测序,获得单个染色体末端的绝对端粒长度。但是目前的测序方法需要全基因组测序来分析个体的端粒分布,达不到对高通量的需求。而且无法保证末端3 '富含G的悬垂单链的完整性。
方法
为了克服这些问题,该研究做了以下优化:
1. 提取高分子量基因组DNA,使用RsaI和HinfI限制性内切酶进行酶切。
2. 设计了6个新的端粒-DNA序列(称为“Telobaits”),分别包含可与端粒3'单链悬突互补的重复序列、EcoRI限制性内切酶位点和3'生物素标记。
3. 用Telobaits捕获到染色体的末端,然后使用链霉亲和素偶联磁珠纯化完整的全长端粒基因组DNA片段,EcoRI限制性内切酶酶切后洗脱。
4. 在端粒中加入条形码进一步允许样品的混样来提高通量。
5. 使用PacBio HiFi测序平台在单碱基分辨率下进行高通量的端粒长度测量。

图1. 实验流程图
结果
1 PacBio HiFi测序对端粒重复序列DNA载体的测序准确性高
PacBio HiFi测序平台基于单分子实时(SMRT)测序实现了平均读长达10-25 kb、准确度90%>Q30。为了验证PacBio HiFi测序平台是否可以作为测量端粒长度的可靠工具,研究人员首先对含有拟南芥端粒5'TTTAGGG-3'重复序列的线性化DNA载体(pWY82)进行了HiFi测序。如图2a所示,研究人员获得了质粒的高精度测序数据。DNA载体的CCS reads的平均准确率中位数超过Q40(99.99%),平均值为Q29(99.87%)。随后,使用准确度Q20以上的HiFi reads进行比对分析,DNA载体非重复区域的比对精度接近100%,重复区域的比对精度>95%(图2b)。拟南芥端粒重复序列的单碱基水平的测序错误与质粒非重复区域的测序错误相似(图2c)。总体而言,非重复序列区的测序精度优于端粒重复序列区。因此,研究人员验证了PacBio HiFi测序是一种可靠的端粒DNA片段检测方法。
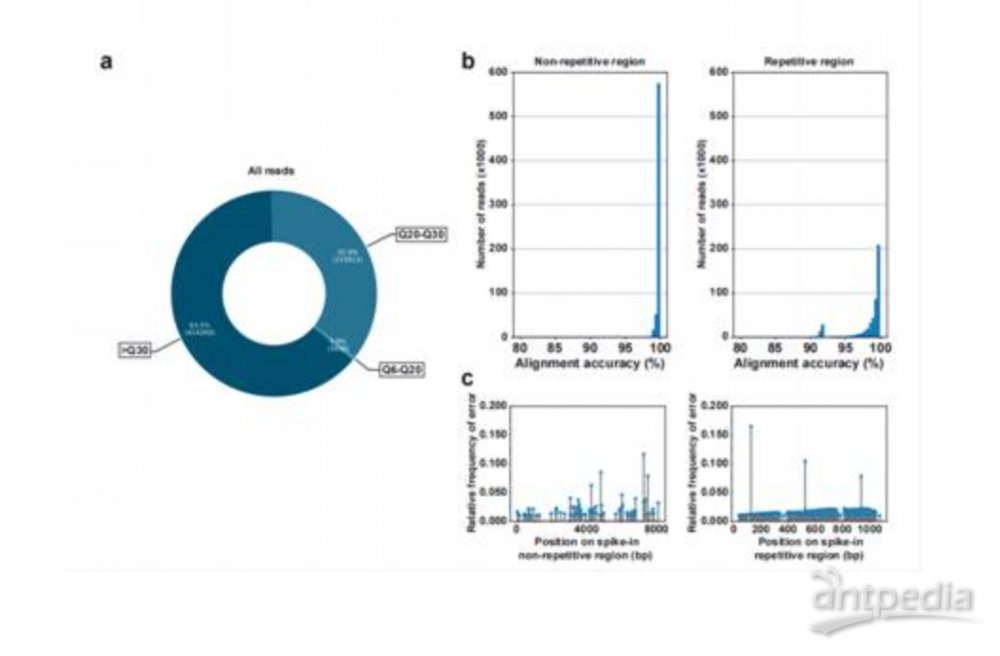
图2. 端粒重复序列DNA载体PacBio HiFi测序。
2 PacBio HiFi测序的单碱基分辨率实现端粒长度的准确测定
接下来,研究人员使用优化的方法从培养的细胞系中富集含有端粒的基因组DNA片段,使用PacBio HiFi测序进行端粒长度测量。
为了准确测定端粒的长度,研究人员重新定义了端粒长度:
1.将端粒的5'起始定义为包含至少两个连续的端粒重复序列TTTAGGG,因为shelterin蛋白复合物的最小结合基序包含1.5个典型重复序列TTTAGGG,端粒的3 '末端为3'单链悬突,这之间的距离被称为原始端粒长度(图3a),这与传统的端粒长度定义(从TTAGGG重复开始到染色体末端)相当。
2.考虑到shelterin蛋白复合物,是以序列特异性的方式与典型的端粒重复序列结合,预计会被TVS的存在破坏。因此,研究人员还计算了端粒中仅含有典型TTTAGGG的端粒长度,并将其定义为端粒长度(图3a)。
研究人员测定了从HCT116、293T、T24、IMR90 (PD47)和WI38 (PD40)细胞中提取的端粒DNA片段的绝对长度(图3b, c)。在单次测序运行或单独测序运行中,不同生物重复之间平均端粒长度与碱基分辨率显示出非常高的一致性,表明HiFi测序重复性好(图3d)。先前的研究表明,T24细胞含有极短的端粒。HiFi测序印证了这一结论,并进一步分析出T24细胞比其他细胞系具有更高比例的极短端粒(<1000 bp)(图3e, f)。使用PacBio HiFi测序测定的细胞系的原始端粒长度和平均端粒长度与使用TRF测定的端粒长度呈现一致性(图3g)。
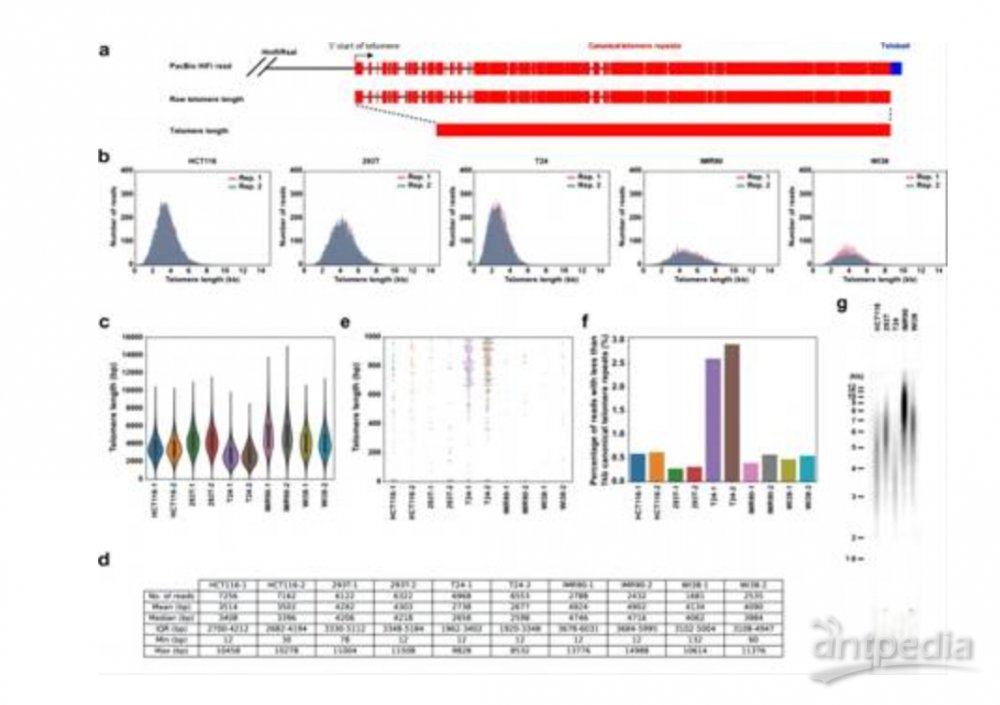
图3. 使用PacBio HiFi测序测定培养细胞HCT116、293T、T24、IMR90(PD47)和WI38(PD40)的端粒长度
为了摸索准确测量端粒长度所需的理想测序深度,研究人员将独立测序运行的HCT116和293T细胞的不同生物重复测序获得的reads结合起来进行bootstrap重采样,计算端粒长度测量平均值的标准误差(SE)。如图4b和c所示,测序深度为~20,000个端粒reads足以将平均值的预期误差降低到± 20nt以下,具有95%的置信度(1.96*SE)。因此,基于HiFi测序设计优化的端粒长度检测方法的灵敏度足以检测出成人端粒每年缩短约43 bp。

图4. PacBio HiFi测序测定端粒长度的理想测序深度。
3 PacBio HiFi测序的高通量端粒长度测定
外周血白细胞(pbl)或外周血单核细胞(pbmc)的端粒长度经常被用作衰老相关研究的生物标志物。为了应用PacBio HiFi测序进行高通量的端粒长度测量,研究人员用HiFi测序平台的两次运行混样测定了104例患者的PBL样本。拆分后每个患者样本获得了至少2000条HiFi reads。随后研究人员计算了每位患者的平均端粒长度或原始端粒长度,并检查了其与年龄的相关性(图5a, b)。与预期一致,端粒长度或原始端粒长度与年龄的增加呈负相关。这些结果表明PacBio HiFi测序是一种可靠的方法,可以在单碱基分辨率水平上评估端粒长度。

图5. 患者PBL样品中的高通量端粒长度测定。
4 人端粒G链分析
研究人员的长读长端粒测序方法也允许准确分析染色体末端的G链。早期研究表明,哺乳动物端粒中的G链分析不如四膜虫精确。而这项研究从患者样本和培养细胞中获得的单碱基准确率的PacBio HiFi测序结果足以分析G链的末端组成(图5c)。因此,分析表明人类端粒G链的加工过程与在四膜虫和游仆虫中观察到的非常相似,超过60%的G链以GGTTAG结尾(图5c)。
5 TVS在端粒区的独特分布和序列
与已有的研究一致,HiFi数据显示在端粒重复区域存在TVS,并且它们在长度和序列上表现出非常高的异质性(图6a),丰度和大小分布也存在非常高的个体特异质性(图7a)。并且患者PBL样本中的TVS,长度分布从1到6个核苷酸不等,也并非随机分布(图6b)。以Chr.7q为例,在端粒重复区域的亚端粒末端附近,主要包含更大、更丰富的TVS(图7b)。

图6. 患者PBL样本中端粒变异序列(TVS)的独特长度分布。
6 TVS破坏了端粒上shelterin蛋白复合物的结合
在人体细胞中,Shelterin复合物由六种蛋白质组成,包括TRF1、TRF2、POT1、RAP1、TIN2和TPP1。典型端粒序列的突变会显著降低TRF1和TRF2的结合亲和力,从而损害了染色体末端保护。因此,当端粒变得非常短时,亚端粒区域附近更大、更丰富的TVS的存在预示着较低的shelterin蛋白结合密度,这可能导致生命后期端粒保护的急剧恶化。
为了预示这种现象,研究人员计算了每个患者样本中所有染色体末端靠近亚端粒区域的典型端粒重复序列的前1kb内TRF1/TRF2结合基序的最大数量。如图7d所示,研究人员在患者样本中观察到该区域TRF1/TRF2结合密度的显著差异。进一步观察单个染色体的末端,研究人员注意到来自患者样本P14和P15的Chr.7q,由于TVS的存在,尽管典型端粒重复长度相同,均为1 kb,但两条染色体末端具有不同的原始端粒长度(1126 bp vs 1627 bp)和TRF1/TRF2结合位点数量(81 vs 62)(图7e)。即使仅在染色体一端,TRF1/TRF2结合密度的降低预示着shelterin蛋白复合物对端粒的保护较低,从而增加端粒脱帽的可能性,并在端粒变得非常短的情况下诱导DNA损伤反应,特别是在生命的后期可能对人类衰老和疾病产生深远的负面影响。
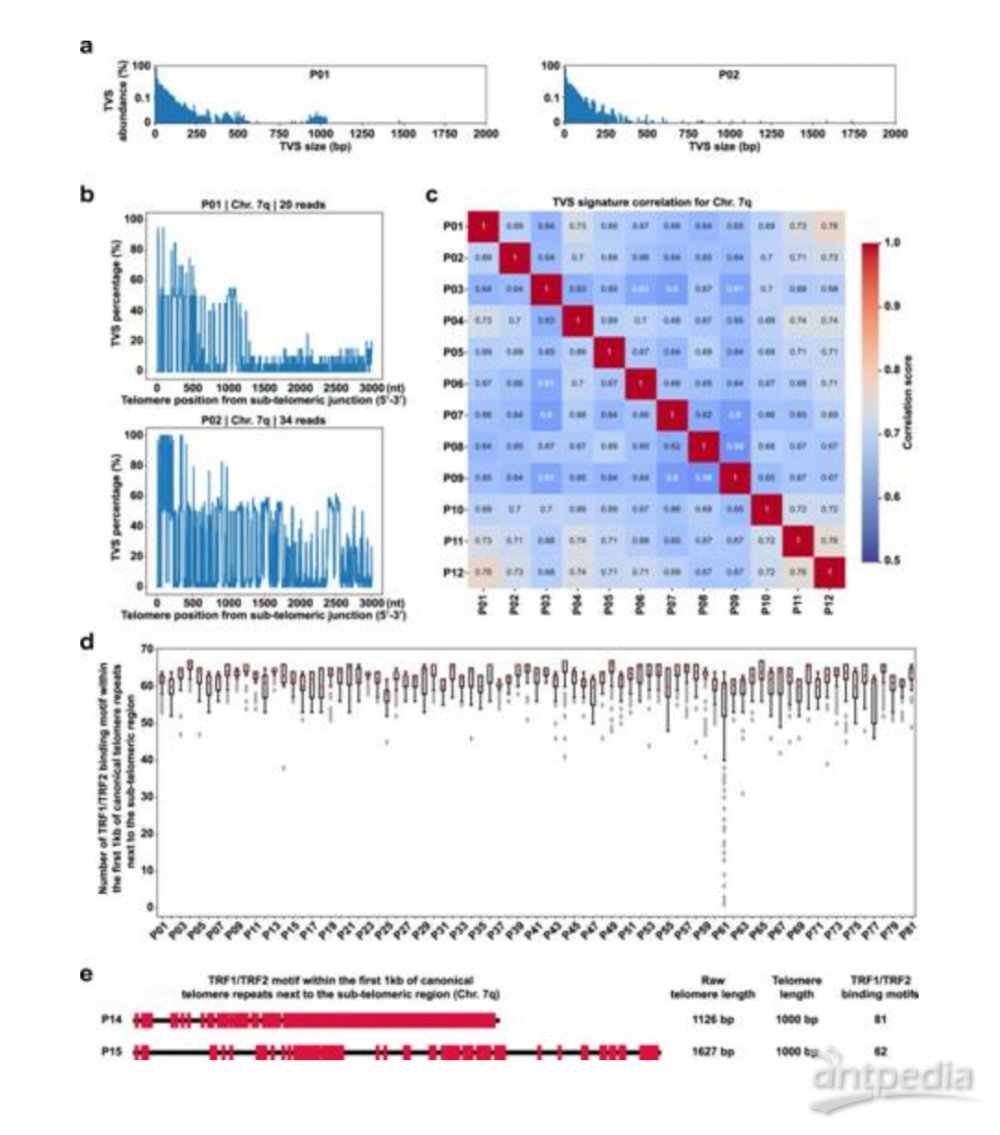
图7. 个体特异性的TVS的存在可能影响不同人的染色体末端保护。
讨论
在这项研究中,研究人员提出了一种利用PacBio HiFi测序平台富集含端粒基因组DNA片段进行单分子实时测序的高通量方法,并证明了长(10-25 kb)而准(90% > Q30)的PacBio HiFi测序是一种可靠的方法,可以在单碱基分辨率的水平上评估端粒的绝对长度,并且足以检测成人端粒每年缩短约43 bp的速度。还表明了不仅端粒绝对长度,典型端粒重复序列的连续性也是预测一般人群寿命和与衰老相关疾病的关键参数。
综上,端粒长度变化可能是一种重要的生物标志物,该方法检测端粒长度微小变化的灵敏度不仅可以使其成为估计端粒长度变化的重要工具,而且还可以用于开发潜在的干预措施,以减缓未来的人口老龄化。