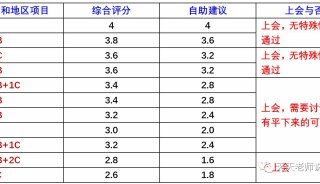
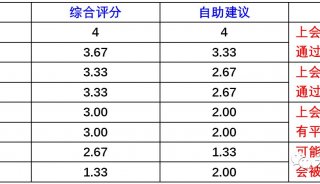
利用 TSQ Quantiva 定量肉类掺假比例(三)
4.利用 QED 进行定性确证
QED(Qualitative Enhanced Data)扫描功能是指通过监测 SRM 实时采集信号,利用二级子离子强度触发,采集对应母离子二级碎裂全扫描谱图,在定量的同时,获得更确切的定性信息的功能。通过 QED 技术,我们采集了 Pep003 的 QED 质谱图,图 4 为理论多肽 Pep003 实际谱图与理论 b、y 离子匹配结果:

图 4. Pep003 的 QED 全扫描子离子图
讨论
1.监测的专属性多肽的数目
在发现性研究中,文献中报导的 6 条猪专属性多肽 [4-6],我们只找到了其中的一条,还有两条是单氨基酸突变,另外三条没有找到;文献中报导的 21 条鸡专属性多肽我们找到了 17 条 [6, 7],其中有一条文献报道有乙酰化修饰,而我们检索过程没有加这个修饰。现代家猪由野猪驯化而来,经过了约 9000 年的长期的驯化与饲养过程中,亚种间和亚种内核型都发生了很大的差异,不同的地域或者品系的猪在蛋白表达上都有极大的差异 [8];而单氨基酸多态性(Single amino acid polymorphism,SAP)是自然界普遍存在的现象,地域和个体差异都能造成这种结果。基于上面的这些信息,由于动物基因型的差别,以及样品处理过程带来的影响,每一个物种仅选择一条多肽进行定量,很有可能造成大面积的假阴性的检出结果,有必要提高每个物种监测的多肽的数目,降低假阴性的概率。
2.可以利用一些亲缘关系相近的物种之间共有的多肽,实现同时筛选的目的
鸡与鸭作为常见的肉食性禽类,相对其他三种肉类来说,亲缘关系要更近一些,而且我们确实发现,它们共有的特异性多肽的条数(336条)甚至大于我们找到的鸭特异性多肽的条数(125条)。通过这种方式,还能够部分的弥补很多物种蛋白数据库不完善的情况(鸭的蛋白数据库就不全)。同时,鸡鸭同时共有而又相对兽类特异的多肽,往往也增加了其他禽类共有的可能性,一定程度上更经济的部分实现一些未知禽类物种添加的可能性。
结论
我们基于赛默飞全方位的质谱解决方案,建立了从掺假的发现,到定量多肽的选择,以及定量的实现的完整流程,并对发现的部分多肽进行了掺假实验与定量能力考察。基于实验结果,对于每一个物种,我们应该同时监测尽可能多的多肽,尽量避免假阴性的结果。我们同时选取鸡和鸭的六条特征性多肽,同时对两种禽类肉掺假进行了监测,并确定了最低的掺假监测限为 0.5%。考虑到掺假比例的经济性与可操作性,远远超过了实际监测的需求。
与传统的基于 PCR 和抗体的检测方法相比,质谱具有大致相当的灵敏度,但是拥有更好的避免假阴性与假阳性的结果的能力,且能够避免由于加工所带来的 PCR 或者抗体相关的空间结构破坏所带来的影响。
与上述掺假相比,还有一种相对来说,更为严重,性质更恶劣的掺假——病死肉的掺假。基于上述的思路,我们认为,通过进一步系统的研究,质谱也能够成为一种监测病死肉的手段,斩断病死肉流入餐桌的魔爪,与我们全方位的农残筛查与检测手段一起,为食品安全提供全方位的保障。同时,利用这种研究方法,我们还能助力有机肉类产品生产商,提供从肉类良种选择依据到肉类质量标准建立的可能性。
参考文献:
1.Nakyinsige, K., Y.B. Man, and A.Q. Sazili, Halal authenticity issues in meat and meat products. Meat Sci, 2012. 91(3): p. 207-14.
2.Sentandreu, M.A. and E. Sentandreu, Peptide biomarkers as a way to determine meat authenticity. Meat Sci, 2011. 89(3): p. 280-5.
3.http://www.fao.org/ag/againfo/themes/en/meat/background.html.
4.von Bargen, C., et al., New sensitive high-performance liquid chromatography-tandem mass spectrometry method for the detection of horse and pork in halal beef. J Agric Food Chem, 2013. 61(49): p. 11986-94.
5.von Bargen, C., J. Brockmeyer, and H.U. Humpf, Meat authentication: a new HPLC-MS/MS based method for the fast and sensitive detection of horse and pork in highly processed food. J Agric Food Chem,2014. 62(39): p. 9428-35.
6.Montowska, M., et al., Rapid detection of peptide markers for authentication purposes in raw and cooked meat using ambient liquid extraction surface analysis mass spectrometry. Anal Chem, 2014. 86(20): p. 10257-65.
7.Sentandreu, M.A., et al., A proteomic-based approach for detection of chicken in meat mixes. J Proteome Res, 2010. 9(7): p. 3374-83.
8.Ramos-Onsins, S.E., et al., Mining the pig genome to investigate the domestication process. Heredity (Edinb), 2014. 113(6): p. 471-84.
-
企业风采