产品介绍|蛋白质组学高质量标准品

293T细胞是在293细胞的基础上通过转染携带T抗原的质粒得到的细胞系,其主要特点是在转染过程中易于产生高效的转染,因此广泛应用于基因转染和表达实验中。293T蛋白消化标准品是一种经过高度验证的哺乳动物蛋白消化物,可用作复杂蛋白质组学样品的质谱 (MS) 分析的质控样品。在分析复杂样品之前经常采用消化物标准品可随时间监测和标准化样品间的 LC/MS 性能。
氨探生物出品了293T蛋白消化标准品,该蛋白消化标准品是一种冻干的胰蛋白酶肽混合物,可用作液相色谱(LC)分离、MS方法开发和MS性能基准测试的质量控制标准。该消化物专为LC/MS 实验而配制,不含盐或去污剂。
• 阳性对照: 人肾上皮细胞系的蛋白质酶切产物,总蛋白质数超过15,000种;
• 彻底酶切: 使用LysC和Trypsin制备,以确保漏切肽段比例小于10%;
• 经验证的肽质量: 低于10%的蛋氨酸氧化和赖氨酸氨甲酰化;
• 严格检测: 高质量、一致的蛋白酶切,批次特定的检验报告;
• 稳定: 以稳定的冻干形式提供。

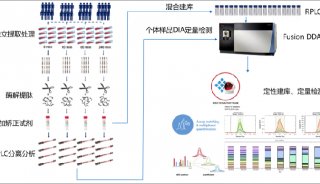
图1. 实验步骤流程图
• 质谱检测条件
• 搜库参数
• 质控参数
浓度 1µg/µL | 浓度=1.0 µg/µL | pass | |
LC-MS TIC图 | LC-MS图与下图对照一致 | LC-MS图与下图对照一致 | pass |
肽段鉴定数目 | 肽段数目 ≥ 18000 | 肽段数目 = 21598 | pass |
蛋白鉴定数目 | 蛋白数目 ≥ 3800 | 蛋白数目 = 4366 | pass |
全酶切肽段比例 | 全胰蛋白酶切≥ 85 % | 全胰蛋白酶切= 92% | pass |
Cys还原烷基化肽段比例 | 半胱氨酸还原烷基化 ≥ 12% | 半胱氨酸还原烷基化= 15.96% | pass |
Oxidiation肽段比例 | 甲硫氨酸氧化 ≤ 10 % | 甲硫氨酸氧化= 2.07% | pass |
Deamidation肽段比例 | 脱酰胺 ≤ 5 % | 天冬酰胺氧化 = 0.91% | pass |
* Peptide missed cleavage, alkylation, oxidation and modification determined by Preview™ Software (Protein Metrics) using a human protein Swiss-Prot database.

图2. 293T蛋白质酶切肽段在Q Exactive HF-X 质谱仪上经过60 min鉴定得到的Base peak
氨探生物出品的293T蛋白消化标准品,基于Q Exactive HF-X质谱,进样500 ng,选用数据依赖型采集(DDA)方式60分钟梯度采集数据,运用人类Swissport蛋白数据库通过Proteome Discoverer v2.4 (Thermo Scientific, USA)获取肽段和蛋白含量数据,鉴定到21,598个肽段,4366个蛋白,且胰蛋白酶肽漏切比例小于10%,同时非特异性修饰较少。
293T细胞裂解物通过LysC和胰蛋白酶进行酶切,可以尽量减少胰蛋白酶缺失裂解并改善蛋白序列覆盖率。此外,与用于质谱的其他市售蛋白消化标准品不同,293T蛋白消化标准品可满足严格的质量检测规范,包括肽质量、酶切效率和消化物批次间的均匀性。
若您对我们的产品感兴趣,或有宝贵的意见和建议,请联系我们:info@untangledbio.com。
1.Paulo JA, O''Connell JD, Gygi SP.A Triple Knockout (TKO) Proteomics Standard for Diagnosing Ion Interference in Isobaric Labeling Experiments.J Am Soc Mass Spectrom.2016 10:1620-5.
2.Nagaraj, N.; Wisniewski, J. R.; Geiger, T.; Cox, J.; Kircher, M.; Kelso, J.; Paabo, S.; Mann, M., Deep proteome and transcriptome mapping of a human cancer cell line. Mol Syst Biol 2011, 7, 548.
3.Kelstrup, C. D.; Jersie-Christensen, R. R.; Batth, T. S.; Arrey, T. N.; Kuehn, A.; Kellmann, M.; Olsen, J. V., Rapid and Deep Proteomes by Faster Sequencing on a Benchtop Quadrupole Ultra-High-Field Orbitrap Mass Spectrometer. J Proteome Res 2014.
4.Rosenberger, G.; Koh, C. C.; Guo, T.; Röst, H. L.; Kouvonen, P.; Collins, B. C.; Heusel, M.; Liu, Y.; Caron, E.; Vichalkovski, A.; Faini, M.; Schubert, O. T.; Faridi, P.; Ebhardt, H. A.; Matondo, M.; Lam, H.; Bader, S. L.; Campbell, D. S.; Deutsch, E. W.; Moritz, R. L.; Tate, S.; Aebersold, R., A repository of assays to quantify 10,000 human proteins by SWATH-MS. Scientific Data 2014, 1, 140031.
解构健康奥秘、探寻生命答案,氨探生物以一流的分子表型组平台和成熟的临床转化应用体系,为优秀的研究团队进行技术和数据赋能,致力于实现分子表型水平的精准诊疗。