5分钟了解蛋白质组学 基础一览
1、什么是蛋白质组?
蛋白质是由称为氨基酸的构建块组成的生物分子。蛋白质是生命所必需的,具有结构、代谢、运输、免疫、信号和调节等许多作用[1]。
术语“蛋白质组”是由澳大利亚博士生Marc Wilkins于1994年在意大利锡耶纳举行的研讨会上提出的[2]。它是一个概括性的术语,指的是一个生物体可以表达的所有蛋白质。每个物种都有自己的、独特的蛋白质组。
与基因组(每个生物体内的全套基因)不同,蛋白质组的组成随着时间和整个生物体的变化而不断变化[3]。因此,当科学家提到蛋白质组时,他们有时也是指某一特定时间点的蛋白质组(如胚胎与成熟生物体),或指生物体内某一特定细胞类型或组织的蛋白质组。

图1 | 人类基因组中大约有2万个基因,人类转录组中大约有10万个转录物,人类蛋白质组中超过100万个蛋白质形态
2、什么是蛋白质组学?
蛋白质组学是对蛋白质组的研究,研究不同的蛋白质之间如何相互作用以及它们在生物体内发挥的作用[4]。
虽然蛋白质的表达可以通过研究mRNA的表达来推断,mRNA是基因和蛋白质之间的中间人,但mRNA的表达水平并不总是与蛋白质的表达水平有很好的相关性[1,3]。此外,对mRNA的研究并不考虑蛋白质的翻译后修饰、裂解、复合物的形成和定位,或可以产生的许多变体mRNA转录本;所有这些都是蛋白质功能的关键。
1975年,随着二维蛋白电泳(2D PAGE)技术的发展,第一批符合“蛋白质组学”研究标签的实验开始进行[5]。
然而,只有在20多年后,随着质谱技术的发展,才有可能对每个样品的多个蛋白质进行真正的高通量鉴定[6]。
从那时起,质谱的灵敏度和准确性已经发展到可以可靠地检测到低至阿托摩尔(atto mol, amol)范围的蛋白质(每10^18个分子中有1个目标蛋白分子)[7],而且其他各种蛋白质组学技术也得到了发展和优化。
3、蛋白质组学可以回答哪些关键问题?
广义上讲,蛋白质组学研究在蛋白质水平上为健康和疾病的细胞过程提供了一个全局的视角[3,4]。为此,每项蛋白质组学研究通常都会在目标生物体的蛋白质组中一次集中研究以下一个或多个方面,以慢慢建立现有的知识:
| 蛋白质鉴定 | 哪些蛋白质在特定的细胞类型、组织或整个生物体中正常表达,或者哪些蛋白质的表达有差异? |
| 蛋白质定量 | 测量总的(“稳态”)蛋白质丰度,以及调查蛋白质的周转率(即,蛋白质在产生和降解之间的循环速度)。 |
| 蛋白质的定位 | 蛋白质在哪里表达和/或积累与表达时间一样对蛋白质的功能至关重要,因为细胞定位控制着哪些分子相互作用的伙伴和目标可以使用。 |
| 翻译后修饰 | 翻译后修饰可以影响蛋白质的激活、定位、稳定性、相互作用和信号转导以及其他蛋白质特征,从而增加了一个重要的生物复杂性层。 |
| 功能性蛋白质组学 | 这一领域的蛋白质组学侧重于确定特定的单个蛋白质、一类蛋白质(如激酶)或整个蛋白质互动网络的生物功能。 |
| 结构蛋白质组学 | 结构研究对蛋白质功能、药物发现的蛋白质目标的“可药性”和药物设计产生重要的洞察力。 |
| 蛋白质-蛋白质相互作用 | 研究蛋白质之间如何相互作用,哪些蛋白质相互作用,以及何时何地相互作用。 |
4、蛋白质组学技术
4.1、低通量方法
4.1.1、基于抗体的方法
诸如ELISA(酶联免疫吸附试验)和Western blotting等技术依赖于针对特定蛋白质或表位的抗体的可用性,以识别蛋白质并量化其表达水平。
4.1.2、基于凝胶的方法
二维凝胶电泳(2DE或2D-PAGE)是最早开发的蛋白质组学技术,它使用电流根据蛋白质的电荷(第一维)和质量(第二维)在凝胶中分离。差分凝胶电泳(DIGE)是2DE的一种改良形式,使用不同的荧光染料,可以在同一凝胶上同时比较两到三个蛋白质样品。这些基于凝胶的方法被用来在进一步分析前分离蛋白质,例如质谱分析(MS),以及用于相对表达谱分析。
4.1.3、基于色谱的方法
基于色谱的方法可用于从复杂的生物混合物(如细胞裂解液)中分离和纯化蛋白质。例如,离子交换色谱法根据电荷分离蛋白质,尺寸排除色谱法根据分子大小分离蛋白质,亲和色谱法采用特定的亲和配体和其目标蛋白质之间的可逆相互作用(例如,使用凝集素纯化IgM和IgA分子)。这些方法可用于纯化和鉴定感兴趣的蛋白质,以及为进一步分析准备蛋白质,如下游的MS[8]。
4.2、高通量方法
4.2.1、分析微阵列、功能微阵列和反相微阵列
蛋白质微阵列将少量的样品应用于“芯片”进行分析(有时是以玻璃片的形式,表面经过化学修饰)。
特定的抗体可以被固定在芯片表面,用于捕获复杂样品中的目标蛋白。这被称为分析蛋白微阵列,这些类型的微阵列被用来测量样品中蛋白质的表达水平和结合亲和力。
功能蛋白微阵列被用来描述蛋白功能,如蛋白-RNA相互作用和酶-底物周转。在反相蛋白质芯片中,来自例如健康与病变组织或未处理与处理过的细胞的蛋白质被结合到芯片上,然后用针对目标蛋白质的抗体对芯片进行检测。

图2 | 正相和反相蛋白质芯片的区别
4.2.2、基于质谱的蛋白质组学
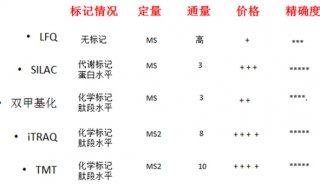
有几种“无凝胶”的方法来分离蛋白质,包括同位素编码亲和标签(ICAT)、细胞培养中的氨基酸稳定同位素标记(SILAC)和相对和绝对定量的同位素标签(iTRAQ)。这些方法既可以进行定量,也可以进行比较/鉴别蛋白质组学。
还有其他一些定量较少的技术,如多维蛋白质鉴定技术(MudPIT),其优点是更快和更简单。其他无凝胶的、用于蛋白质分离的色谱技术包括气相色谱法(GC)和液相色谱法(LC)[8,9]。
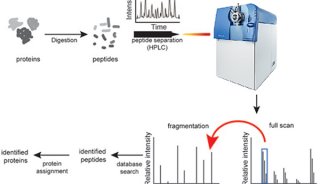
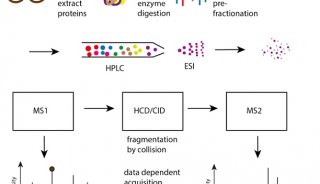
4.3、质谱分析工作流程
无论蛋白质样品是如何分离的,下游的质谱工作流程包括三个主要步骤:
蛋白质/肽被质谱仪的离子源离子化;
产生的离子根据其质量和电荷比被质量分析器分离;
离子被检测。
当使用MS上游的无凝胶技术,如iTRAQ或SILAC,样品直接用于进入质谱仪。当使用基于凝胶的技术时,蛋白质点首先从凝胶中切出并被酶解,然后用LC分离或直接用MS分析。
有两种主要的电离源,即:
基质辅助激光解吸/离子化(MALDI);
电喷雾离子化(ESI)。在蛋白质组学的真实应用中,由于样品量非常少,一般采用纳升级电喷雾离子化(nano ESI)或微升级电喷雾离子化(micro ESI)。原理与ESI类似。


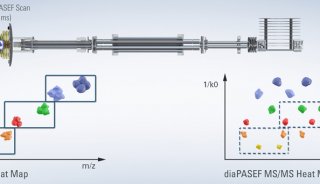
图3 | 基于MS的蛋白质组学的两个主要电离源 MALDI(上),nanoESI(下)
其他电离源还包括化学电离CI、电子轰击EI、ICP、辉光放电电离等电,但TA们不用于蛋白质组学,因为对生物分子不适合。
有6种主要的质量分析器:四极杆Q、离子阱IT、飞行时间TOF、磁质谱、轨道阱Orbitrap、傅里叶变换离子回旋共振FTICR。在实际的应用中,大部分定性应用采用复合(Hybrid)的质量分析器如:Q-TOF,Q-Orbi(四极杆-轨道阱串联)、LTQ-Orbitrap(离子阱-轨道阱串联)。当前,在离子传输区,许多生产厂家还使用了淌度分离或其它的阱来富集离子,从而提升组学分析的离子利用率(duty cycle)。在定量的应用中,大部分采用串联四极杆(QQQ)或四极杆-线性离子阱(Q-Trap)。
5、什么是串联质谱?
肽可以进行多轮破碎和质量分析,这一过程被称为串联MS、MS/MS或MSn。通过将相同或不同的质量分析器串联起来,如四极杆-TOF(Q-TOF)或三重四极杆(QQQ)MS,可以利用不同质量分析器的优势,进一步提高整个蛋白质组分析的能力。
简单的质谱设置,如MALDI-TOF仅用于肽的质量测量,而串联质谱仪则用于确定肽的序列。
6、自上而下蛋白质组学与自下而上蛋白质组学
自上而下(top-down)蛋白质组学,感兴趣的样品中的蛋白质首先被分离,然后再被单独表征[1,10]。
自下而上(bottom-up)蛋白质组学,又被称为“鸟枪法”蛋白质组学,样品中的所有蛋白质首先被消化成复杂的多肽混合物,然后对这些多肽进行分析以确定样品中存在哪些蛋白质[1,10]。
| 名称 | 说明 | 方法 |
| 自上而下蛋白质组学 | 在对感兴趣的样品中的蛋白质进行单独表征之前,首先要对其进行分离。 | 蛋白质分离是根据质量和电荷进行的,如2DE、DIGE或MS。当使用二维电泳技术时,蛋白质首先在凝胶上被解析,然后被单独消化成肽,由质谱仪进行分析。当直接使用质谱时,含有整个蛋白质的未消化样品被注入质谱仪,蛋白质被分离,然后选择单个蛋白质进行消化,再对消化后的肽段进行一轮质谱分析。 |
| 自下而上蛋白质组学,或“鸟枪法蛋白质组学” | 样品中的所有蛋白质首先被消化成复杂的多肽混合物,然后对这些多肽进行分析以确定样品中存在哪些蛋白质。 | 蛋白质首先被消化,消化后的多肽混合物被分馏并进行质谱分析,通常是在LC-MS/MS配置中。使用自动搜索算法将得到的肽序列与现有数据库进行比较。这些搜索引擎将实验获得的肽谱与硅基消化产生的蛋白质的预测谱相匹配(这被称为“肽谱匹配”)。可能有几种不同的自下而上的工作流程,包括依赖数据和不依赖数据的方法,以及这些方法的混合体。 |
自上而下和自下而上的方法都有各自的优点和缺点,以及各自更适合的应用[10,11]。例如,自上而下的质谱更适合于研究不同的PTMs和蛋白质的异构体。然而,它受到分离复杂的蛋白质混合物所固有的困难和MS对较大的蛋白质(特别是 > 50到70kDa)的敏感性下降的限制[1]。
相比之下,虽然自下而上的质谱中使用的肽(长度约为5到20个氨基酸)更容易分馏、离子化和片段化,但这种方法提供了对样品中最初存在的蛋白质的间接测量,并且严重依赖推断[1]。
一种混合的“自下而上”的方法已经被开发出来,它采用了比传统的自下而上蛋白质组学更大的肽段,从而有可能允许更多独特的肽段匹配。

图4 | 自上而下和自下而上蛋白质组学之间的差异
7、蛋白质组学的数据分析
蛋白质组学研究,特别是采用高通量技术的研究,可以产生大量的数据[12]。除了产生的大量数据外,蛋白质组学的数据分析对于某些技术来说也是相对复杂的,如鸟枪法质谱[13]。更加复杂的是可用于蛋白质组分析的生物信息学工具的范围[14-17]。
蛋白质组研究人员在试图优化他们的蛋白质组数据的存储和分析方式时,面临着许多障碍[12]。
在计划蛋白质组实验时,科学家不仅需要考虑试剂和实验室设备的成本,还需要考虑数据存储和分析的成本,他们还必须评估所需的生物信息学技能和计算资源水平。
蛋白质组学研究往往需要多个数据处理和分析步骤,需要按照特定的顺序进行[12]。为了满足这一需求,研究人员越来越多地将所需的脚本、工具和软件组装成适合其特定研究问题的定制蛋白质组分析管道。
8、蛋白质组学的应用
蛋白质组学的应用多得令人难以置信,而且种类繁多。下表列出了其中一些应用:
| 蛋白质组学的应用 | 描述和示例 |
| 个性化医疗 | 根据每个病人的基因和表观遗传构成,为其量身定制疾病治疗,以提高疗效,减少不良反应。虽然到目前为止,基因组学和转录组学是此类研究的主要焦点,但蛋白质组学数据可能会进一步增加针对患者的管理维度。 |
| 生物标志物的发现 | 鉴定蛋白质标记物,例如,胶质母细胞瘤的诊断和预后,以及评估患者对治疗干预的反应,如干细胞移植。 |
| 药物发现和开发 | 识别潜在的药物靶点,检查选定的蛋白质靶点的可药性,开发针对候选治疗性蛋白质靶点的药物(例如,针对肝细胞癌)。 |
| 系统生物学 | 对疾病途径和宿主-病原体相互作用的全系统调查,以确定潜在的生物标志物和治疗目标;对药物作用、毒性、抗性和疗效的全系统调查。 |
| 农业 | 研究植物与病原体之间的相互作用,提高作物对洪水、干旱和其他环境压力的抵抗力的工程。 |
| 食品科学 | 食品安全和质量控制,过敏原检测和提高食品的营养价值。 |
| 古蛋白组学 | 研究古代蛋白质以进一步了解进化和考古。 |
| 天体生物学 | 调查哺乳动物的免疫系统如何应对在太空中发现的外来微生物,以及研究在陨石上发现的前生物有机物质。 |
9、蛋白质组学的未来
目前,蛋白质组学工作流程在很大程度上依赖于质谱[1]。尽管这项技术已被证明是强大的,但研究人员现在正在展望蛋白质组学的未来,即“超越质谱”。尽管质谱的灵敏度很高,但样品中仍需要有数百万的目标分子才能被检测到。这意味着低浓度的目标分子(如血清生物标志物)在复杂的环境中(如人类血清)可能无法检测到,除非首先富集。
科学家们仍在寻找高通量蛋白质组技术的解决方案:
在目标蛋白质组的动态范围内具有出色的灵敏度(例如,人类蛋白质组的灵敏度为10^7);
可以直接读取整个蛋白质序列并识别其PTMs,以及;
不需要从理论蛋白质匹配数据库中进行推断[1]。
有几种有前途的技术,虽然目前受到灵敏度、通量或成本的限制,但可能会在蛋白质组学领域占据主导地位[1]。这些技术包括新生的荧光指纹方法和尚未开发的用于蛋白质高通量单分子测序的亚纳米孔阵列。
随着蛋白质组学技术的发展,蛋白质组学数据分析的方法也将同样快速发展。例如,云计算、软件容器和工作流系统等数据技术的发展势头强劲,这些技术将使人们能够自由的获得用于蛋白质组数据分析的顶级计算资源,而不受研究人员的位置、IT基础设施或计算专长的影响[12,18,19]。
参考文献
Timp W, Timp G. Beyond mass spectrometry, the next step in proteomics. Sci Adv. 2020;6(2):eaax8978. doi:10.1126/sciadv.aax8978.
Wilkins M. Proteomics data mining. Expert Rev Proteomics. 2009;6(6):599-603. doi:10.1586/epr.09.81.
Beynon RJ. The dynamics of the proteome: strategies for measuring protein turnover on a proteome-wide scale. Brief Funct Genomic Proteomic. 2005;3(4):382-390. doi:10.1093/bfgp/3.4.382.
Garrels JI. Proteome. In: Brenner S, Miller JH, eds. Encyclopaedia of Genetics. London: Academic Press; 2001:1575-1578.
Graves PR, Haystead TA. Molecular biologist's guide to proteomics. Microbiol Mol Biol Rev. 2002;66(1):39-63. doi:10.1128/mmbr.66.1.39-63.2002.
Andersen JS, Mann M. Functional genomics by mass spectrometry. FEBS Lett. 2000;480(1):25-31. doi:10.1016/s0014-5793(00)01773-7.
Bekker-Jensen DB, Martínez-Val A, Steigerwald S, et al. A compact quadrupole-orbitrap mass spectrometer with FAIMS interface improves proteome coverage in short LC gradients. Mol Cell Proteomics. 2020;19(4):716-729. doi:10.1074/mcp.TIR119.0019061.
Aslam B, Basit M, Nisar MA, Khurshid M, Rasool MH. Proteomics: Technologies and their applications. J Chromatogr Sci. 2017;55(2):182-196. doi:10.1093/chromsci/bmw167.
Chandramouli K, Qian PY. Proteomics: challenges, techniques and possibilities to overcome biological sample complexity. Hum Genomics Proteomics. 2009;2009:239204. doi:10.4061/2009/239204.
Zhang Y, Fonslow BR, Shan B, Baek MC, Yates JR 3rd. Protein analysis by shotgun/bottom-up proteomics. Chem Rev. 2013;113(4):2343-2394. doi:10.1021/cr3003533.
Zhang H, Ge Y. Comprehensive analysis of protein modifications by top-down mass spectrometry. Circ Cardiovasc Genet. 2011;4(6):711. doi:10.1161/CIRCGENETICS.110.957829.
Perez‐Riverol Y, Moreno P. Scalable data analysis in proteomics and metabolomics using BioContainers and workflows engines. Proteomics. 2020;20:1900147. doi:10.1002/pmic.201900147.
Hu A, Noble WS, Wolf-Yadlin A. Technical advances in proteomics: new developments in data-independent acquisition. F1000Res. 2016;5:F1000 Faculty Rev-419. doi:10.12688/f1000research.7042.1.
Ison J, Rapacki K, Ménager H, et al. Tools and data services registry: a community effort to document bioinformatics resources. Nucleic Acids Res. 2016;44(D1):D38-D47. doi:10.1093/nar/gkv1116
Henry VJ, Bandrowski AE, Pepin AS, Gonzalez BJ, Desfeux A. OMICtools: an informative directory for multi-omic data analysis. Database. 2014;2014:bau069. doi:10.1093/database/bau069.
Afgan E, Baker D, Batut B, et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Res. 2018;46(W1):W537-W544. doi:10.1093/nar/gky379.
Tsiamis V, Ienasescu H, Gabrielaitis D, Palmblad M, Schwämmle V, Ison J. One thousand and one software for proteomics: Tales of the toolmakers of science. J Proteome Res. 2019;18(10):3580-3585. doi:10.1021/acs.jproteome.9b00219.
Cole BS, Moore JH. Eleven quick tips for architecting biomedical informatics workflows with cloud computing. PLoS Comput Biol. 2018;14(3):e1005994. doi:10.1371/journal.pcbi.1005994.
Lawlor B, Sleator RD. The democratization of bioinformatics: A software engineering perspective. GigaScience. 2020;9(6):giaa063. doi:10.1093/gigascience/giaa063.
-
精英视角

-
产品技术

-
精英视角

-
会议会展

-
综述
-
企业风采
-
技术原理